%EC%9C%A0%EC%A0%84%EC%9E%90
-
 세계 최초 유전자 가위로 원하는 RNA ‘콕’ 집어 변형 성공
RNA 유전자 가위는 코로나바이러스와 같은 바이러스의 RNA를 제거하여 감염을 억제하거나 질병 원인 유전자 발현을 조절할 수 있어, 부작용이 적은 차세대 유전자 치료제로 크게 주목받고 있다. 우리 연구진은 세포 내 존재하는 수많은 RNA(유전 정보를 전달하고 단백질을 만드는 데 중요한 역할을 하는 분자) 중에서 원하는 RNA만을 정확하게 찾아서 아세틸화(화학 변형)할 수 있는 기술을 세계 최초로 개발했고, 이는 RNA 기반 치료의 새 장을 열 수 있는 핵심 기술이 될 것으로 기대된다.
우리 대학 생명과학과 허원도 석좌교수 연구팀이 최근 유전자 조절 및 RNA 기반 기술 분야에서 각광받는 RNA 유전자 가위 시스템(CRISPR-Cas13)을 이용해 우리 몸 안의 특정한 RNA에 아세틸화를 가할 수 있는 혁신적 기술을 개발했다고 10일 밝혔다.
RNA는 ‘화학 변형(chemical modification)’이란 과정을 통해 그 특성과 기능이 변화할 수 있다. 화학 변형이란 RNA 염기 서열 자체의 변함없이 특정 화학 그룹이 추가됨으로써 RNA의 성질과 역할을 변화시키는 유전자 조절 과정이다. 그중 하나가 시티딘 아세틸화(N4-acetylcytidine)라는 화학 변형인데, 지금까지는 이 화학 변형이 세포 내에서 어떤 기능을 수행하는지 정확히 알려져 있지 않았다. 특히, 인간 세포의 mRNA(단백질을 만드는 RNA)에 이 변형이 실제로 있는지, 어떤 역할을 하는지 등에 대한 논란이 이어졌다.
연구팀은 이러한 한계를 극복하기 위해 원하는 RNA만을 정밀하게 표적하는 유전자 가위인 Cas13에 RNA를 아세틸화시키는 NAT10의 고활성 변이체(eNAT10)를 결합한 ‘표적 RNA 아세틸화 시스템(dCas13-eNAT10)’을 개발했다. 즉, 원하는 RNA만 정확하게 골라서 아세틸화시키는 ‘표적 RNA 변형 기술’을 만든 것이다.
연구팀은 표적 RNA 아세틸화 시스템과 세포 내 특정 RNA를 찾아 안내하는 가이드 RNA에 의해 원하는 RNA에 아세틸화 화학 변형을 가할 수 있음을 증명했다. 이를 통해 아세틸화 화학 변형된 메신저 RNA (mRNA)에서 단백질 생산이 증가한다는 사실을 확인했다.
또한, 연구팀은 개발한 시스템을 이용해 RNA 아세틸화가 RNA를 세포핵에서 세포질로 이동시킨다는 사실을 최초로 밝혀냈다. 이번 연구는 아세틸화 화학 변형이 세포 내 RNA ‘위치 이동’도 조절할 수 있다는 가능성을 보여주는 결과다.
연구팀은 개발한 기술이 AAV(아데노-관련 바이러스)라는 유전자 치료에 널리 이용되는 운반체 바이러스를 통해 실험 쥐의 간에 전달하여 동물의 몸속에서도 정확히 RNA 아세틸화 조절이 가능할 수 있음을 입증했다. 이는 RNA를 화학 변형하는 기술이 생체 내 적용에 확장될 수 있음을 보여주는 최초의 사례다. 이는 RNA 기반 유전자 치료 기술로의 응용 가능성을 여는 성과로 평가받는다.
RNA 유전자가위를 활용한 코로나 치료기술과 빛으로 RNA 유전자가위 활성화 기술을 개발하였던 허원도 교수는 “기존 RNA 화학 변형 연구는 특정성, 시간성, 공간성 조절이 어려웠지만, 이번 기술은 원하는 RNA에 선택적으로 아세틸화를 가할 수 있어 RNA 아세틸화의 기능을 정확하고 세밀하게 연구할 수 있는 길을 열였다”며, “이번에 개발한 RNA 화학 변형 기술은 향후 RNA 기반 치료제 및 생체 내 RNA 작동을 조절하는 도구로 폭넓게 활용될 수 있을 것”이라고 전했다.
우리 대학 생명과학과 유지환 박사과정이 제1 저자로 수행한 이 연구는 국제 학술지 ‘네이처 케미컬 바이올로지 (Nature Chemical Biology)’에 2025년 6월 2일 자로 게재됐다.
(논문명: Programmable RNA acetylation with CRISPR-Cas13, Impact factor: 12.9, DOI: https://doi.org/10.1038/s41589-025-01922-3)
한편, 이번 연구는 삼성미래기술육성재단과 한국연구재단 바이오·의료기술개발사업의 지원을 받아 수행됐다.
2025.06.10 조회수 2683
세계 최초 유전자 가위로 원하는 RNA ‘콕’ 집어 변형 성공
RNA 유전자 가위는 코로나바이러스와 같은 바이러스의 RNA를 제거하여 감염을 억제하거나 질병 원인 유전자 발현을 조절할 수 있어, 부작용이 적은 차세대 유전자 치료제로 크게 주목받고 있다. 우리 연구진은 세포 내 존재하는 수많은 RNA(유전 정보를 전달하고 단백질을 만드는 데 중요한 역할을 하는 분자) 중에서 원하는 RNA만을 정확하게 찾아서 아세틸화(화학 변형)할 수 있는 기술을 세계 최초로 개발했고, 이는 RNA 기반 치료의 새 장을 열 수 있는 핵심 기술이 될 것으로 기대된다.
우리 대학 생명과학과 허원도 석좌교수 연구팀이 최근 유전자 조절 및 RNA 기반 기술 분야에서 각광받는 RNA 유전자 가위 시스템(CRISPR-Cas13)을 이용해 우리 몸 안의 특정한 RNA에 아세틸화를 가할 수 있는 혁신적 기술을 개발했다고 10일 밝혔다.
RNA는 ‘화학 변형(chemical modification)’이란 과정을 통해 그 특성과 기능이 변화할 수 있다. 화학 변형이란 RNA 염기 서열 자체의 변함없이 특정 화학 그룹이 추가됨으로써 RNA의 성질과 역할을 변화시키는 유전자 조절 과정이다. 그중 하나가 시티딘 아세틸화(N4-acetylcytidine)라는 화학 변형인데, 지금까지는 이 화학 변형이 세포 내에서 어떤 기능을 수행하는지 정확히 알려져 있지 않았다. 특히, 인간 세포의 mRNA(단백질을 만드는 RNA)에 이 변형이 실제로 있는지, 어떤 역할을 하는지 등에 대한 논란이 이어졌다.
연구팀은 이러한 한계를 극복하기 위해 원하는 RNA만을 정밀하게 표적하는 유전자 가위인 Cas13에 RNA를 아세틸화시키는 NAT10의 고활성 변이체(eNAT10)를 결합한 ‘표적 RNA 아세틸화 시스템(dCas13-eNAT10)’을 개발했다. 즉, 원하는 RNA만 정확하게 골라서 아세틸화시키는 ‘표적 RNA 변형 기술’을 만든 것이다.
연구팀은 표적 RNA 아세틸화 시스템과 세포 내 특정 RNA를 찾아 안내하는 가이드 RNA에 의해 원하는 RNA에 아세틸화 화학 변형을 가할 수 있음을 증명했다. 이를 통해 아세틸화 화학 변형된 메신저 RNA (mRNA)에서 단백질 생산이 증가한다는 사실을 확인했다.
또한, 연구팀은 개발한 시스템을 이용해 RNA 아세틸화가 RNA를 세포핵에서 세포질로 이동시킨다는 사실을 최초로 밝혀냈다. 이번 연구는 아세틸화 화학 변형이 세포 내 RNA ‘위치 이동’도 조절할 수 있다는 가능성을 보여주는 결과다.
연구팀은 개발한 기술이 AAV(아데노-관련 바이러스)라는 유전자 치료에 널리 이용되는 운반체 바이러스를 통해 실험 쥐의 간에 전달하여 동물의 몸속에서도 정확히 RNA 아세틸화 조절이 가능할 수 있음을 입증했다. 이는 RNA를 화학 변형하는 기술이 생체 내 적용에 확장될 수 있음을 보여주는 최초의 사례다. 이는 RNA 기반 유전자 치료 기술로의 응용 가능성을 여는 성과로 평가받는다.
RNA 유전자가위를 활용한 코로나 치료기술과 빛으로 RNA 유전자가위 활성화 기술을 개발하였던 허원도 교수는 “기존 RNA 화학 변형 연구는 특정성, 시간성, 공간성 조절이 어려웠지만, 이번 기술은 원하는 RNA에 선택적으로 아세틸화를 가할 수 있어 RNA 아세틸화의 기능을 정확하고 세밀하게 연구할 수 있는 길을 열였다”며, “이번에 개발한 RNA 화학 변형 기술은 향후 RNA 기반 치료제 및 생체 내 RNA 작동을 조절하는 도구로 폭넓게 활용될 수 있을 것”이라고 전했다.
우리 대학 생명과학과 유지환 박사과정이 제1 저자로 수행한 이 연구는 국제 학술지 ‘네이처 케미컬 바이올로지 (Nature Chemical Biology)’에 2025년 6월 2일 자로 게재됐다.
(논문명: Programmable RNA acetylation with CRISPR-Cas13, Impact factor: 12.9, DOI: https://doi.org/10.1038/s41589-025-01922-3)
한편, 이번 연구는 삼성미래기술육성재단과 한국연구재단 바이오·의료기술개발사업의 지원을 받아 수행됐다.
2025.06.10 조회수 2683 -
 슈퍼박테리아 방패 ‘바이오필름’ 무력화 치료 플랫폼 개발
병원 내 감염의 주요 원인 중 하나로 알려진 슈퍼박테리아 ‘메티실린 내성 황색포도상구균(MRSA, 이하 포도상구균)’은 기존 항생제에 대한 높은 내성뿐 아니라 강력한 미생물막인 바이오필름(biofilm)을 형성함으로써 외부 치료제를 효과적으로 차단한다. 이에 우리 연구진은 국제 연구진과 함께 미세방울(microbubble)을 이용해 유전자 표적 나노입자를 전달하여 바이오필름을 무너뜨리고 기존 항생제가 무력한 감염증에 대한 혁신적 해결책을 제시하는 플랫폼 개발에 성공했다.
우리 대학 생명과학과 정현정 교수 연구팀이 미국 일리노이대 공현준 교수팀과의 공동연구를 통해, 포도상구균이 형성한 세균성 바이오필름을 효과적으로 제거하기 위해 유전자 억제제를 세균 내부로 정확하게 전달하는 미세방울 기반 나노-유전자 전달 플랫폼(BTN‑MB)를 개발했다고 29일 밝혔다.
연구팀은 먼저, 포도상구균의 주요 유전자 3종<바이오필름 형성(icaA), 세포 분열(ftsZ), 항생제 내성(mecA)>을 동시에 억제하는 짧은 DNA 조각(oligonucleotide)을 설계하고, 이를 탑재해 균내로 효과적으로 전달할 수 있는 나노입자(BTN)를 고안했다.
여기에 더해, 미세방울(microbubble, 이하 MB)을 사용해 포도상구균이 형성한 바이오필름인 미생물막의 투과성을 높인다. 연구팀은 두 가지 기술을 병용해, 세균의 증식과 내성 획득을 원천적으로 차단하는 이중 타격 전략을 구현했다.
이 치료 시스템은 두 단계로 작동한다. 먼저, 미세방울(MB)이 포도상구균이 형성한 세균성 생체막내 압력 변화로 나노입자(BTN)의 침투를 가능하게 만든다. 이어서, BTN이 생체막의 틈을 타 세균 내부로 침투해 유전자 억제제를 정확하게 전달한다. 이를 통해 포도상구균의 유전자 조절을 일으켜 생체막 재형성, 세포 증식, 그리고 항생제 내성 발현이 동시에 차단된다.
돼지 피부 감염 생체막 모델과 포도상구균 감염 마우스 상처 모델에서 시행한 실험 결과, BTN‑MB 치료군은 생체막 두께가 크게 감소했으며, 세균 수와 염증 반응도 현저히 줄어드는 뛰어난 치료 효과를 확인할 수 있었다.
이러한 결과는 기존 항생제 단독 치료로는 달성하기 어려운 수준이며, 향후 다양한 내성균 감염 치료에도 적용할 수 있는 가능성을 보여준다.
연구를 주도한 정현정 교수는 “이번 연구는 기존 항생제로는 해결할 수 없는 슈퍼박테리아 감염에 대해 나노기술, 유전자 억제, 물리적 접근법을 융합해 새로운 치료 해법을 제시한 것”이라며, “향후 전신 적용 및 다양한 감염 질환으로의 확장을 목표로 연구를 지속할 것”이라고 설명했다.
해당 연구는 우리 대학 생명과학과 정주연 학생과 일리노이대 안유진 박사가 제1 저자로 참여했으며, 국제학술지‘어드밴스드 펑셔널 머터리얼스(Advanced Functional Materials)’에 5월 19일 자로 온라인 게재됐다.
※ 논문 제목: Microbubble-Controlled Delivery of Biofilm-Targeting Nanoparticles to Treat MRSA Infection
※ DOI: https://doi.org/10.1002/adfm.202508291
한편, 이번 연구는 한국연구재단과 보건복지부의 지원을 받아 수행됐다.
2025.05.29 조회수 2424
슈퍼박테리아 방패 ‘바이오필름’ 무력화 치료 플랫폼 개발
병원 내 감염의 주요 원인 중 하나로 알려진 슈퍼박테리아 ‘메티실린 내성 황색포도상구균(MRSA, 이하 포도상구균)’은 기존 항생제에 대한 높은 내성뿐 아니라 강력한 미생물막인 바이오필름(biofilm)을 형성함으로써 외부 치료제를 효과적으로 차단한다. 이에 우리 연구진은 국제 연구진과 함께 미세방울(microbubble)을 이용해 유전자 표적 나노입자를 전달하여 바이오필름을 무너뜨리고 기존 항생제가 무력한 감염증에 대한 혁신적 해결책을 제시하는 플랫폼 개발에 성공했다.
우리 대학 생명과학과 정현정 교수 연구팀이 미국 일리노이대 공현준 교수팀과의 공동연구를 통해, 포도상구균이 형성한 세균성 바이오필름을 효과적으로 제거하기 위해 유전자 억제제를 세균 내부로 정확하게 전달하는 미세방울 기반 나노-유전자 전달 플랫폼(BTN‑MB)를 개발했다고 29일 밝혔다.
연구팀은 먼저, 포도상구균의 주요 유전자 3종<바이오필름 형성(icaA), 세포 분열(ftsZ), 항생제 내성(mecA)>을 동시에 억제하는 짧은 DNA 조각(oligonucleotide)을 설계하고, 이를 탑재해 균내로 효과적으로 전달할 수 있는 나노입자(BTN)를 고안했다.
여기에 더해, 미세방울(microbubble, 이하 MB)을 사용해 포도상구균이 형성한 바이오필름인 미생물막의 투과성을 높인다. 연구팀은 두 가지 기술을 병용해, 세균의 증식과 내성 획득을 원천적으로 차단하는 이중 타격 전략을 구현했다.
이 치료 시스템은 두 단계로 작동한다. 먼저, 미세방울(MB)이 포도상구균이 형성한 세균성 생체막내 압력 변화로 나노입자(BTN)의 침투를 가능하게 만든다. 이어서, BTN이 생체막의 틈을 타 세균 내부로 침투해 유전자 억제제를 정확하게 전달한다. 이를 통해 포도상구균의 유전자 조절을 일으켜 생체막 재형성, 세포 증식, 그리고 항생제 내성 발현이 동시에 차단된다.
돼지 피부 감염 생체막 모델과 포도상구균 감염 마우스 상처 모델에서 시행한 실험 결과, BTN‑MB 치료군은 생체막 두께가 크게 감소했으며, 세균 수와 염증 반응도 현저히 줄어드는 뛰어난 치료 효과를 확인할 수 있었다.
이러한 결과는 기존 항생제 단독 치료로는 달성하기 어려운 수준이며, 향후 다양한 내성균 감염 치료에도 적용할 수 있는 가능성을 보여준다.
연구를 주도한 정현정 교수는 “이번 연구는 기존 항생제로는 해결할 수 없는 슈퍼박테리아 감염에 대해 나노기술, 유전자 억제, 물리적 접근법을 융합해 새로운 치료 해법을 제시한 것”이라며, “향후 전신 적용 및 다양한 감염 질환으로의 확장을 목표로 연구를 지속할 것”이라고 설명했다.
해당 연구는 우리 대학 생명과학과 정주연 학생과 일리노이대 안유진 박사가 제1 저자로 참여했으며, 국제학술지‘어드밴스드 펑셔널 머터리얼스(Advanced Functional Materials)’에 5월 19일 자로 온라인 게재됐다.
※ 논문 제목: Microbubble-Controlled Delivery of Biofilm-Targeting Nanoparticles to Treat MRSA Infection
※ DOI: https://doi.org/10.1002/adfm.202508291
한편, 이번 연구는 한국연구재단과 보건복지부의 지원을 받아 수행됐다.
2025.05.29 조회수 2424 -
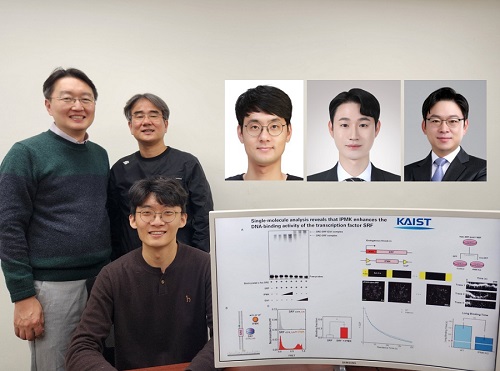 암 발생 현상 등 유전자 발현 조절 원리 규명
다양한 암 발생과 암전이 현상, 줄기세포로부터의 조직 분화 및 발생, 신경 세포의 활성화 과정 등을 근본적으로 일으키는 유전자 발현 조절 단백질의 핵심 유전자 발현 네트워크를 발견했다. 우리 연구진은 이 발견을 기초로 하여 혁신적인 치료 기술 개발에 활용 가능성을 높였다.
우리 대학 생명과학과 김세윤 교수, 이광록 교수, 조원기 교수 공동연구팀이 동물 세포의 유전자 발현을 조절하는 핵심적인 원리를 규명했다고 22일 밝혔다.
이노시톨 대사 효소에 의해 만들어지는 이노시톨 인산 대사체는 진핵 세포의 신호전달 시스템에 필요한 다양한 이차 신호전달물질로 작용하며 암, 비만, 당뇨, 신경계 질환들에 폭넓게 관여한다.
연구팀은 이노시톨 대사 시스템의 핵심 효소인 IPMK 단백질이 동물 세포의 핵심 유전자 발현 네트워크의 중요한 전사 활성화 인자로 작용함을 규명했다.
포도당과 유사한 영양소로 알려진 이노시톨의 대사 반응에 핵심적으로 작용하는 효소인 IPMK 단백질(inositol polyphosphate multikinase)은 유전자 발현을 직접적으로 조절하는 기능을 가지고 있다. 특히 IPMK 효소는 동물 세포의 대표적인 전사 인자(transcription factor)인 혈청 반응 인자(serum response factor, 이하 SRF)에 의한 유전자 전사 과정에 중요하다고 보고된 바 있으나 작용하는 기전에 대하여 알려진 바는 없었다.
SRF 전사 인자는 최소 200~300여 개의 유전자 발현을 직접적으로 조절하는 단백질로서, 동물 세포의 성장과 증식, 세포 사멸, 세포의 이동성 등을 조절하며 심장과 같은 장기 발생에 필수적이다.
연구팀은 IPMK 단백질이 SRF 전사 인자와 직접적으로 결합한다는 사실을 발견하고 이를 통해 SRF 전사 인자의 3차원적 단백질 구조를 변화시킨다는 것을 밝혔다.
연구팀은 IPMK 효소에 의하여 활성화된 SRF 전사 인자를 통해 다양한 유전자들의 전사 과정이 촉진된다는 것을 밝혔다. 즉, IPMK 단백질은 SRF 전사 인자의 단백질 활성을 높이는 데 반드시 필요한 조절 스위치와 같은 역할을 수행하는 것임을 연구팀은 규명했다.
연구팀은 IPMK 효소와 SRF 전사 인자 사이의 직접적인 결합에 문제가 발생할 경우, SRF 전사 인자의 기능과 활성이 낮아져 유전자 발현에 심각한 장애가 발생한다는 점을 최종적으로 검증하였다.
특히 SRF 전사 인자가 가지고 있는 비정형 영역(Intrinsically disordered region, IDR)이 중요한 조절 부위라는 점을 밝힘으로써 비정형 단백질의 생물학적 중요성을 제시했다. 보통 단백질은 접힘을 통해 고유의 구조를 나타내지만 비정형 영역을 포함하게 되는 경우에는 특정한 단백질 구조가 관찰되지 않는다. 학계에서는 이러한 비정형 영역을 가지고 있는 단백질들을 비정형 단백질이라고 구분하고 어떠한 기능을 수행하는지 주목하고 있다.
김세윤 교수는 “이번 연구는 이노시톨 대사 시스템의 핵심 효소인 IPMK 단백질이 동물 세포의 핵심 유전자 발현 네트워크의 중요한 전사 활성화 인자이며 이를 증명하는 핵심 메커니즘을 제시한 중요한 발견”이라며, “SRF 전사 인자로부터 파생되는 다양한 암의 발생과 암전이 현상, 줄기세포로부터의 조직 분화 및 발생, 신경 세포 활성화 과정 등을 근본적으로 이해함으로써 혁신적인 치료 기술 개발 등에 폭넓게 활용되기를 바란다”라는 기대를 전했다.
이 연구는 세계적 국제학술지 ‘핵산 연구 (Nucleic Acids Research)’ (IF=16.7, 생화학 및 분자생물학 분야 상위 1.8%) 온라인판에 1월 7일 게재됐다. (논문명 : Single-molecule analysis reveals that IPMK enhances the DNA-binding activity of the transcription factor SRF) (doi: 10.1093/nar/gkae1281)
한편 이번 연구는 한국연구재단의 중견연구사업, 선도연구센터 지원사업, 글로벌 기초연구실 지원사업과 서경배과학재단, 삼성미래기술육성사업의 지원을 받아 수행됐다.
2025.01.22 조회수 4898
암 발생 현상 등 유전자 발현 조절 원리 규명
다양한 암 발생과 암전이 현상, 줄기세포로부터의 조직 분화 및 발생, 신경 세포의 활성화 과정 등을 근본적으로 일으키는 유전자 발현 조절 단백질의 핵심 유전자 발현 네트워크를 발견했다. 우리 연구진은 이 발견을 기초로 하여 혁신적인 치료 기술 개발에 활용 가능성을 높였다.
우리 대학 생명과학과 김세윤 교수, 이광록 교수, 조원기 교수 공동연구팀이 동물 세포의 유전자 발현을 조절하는 핵심적인 원리를 규명했다고 22일 밝혔다.
이노시톨 대사 효소에 의해 만들어지는 이노시톨 인산 대사체는 진핵 세포의 신호전달 시스템에 필요한 다양한 이차 신호전달물질로 작용하며 암, 비만, 당뇨, 신경계 질환들에 폭넓게 관여한다.
연구팀은 이노시톨 대사 시스템의 핵심 효소인 IPMK 단백질이 동물 세포의 핵심 유전자 발현 네트워크의 중요한 전사 활성화 인자로 작용함을 규명했다.
포도당과 유사한 영양소로 알려진 이노시톨의 대사 반응에 핵심적으로 작용하는 효소인 IPMK 단백질(inositol polyphosphate multikinase)은 유전자 발현을 직접적으로 조절하는 기능을 가지고 있다. 특히 IPMK 효소는 동물 세포의 대표적인 전사 인자(transcription factor)인 혈청 반응 인자(serum response factor, 이하 SRF)에 의한 유전자 전사 과정에 중요하다고 보고된 바 있으나 작용하는 기전에 대하여 알려진 바는 없었다.
SRF 전사 인자는 최소 200~300여 개의 유전자 발현을 직접적으로 조절하는 단백질로서, 동물 세포의 성장과 증식, 세포 사멸, 세포의 이동성 등을 조절하며 심장과 같은 장기 발생에 필수적이다.
연구팀은 IPMK 단백질이 SRF 전사 인자와 직접적으로 결합한다는 사실을 발견하고 이를 통해 SRF 전사 인자의 3차원적 단백질 구조를 변화시킨다는 것을 밝혔다.
연구팀은 IPMK 효소에 의하여 활성화된 SRF 전사 인자를 통해 다양한 유전자들의 전사 과정이 촉진된다는 것을 밝혔다. 즉, IPMK 단백질은 SRF 전사 인자의 단백질 활성을 높이는 데 반드시 필요한 조절 스위치와 같은 역할을 수행하는 것임을 연구팀은 규명했다.
연구팀은 IPMK 효소와 SRF 전사 인자 사이의 직접적인 결합에 문제가 발생할 경우, SRF 전사 인자의 기능과 활성이 낮아져 유전자 발현에 심각한 장애가 발생한다는 점을 최종적으로 검증하였다.
특히 SRF 전사 인자가 가지고 있는 비정형 영역(Intrinsically disordered region, IDR)이 중요한 조절 부위라는 점을 밝힘으로써 비정형 단백질의 생물학적 중요성을 제시했다. 보통 단백질은 접힘을 통해 고유의 구조를 나타내지만 비정형 영역을 포함하게 되는 경우에는 특정한 단백질 구조가 관찰되지 않는다. 학계에서는 이러한 비정형 영역을 가지고 있는 단백질들을 비정형 단백질이라고 구분하고 어떠한 기능을 수행하는지 주목하고 있다.
김세윤 교수는 “이번 연구는 이노시톨 대사 시스템의 핵심 효소인 IPMK 단백질이 동물 세포의 핵심 유전자 발현 네트워크의 중요한 전사 활성화 인자이며 이를 증명하는 핵심 메커니즘을 제시한 중요한 발견”이라며, “SRF 전사 인자로부터 파생되는 다양한 암의 발생과 암전이 현상, 줄기세포로부터의 조직 분화 및 발생, 신경 세포 활성화 과정 등을 근본적으로 이해함으로써 혁신적인 치료 기술 개발 등에 폭넓게 활용되기를 바란다”라는 기대를 전했다.
이 연구는 세계적 국제학술지 ‘핵산 연구 (Nucleic Acids Research)’ (IF=16.7, 생화학 및 분자생물학 분야 상위 1.8%) 온라인판에 1월 7일 게재됐다. (논문명 : Single-molecule analysis reveals that IPMK enhances the DNA-binding activity of the transcription factor SRF) (doi: 10.1093/nar/gkae1281)
한편 이번 연구는 한국연구재단의 중견연구사업, 선도연구센터 지원사업, 글로벌 기초연구실 지원사업과 서경배과학재단, 삼성미래기술육성사업의 지원을 받아 수행됐다.
2025.01.22 조회수 4898 -
 암세포를 정상세포로 되돌려 치료하는 원천기술 개발
지금까지 다양한 항암 치료 기술이 개발됐음에도 현재 시행되고 있는 모든 항암치료의 공통점은 암세포를 사멸시켜서 치료하는 것을 목표로 하고 있다. 이로 인해 암세포가 내성을 획득해 재발하거나 정상세포까지 사멸시켜 큰 부작용을 유발하는 등 근본적인 한계를 지니고 있다.
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 대장암세포를 죽이지 않고 그 상태만을 변환시켜 정상 대장세포와 유사한 상태로 되돌림으로써 부작용 없이 치료할 수 있는 대장암 가역 치료를 위한 원천기술을 개발하였다고 22일 밝혔다.
연구팀은 정상세포의 암화 과정에서 정상적인 세포분화 궤적을 역행한다는 관찰 결과에 주목하고, 이를 기반으로 정상세포의 분화궤적에 대한 유전자네트워크의 디지털트윈을 제작하는 기술을 개발했다.
그리고 이를 시뮬레이션 분석해 정상세포 분화를 유도하는 마스터 분자스위치를 체계적으로 탐색해 발굴한 뒤 대장암세포에 적용했을 때 대장암세포의 상태가 정상화된다는 것을 분자세포 실험과 동물실험을 통해 입증했다.
이번 연구성과는 암세포를 정상세포로 가역화 하는 것이 우연한 현상적 발견에 의존하는 것이 아니라 암세포 유전자 네트워크의 디지털 트윈을 제작하고 분석함으로써 체계적으로 접근해 이루어낼 수 있음을 보인 원천기술 개발이며 이 기술을 다른 다양한 암종에 응용하여 암 가역 치료제 개발이 가능함을 제시한 것에 큰 의미가 있다.
조광현 교수는 "암세포가 정상세포로 변환될 수 있다는 것은 놀라운 현상이다. 이번 성과는 이를 체계적으로 유도해낼 수 있음을 증명한 것이다ˮ라고 말했다.
이어 “이번 연구 결과는 암세포를 정상세포로 되돌리는 가역 치료 개념을 최초로 제시한 성과들을 바탕으로 정상세포의 분화궤적을 체계적으로 분석해 암 가역화 치료타겟을 발굴하는 원천기술을 개발한 것이다”라고 강조했다.
우리 대학 공정렬 박사, 이춘경 박사과정 학생, 김훈민 박사과정 학생, 김주희 박사과정 학생 등이 참여한 이번 연구 결과는 와일리(Wiley)에서 출간하는 국제저널 `어드밴스드 사이언스(Advanced Science)' 12월 11일 字 온라인판 논문으로 출판됐다. (논문명: Control of cellular differentiation trajectories for cancer reversion) DOI: https://doi.org/10.1002/advs.202402132
한편 이번 연구는 과학기술정보통신부와 한국연구재단의 중견연구사업 및 기초연구실사업의 지원을 통해 수행되었으며 연구 성과는 바이오리버트(주)로 기술이전 되어 실제 암 가역치료제 개발에 활용될 예정이다.
2024.12.23 조회수 10468
암세포를 정상세포로 되돌려 치료하는 원천기술 개발
지금까지 다양한 항암 치료 기술이 개발됐음에도 현재 시행되고 있는 모든 항암치료의 공통점은 암세포를 사멸시켜서 치료하는 것을 목표로 하고 있다. 이로 인해 암세포가 내성을 획득해 재발하거나 정상세포까지 사멸시켜 큰 부작용을 유발하는 등 근본적인 한계를 지니고 있다.
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 대장암세포를 죽이지 않고 그 상태만을 변환시켜 정상 대장세포와 유사한 상태로 되돌림으로써 부작용 없이 치료할 수 있는 대장암 가역 치료를 위한 원천기술을 개발하였다고 22일 밝혔다.
연구팀은 정상세포의 암화 과정에서 정상적인 세포분화 궤적을 역행한다는 관찰 결과에 주목하고, 이를 기반으로 정상세포의 분화궤적에 대한 유전자네트워크의 디지털트윈을 제작하는 기술을 개발했다.
그리고 이를 시뮬레이션 분석해 정상세포 분화를 유도하는 마스터 분자스위치를 체계적으로 탐색해 발굴한 뒤 대장암세포에 적용했을 때 대장암세포의 상태가 정상화된다는 것을 분자세포 실험과 동물실험을 통해 입증했다.
이번 연구성과는 암세포를 정상세포로 가역화 하는 것이 우연한 현상적 발견에 의존하는 것이 아니라 암세포 유전자 네트워크의 디지털 트윈을 제작하고 분석함으로써 체계적으로 접근해 이루어낼 수 있음을 보인 원천기술 개발이며 이 기술을 다른 다양한 암종에 응용하여 암 가역 치료제 개발이 가능함을 제시한 것에 큰 의미가 있다.
조광현 교수는 "암세포가 정상세포로 변환될 수 있다는 것은 놀라운 현상이다. 이번 성과는 이를 체계적으로 유도해낼 수 있음을 증명한 것이다ˮ라고 말했다.
이어 “이번 연구 결과는 암세포를 정상세포로 되돌리는 가역 치료 개념을 최초로 제시한 성과들을 바탕으로 정상세포의 분화궤적을 체계적으로 분석해 암 가역화 치료타겟을 발굴하는 원천기술을 개발한 것이다”라고 강조했다.
우리 대학 공정렬 박사, 이춘경 박사과정 학생, 김훈민 박사과정 학생, 김주희 박사과정 학생 등이 참여한 이번 연구 결과는 와일리(Wiley)에서 출간하는 국제저널 `어드밴스드 사이언스(Advanced Science)' 12월 11일 字 온라인판 논문으로 출판됐다. (논문명: Control of cellular differentiation trajectories for cancer reversion) DOI: https://doi.org/10.1002/advs.202402132
한편 이번 연구는 과학기술정보통신부와 한국연구재단의 중견연구사업 및 기초연구실사업의 지원을 통해 수행되었으며 연구 성과는 바이오리버트(주)로 기술이전 되어 실제 암 가역치료제 개발에 활용될 예정이다.
2024.12.23 조회수 10468 -
 페트병 대체할 미생물 플라스틱 생산 성공하다
현재, 전 세계는 플라스틱 폐기물로 인한 환경 문제로 인해 큰 골머리를 앓고 있다. KAIST 연구진이 생분해성을 가지면서 기존 페트병을 대체할 미생물 기반의 플라스틱 생산에 성공해서 화제다.
우리 대학은 생명화학공학과 이상엽 특훈교수 연구팀이 시스템 대사공학을 이용해 PET(페트병) 대체 유사 방향족 폴리에스터 단량체를 고효율로 생산하는 미생물 균주 개발에 성공했다고 7일 밝혔다.
유사 방향족 다이카복실산은 고분자로 합성시 방향족 폴리에스터(PET)보다 나은 물성 및 높은 생분해성을 가지고 있어 친환경적인 고분자 단량체*로서 주목받고 있다. 화학적인 방법을 통한 유사 방향족 다이카복실산 생산은 낮은 수율과 선택성, 복잡한 반응 조건과 유해 폐기물 생성이라는 문제점을 지니고 있다.
*단량체: 고분자를 만드는 재료로 단량체를 서로 연결해 고분자를 합성함
이를 해결하기 위해 이상엽 특훈교수 연구팀은 대사공학을 활용, 아미노산 생산에 주로 사용되는 세균인 코리네박테리움에서 2-피론-4,6-다이카복실산과 4종의 피리딘 다이카복실산 (2,3-, 2,4-, 2,5-, 2,6-피리딘 다이카복실산)을 포함한 5종의 유사 방향족 다이카복실산을 고효율로 생산하는 미생물 균주를 개발했다.
연구팀은 대사공학 기법을 통해 여러 유사 방향족 다이카복실산의 전구체로 사용되는 프로토카테츄산의 대사 흐름을 강화하고 전구체의 손실을 방지하는 플랫폼 미생물 균주를 구축했다.
이를 기반으로 전사체 분석을 통해 유전자 조작 타겟을 발굴해 76.17g/L의 2-피론-4,6-다이카복실산을 생산하였고, 3종의 피리딘 다이카복실산 생산 대사회로를 신규 발굴 및 구축하여 2.79g/L의 2,3-피리딘 다이카복실산, 0.49g/L의 2,4-피리딘 다이카복실산, 1.42g/L의 2,5-피리딘 다이카복실산을 생산하는 데 성공했다.
또한, 연구팀은 2,6-피리딘 다이카복실산 생합성 경로 구축 및 강화를 통해 15.01g/L의 생산을 확인하며 총 5종의 유사 방향족 다이카복실산을 고효율로 생산하는 데 성공했다.
결론적으로, 2,4-, 2,5-, 2,6-피리딘 다이카복실산을 세계 최고 농도로 생산하는 데 성공하였다. 특히 2,4-, 2,5-피리딘 다이카복실산은 기존에 극미량 (mg/L) 생산되던 것을 g/L 규모의 생산까지 달성하였다.
이번 연구를 기반으로 다양한 폴리에스터 생산 산업공정으로의 응용이 기대되며, 유사 방향족 폴리에스터 생산에 관한 연구에도 적극 활용될 수 있으리라 기대된다.
교신저자인 이상엽 특훈교수는 “미생물을 기반으로 유사 방향족 폴리에스터 단량체를 고효율로 생산하는 친환경 기술을 개발했다는 점에 의의가 있다”며 “이번 연구가 앞으로 미생물 기반의 바이오 단량체 산업이 석유 화학 기반의 화학산업을 대체하는 데 일조할 것”이라고 밝혔다.
해당 연구 결과는 국제 학술지인 `미국 국립과학원 회보(PNAS)'에 10월 30일 자 게재됐다.
※ 논문명 : Metabolic engineering of Corynebacterium glutamicum for the production of pyrone and pyridine dicarboxylic acids
※ 저자 정보 : 조재성(한국과학기술원, 공동 제1저자), 찌웨이 루오(한국과학기술원, 공동 제1저자), 문천우(한국과학기술원, 공동 제1저자), Cindy Prabowo (한국과학기술원, 공동저자), 이상엽(한국과학기술원, 교신저자) 포함 총 5명
한편, 이번 연구는 과기정통부가 지원하는 석유대체 친환경 화학기술개발사업의 ‘바이오화학산업 선도를 위한 차세대 바이오리파이너리 원천기술 개발’ 과제(과제 책임자 이상엽 특훈교수)의 지원을 받아 수행됐다.
2024.11.07 조회수 10244
페트병 대체할 미생물 플라스틱 생산 성공하다
현재, 전 세계는 플라스틱 폐기물로 인한 환경 문제로 인해 큰 골머리를 앓고 있다. KAIST 연구진이 생분해성을 가지면서 기존 페트병을 대체할 미생물 기반의 플라스틱 생산에 성공해서 화제다.
우리 대학은 생명화학공학과 이상엽 특훈교수 연구팀이 시스템 대사공학을 이용해 PET(페트병) 대체 유사 방향족 폴리에스터 단량체를 고효율로 생산하는 미생물 균주 개발에 성공했다고 7일 밝혔다.
유사 방향족 다이카복실산은 고분자로 합성시 방향족 폴리에스터(PET)보다 나은 물성 및 높은 생분해성을 가지고 있어 친환경적인 고분자 단량체*로서 주목받고 있다. 화학적인 방법을 통한 유사 방향족 다이카복실산 생산은 낮은 수율과 선택성, 복잡한 반응 조건과 유해 폐기물 생성이라는 문제점을 지니고 있다.
*단량체: 고분자를 만드는 재료로 단량체를 서로 연결해 고분자를 합성함
이를 해결하기 위해 이상엽 특훈교수 연구팀은 대사공학을 활용, 아미노산 생산에 주로 사용되는 세균인 코리네박테리움에서 2-피론-4,6-다이카복실산과 4종의 피리딘 다이카복실산 (2,3-, 2,4-, 2,5-, 2,6-피리딘 다이카복실산)을 포함한 5종의 유사 방향족 다이카복실산을 고효율로 생산하는 미생물 균주를 개발했다.
연구팀은 대사공학 기법을 통해 여러 유사 방향족 다이카복실산의 전구체로 사용되는 프로토카테츄산의 대사 흐름을 강화하고 전구체의 손실을 방지하는 플랫폼 미생물 균주를 구축했다.
이를 기반으로 전사체 분석을 통해 유전자 조작 타겟을 발굴해 76.17g/L의 2-피론-4,6-다이카복실산을 생산하였고, 3종의 피리딘 다이카복실산 생산 대사회로를 신규 발굴 및 구축하여 2.79g/L의 2,3-피리딘 다이카복실산, 0.49g/L의 2,4-피리딘 다이카복실산, 1.42g/L의 2,5-피리딘 다이카복실산을 생산하는 데 성공했다.
또한, 연구팀은 2,6-피리딘 다이카복실산 생합성 경로 구축 및 강화를 통해 15.01g/L의 생산을 확인하며 총 5종의 유사 방향족 다이카복실산을 고효율로 생산하는 데 성공했다.
결론적으로, 2,4-, 2,5-, 2,6-피리딘 다이카복실산을 세계 최고 농도로 생산하는 데 성공하였다. 특히 2,4-, 2,5-피리딘 다이카복실산은 기존에 극미량 (mg/L) 생산되던 것을 g/L 규모의 생산까지 달성하였다.
이번 연구를 기반으로 다양한 폴리에스터 생산 산업공정으로의 응용이 기대되며, 유사 방향족 폴리에스터 생산에 관한 연구에도 적극 활용될 수 있으리라 기대된다.
교신저자인 이상엽 특훈교수는 “미생물을 기반으로 유사 방향족 폴리에스터 단량체를 고효율로 생산하는 친환경 기술을 개발했다는 점에 의의가 있다”며 “이번 연구가 앞으로 미생물 기반의 바이오 단량체 산업이 석유 화학 기반의 화학산업을 대체하는 데 일조할 것”이라고 밝혔다.
해당 연구 결과는 국제 학술지인 `미국 국립과학원 회보(PNAS)'에 10월 30일 자 게재됐다.
※ 논문명 : Metabolic engineering of Corynebacterium glutamicum for the production of pyrone and pyridine dicarboxylic acids
※ 저자 정보 : 조재성(한국과학기술원, 공동 제1저자), 찌웨이 루오(한국과학기술원, 공동 제1저자), 문천우(한국과학기술원, 공동 제1저자), Cindy Prabowo (한국과학기술원, 공동저자), 이상엽(한국과학기술원, 교신저자) 포함 총 5명
한편, 이번 연구는 과기정통부가 지원하는 석유대체 친환경 화학기술개발사업의 ‘바이오화학산업 선도를 위한 차세대 바이오리파이너리 원천기술 개발’ 과제(과제 책임자 이상엽 특훈교수)의 지원을 받아 수행됐다.
2024.11.07 조회수 10244 -
 암세포 약물반응 예측 ‘그레이박스’ 개발
지난 수십 년간 많은 의생명과학자의 집중적인 연구에도 불구하고 여전히 국내 사망원인 1위는 암이다. 이처럼 암 치료가 난해한 이유는 환자마다 암 발생의 원인이 되는 유전자 돌연변이와 그로 인한 유전자 네트워크 변형이 서로 달라서 전통적인 실험생물학 접근만으로 표적치료를 적용하는 데에는 본질적인 한계가 있기 때문이다. 한편 딥러닝과 같은 소위 블랙박스(black-box) 방식의 인공지능 기술을 활용해 실험을 대체하고 데이터 학습을 통해 약물 반응을 예측할 수 있으나 이에 대한 생물학적 근거를 설명할 수 없어 결과를 신뢰하기 어려웠다.
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 인공지능과 시스템생물학을 융합해 암세포의 약물 반응 예측 및 메커니즘 분석을 동시에 이룰 수 있는 새로운 개념의 ‘그레이박스’ 기술을 개발했다고 3일 밝혔다.
조광현 교수 연구팀은 높은 예측 성능을 보이지만 그 근거를 알 수 없어 블랙박스로 불리는 딥러닝과 복잡한 대규모 모델의 경우 예측 성능의 한계를 지니지만 예측 결과에 대한 상세한 근거를 제시할 수 있어서 화이트박스로 불리는 시스템생물학 기술을 융합함으로써 두 기술의 한계를 동시에 극복할 수 있는 소위 ‘그레이박스’ 기술을 착안했다.
연구팀은 다양한 암종의 돌연변이 및 표적항암제 타겟 유전자 정보를 집대성해 분자 조절 네트워크 모델을 구축함으로써 여러 암종과 항암제의 약물 반응 예측에 활용될 수 있는 범용적 골격 모델을 우선 정립했다. 특히 다양한 암종에서 돌연변이가 빈번하게 발생하는 유전자들을 중심으로 전암(pan-cancer) 유전자 네트워크를 제작했고 표적항암제별 약물 반응과 관련된 돌연변이 및 연관 유전자들로 구성된 부분네트워크(sub-network)를 추출함으로써 약물 반응 예측을 위한 시스템생물학 모델을 제작했다.
연구팀은 이렇게 제작된 모델의 매개변수를 딥러닝 블랙박스 최적화기를 통해 결정하는 방식으로 트라메티닙, 아파티닙, 팔보시클립 세 개의 표적항암제 및 대장암, 유방암, 위암 세 개의 암종에 대한 그레이박스 모델을 구축했다. 완성된 모델의 약물 반응 컴퓨터시뮬레이션 결과는 각 암종별 약물반응의 민감도 차이를 보이는 세포주(cancer cell lines) 실험을 통해 비교 검증됐다.
특히 개발된 모델은 미국 국립암연구소(National Cancer Institute)의 돌연변이 정보 기반 약물 반응 예측으로는 동일한 반응을 보일 것으로 예상된 암세포주들이 실제로는 서로 다른 약물 반응을 보일 수 있다는 것을 정확히 예측했으며, 약물 반응의 차이가 발생하는 원인 또한 세포 주별 분자 네트워크 동역학의 차이로 상세히 설명할 수 있었다.
이번 연구 성과는 학습에 의한 시뮬레이션 모델 최적화를 통해 블랙박스 모델인 인공지능 기술의 높은 예측력과 화이트박스 모델인 시스템생물학 기술의 해석력을 동시에 달성한 새로운 약물 반응 예측 기술 개발이어서 그 의미가 크다. 특히, 발생 원인이 이질적이고 복잡한 네트워크 질환인 암에 대해 범용적으로 활용가능한 약물 반응 예측 원천기술이므로 향후 기술 고도화를 통해 다양한 종류의 암종 및 환자 맞춤형 치료 전략 제시에 활용될 수 있을 것으로 기대된다.
조광현 교수는 "인공지능 기술의 높은 예측력과 시스템생물학 기술의 우수한 해석력을 동시에 갖춘 새로운 융합원천기술로서 향후 고도화를 통해 신약 개발 산업의 활용이 기대된다ˮ고 말했다.
바이오및뇌공학과 김윤성 박사, 한영현 박사 등이 참여한 이번 연구 결과는 셀 프레스(Cell Press)에서 출간하는 국제저널 `셀 리포트 메소드(Cell Reports Methods)' 5월 20일 字 표지논문으로 출판됐다. (논문명: A grey box framework that optimizes a white box logical model using a black box optimizer for simulating cellular responses to perturbations)
논문링크: https://www.cell.com/cell-reports-methods/fulltext/S2667-2375(24)00117-6
한편 이번 연구는 삼성미래기술육성사업 및 과학기술정보통신부와 한국연구재단의 중견연구사업 등의 지원으로 수행됐다.
2024.06.03 조회수 7500
암세포 약물반응 예측 ‘그레이박스’ 개발
지난 수십 년간 많은 의생명과학자의 집중적인 연구에도 불구하고 여전히 국내 사망원인 1위는 암이다. 이처럼 암 치료가 난해한 이유는 환자마다 암 발생의 원인이 되는 유전자 돌연변이와 그로 인한 유전자 네트워크 변형이 서로 달라서 전통적인 실험생물학 접근만으로 표적치료를 적용하는 데에는 본질적인 한계가 있기 때문이다. 한편 딥러닝과 같은 소위 블랙박스(black-box) 방식의 인공지능 기술을 활용해 실험을 대체하고 데이터 학습을 통해 약물 반응을 예측할 수 있으나 이에 대한 생물학적 근거를 설명할 수 없어 결과를 신뢰하기 어려웠다.
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 인공지능과 시스템생물학을 융합해 암세포의 약물 반응 예측 및 메커니즘 분석을 동시에 이룰 수 있는 새로운 개념의 ‘그레이박스’ 기술을 개발했다고 3일 밝혔다.
조광현 교수 연구팀은 높은 예측 성능을 보이지만 그 근거를 알 수 없어 블랙박스로 불리는 딥러닝과 복잡한 대규모 모델의 경우 예측 성능의 한계를 지니지만 예측 결과에 대한 상세한 근거를 제시할 수 있어서 화이트박스로 불리는 시스템생물학 기술을 융합함으로써 두 기술의 한계를 동시에 극복할 수 있는 소위 ‘그레이박스’ 기술을 착안했다.
연구팀은 다양한 암종의 돌연변이 및 표적항암제 타겟 유전자 정보를 집대성해 분자 조절 네트워크 모델을 구축함으로써 여러 암종과 항암제의 약물 반응 예측에 활용될 수 있는 범용적 골격 모델을 우선 정립했다. 특히 다양한 암종에서 돌연변이가 빈번하게 발생하는 유전자들을 중심으로 전암(pan-cancer) 유전자 네트워크를 제작했고 표적항암제별 약물 반응과 관련된 돌연변이 및 연관 유전자들로 구성된 부분네트워크(sub-network)를 추출함으로써 약물 반응 예측을 위한 시스템생물학 모델을 제작했다.
연구팀은 이렇게 제작된 모델의 매개변수를 딥러닝 블랙박스 최적화기를 통해 결정하는 방식으로 트라메티닙, 아파티닙, 팔보시클립 세 개의 표적항암제 및 대장암, 유방암, 위암 세 개의 암종에 대한 그레이박스 모델을 구축했다. 완성된 모델의 약물 반응 컴퓨터시뮬레이션 결과는 각 암종별 약물반응의 민감도 차이를 보이는 세포주(cancer cell lines) 실험을 통해 비교 검증됐다.
특히 개발된 모델은 미국 국립암연구소(National Cancer Institute)의 돌연변이 정보 기반 약물 반응 예측으로는 동일한 반응을 보일 것으로 예상된 암세포주들이 실제로는 서로 다른 약물 반응을 보일 수 있다는 것을 정확히 예측했으며, 약물 반응의 차이가 발생하는 원인 또한 세포 주별 분자 네트워크 동역학의 차이로 상세히 설명할 수 있었다.
이번 연구 성과는 학습에 의한 시뮬레이션 모델 최적화를 통해 블랙박스 모델인 인공지능 기술의 높은 예측력과 화이트박스 모델인 시스템생물학 기술의 해석력을 동시에 달성한 새로운 약물 반응 예측 기술 개발이어서 그 의미가 크다. 특히, 발생 원인이 이질적이고 복잡한 네트워크 질환인 암에 대해 범용적으로 활용가능한 약물 반응 예측 원천기술이므로 향후 기술 고도화를 통해 다양한 종류의 암종 및 환자 맞춤형 치료 전략 제시에 활용될 수 있을 것으로 기대된다.
조광현 교수는 "인공지능 기술의 높은 예측력과 시스템생물학 기술의 우수한 해석력을 동시에 갖춘 새로운 융합원천기술로서 향후 고도화를 통해 신약 개발 산업의 활용이 기대된다ˮ고 말했다.
바이오및뇌공학과 김윤성 박사, 한영현 박사 등이 참여한 이번 연구 결과는 셀 프레스(Cell Press)에서 출간하는 국제저널 `셀 리포트 메소드(Cell Reports Methods)' 5월 20일 字 표지논문으로 출판됐다. (논문명: A grey box framework that optimizes a white box logical model using a black box optimizer for simulating cellular responses to perturbations)
논문링크: https://www.cell.com/cell-reports-methods/fulltext/S2667-2375(24)00117-6
한편 이번 연구는 삼성미래기술육성사업 및 과학기술정보통신부와 한국연구재단의 중견연구사업 등의 지원으로 수행됐다.
2024.06.03 조회수 7500 -
 사회처럼 건강한 유전자 커뮤니티의 모습을 찾다
구성원들 사이의 활발한 교류로 결속력이 높은 사회적 커뮤니티가 건강한 개인을 만들 듯, 유전자 커뮤니티의 결속력도 개인의 건강 상태에 영향을 미칠 수 있을까? 한국 연구진이 유전자 커뮤니티의 결속력 또한 개인의 건강 상태를 결정하고 환자 맞춤형 의료를 위해 활용될 수 있음을 보여 화제다.
우리 대학 바이오및뇌공학과 이도헌 교수 연구팀이 개인화된 유전자 네트워크에서 환자 특이적으로 결속력이 약화된 유전자 커뮤니티를 찾아내 환자 맞춤형으로 약물 표적을 예측할 수 있는 기술을 개발했다고 23일 밝혔다.
최근 고령화와 생활 습관 변화 등에 따라 암, 심혈관계 질환, 대사 질환 등 많은 복합질병의 발병률이 크게 증가하는 실정이다. 이에 전문가들은 개별 환자의 특성을 고려한 ‘환자 맞춤형 의료’를 제공해 그 치료 효과를 높임으로써 개인적, 사회적 의료비 부담을 경감해야 한다고 지적한다.
이도헌 교수 연구팀은 이러한 요구에 발맞춰 개인화된 유전자 네트워크를 정교하게 구축하고 해당 네트워크에서 각 유전자 커뮤니티의 결속력을 정확하게 측정할 수 있는 코지넷(COSINET, COmmunity COhesion Scores in Individualized gene Network Estimated from single Transcripotmics data) 기술을 개발했다.
연구진들은 수백 개의 정상 조직 유전자 발현 데이터를 근거로 유의미한 상관관계를 보이는 유전자 상호작용을 기반으로 정상 조직의 유전자 네트워크를 구축했다. 그리고 유전자 커뮤니티들의 유전자 상호작용마다 보이는 상관관계를 선형 회귀 분석을 통해 모델링한 뒤, 개별 환자의 유전자 발현량이 해당 예측 모델을 잘 따르는지를 통계적으로 분석했다. 이를 통해 환자 특이적으로 그 상호작용이 소실된 유전자 쌍을 정상 조직 유전자 네트워크에서 제거함으로써 개인화된 유전자 네트워크를 구축했다.
더 나아가 개인화된 유전자 네트워크에서 유전자들 사이의 최단 거리를 기반으로 소실된 유전자 상호작용이 각 유전자 커뮤니티 결속력 약화에 미치는 영향력을 정확하게 측정했다.
연구진들은 환자 특이적으로 그 결속력이 크게 감소한 유전자 커뮤니티를 통해 환자 특이적인 질병 기전을 설명할 수 있음을 보이고, 해당 유전자 커뮤니티에서 환자 특이적으로 결속력 약화에 크게 기여하는 유전자들을 찾아, 보다 효과적인 환자 맞춤형 약물 표적을 제안했다. 연구진들은 이러한 약물 표적 발굴 기술이 기존 기술 대비 약 4배 이상 효과적임을 증명했다.
이도헌 교수는 “여러 유전자가 관여하는 복합질병은 개별 유전자보다는 유전자들 사이의 상호작용을 고려하는 시스템적 관점에서 바라봐야 하며 현재 임상 현장에서 환자 맞춤형 의료를 위해 쓰이는 단일 유전자 기반의 바이오마커들은 복합질병의 이질성과 복잡성을 충분히 담아내기에는 한계가 있다. 따라서 이번 연구에서 개발한 개인화된 유전자 네트워크에서 유전자 커뮤니티의 결속력에 기반한 코지넷(COSINET) 기술이 복합질병의 환자 맞춤형 의료 실현을 위한 새로운 시각을 열어 줄 수 있을 것”이라고 말했다.
바이오및뇌공학과 이도헌 교수와 왕승현 박사과정이 공동으로 진행한 이번 연구는 영국 옥스퍼드대학교에서 발간하는 생명정보학 분야 최고 학술지인 `생명정보학 브리핑(Briefings in Bioinformatics)’ 2024년 5월호에 게재되고 온라인으로는 4월 15일 발표됐다.
(논문 제목: Community cohesion looseness in gene networks reveals individualized drug targets and resistance, https://academic.oup.com/bib/article/25/3/bbae175/7645997)
한편 이번 연구는 과학기술정보통신부 데이터 기반 디지털 바이오 선도 사업의 지원을 받아 수행됐다.
2024.04.23 조회수 6607
사회처럼 건강한 유전자 커뮤니티의 모습을 찾다
구성원들 사이의 활발한 교류로 결속력이 높은 사회적 커뮤니티가 건강한 개인을 만들 듯, 유전자 커뮤니티의 결속력도 개인의 건강 상태에 영향을 미칠 수 있을까? 한국 연구진이 유전자 커뮤니티의 결속력 또한 개인의 건강 상태를 결정하고 환자 맞춤형 의료를 위해 활용될 수 있음을 보여 화제다.
우리 대학 바이오및뇌공학과 이도헌 교수 연구팀이 개인화된 유전자 네트워크에서 환자 특이적으로 결속력이 약화된 유전자 커뮤니티를 찾아내 환자 맞춤형으로 약물 표적을 예측할 수 있는 기술을 개발했다고 23일 밝혔다.
최근 고령화와 생활 습관 변화 등에 따라 암, 심혈관계 질환, 대사 질환 등 많은 복합질병의 발병률이 크게 증가하는 실정이다. 이에 전문가들은 개별 환자의 특성을 고려한 ‘환자 맞춤형 의료’를 제공해 그 치료 효과를 높임으로써 개인적, 사회적 의료비 부담을 경감해야 한다고 지적한다.
이도헌 교수 연구팀은 이러한 요구에 발맞춰 개인화된 유전자 네트워크를 정교하게 구축하고 해당 네트워크에서 각 유전자 커뮤니티의 결속력을 정확하게 측정할 수 있는 코지넷(COSINET, COmmunity COhesion Scores in Individualized gene Network Estimated from single Transcripotmics data) 기술을 개발했다.
연구진들은 수백 개의 정상 조직 유전자 발현 데이터를 근거로 유의미한 상관관계를 보이는 유전자 상호작용을 기반으로 정상 조직의 유전자 네트워크를 구축했다. 그리고 유전자 커뮤니티들의 유전자 상호작용마다 보이는 상관관계를 선형 회귀 분석을 통해 모델링한 뒤, 개별 환자의 유전자 발현량이 해당 예측 모델을 잘 따르는지를 통계적으로 분석했다. 이를 통해 환자 특이적으로 그 상호작용이 소실된 유전자 쌍을 정상 조직 유전자 네트워크에서 제거함으로써 개인화된 유전자 네트워크를 구축했다.
더 나아가 개인화된 유전자 네트워크에서 유전자들 사이의 최단 거리를 기반으로 소실된 유전자 상호작용이 각 유전자 커뮤니티 결속력 약화에 미치는 영향력을 정확하게 측정했다.
연구진들은 환자 특이적으로 그 결속력이 크게 감소한 유전자 커뮤니티를 통해 환자 특이적인 질병 기전을 설명할 수 있음을 보이고, 해당 유전자 커뮤니티에서 환자 특이적으로 결속력 약화에 크게 기여하는 유전자들을 찾아, 보다 효과적인 환자 맞춤형 약물 표적을 제안했다. 연구진들은 이러한 약물 표적 발굴 기술이 기존 기술 대비 약 4배 이상 효과적임을 증명했다.
이도헌 교수는 “여러 유전자가 관여하는 복합질병은 개별 유전자보다는 유전자들 사이의 상호작용을 고려하는 시스템적 관점에서 바라봐야 하며 현재 임상 현장에서 환자 맞춤형 의료를 위해 쓰이는 단일 유전자 기반의 바이오마커들은 복합질병의 이질성과 복잡성을 충분히 담아내기에는 한계가 있다. 따라서 이번 연구에서 개발한 개인화된 유전자 네트워크에서 유전자 커뮤니티의 결속력에 기반한 코지넷(COSINET) 기술이 복합질병의 환자 맞춤형 의료 실현을 위한 새로운 시각을 열어 줄 수 있을 것”이라고 말했다.
바이오및뇌공학과 이도헌 교수와 왕승현 박사과정이 공동으로 진행한 이번 연구는 영국 옥스퍼드대학교에서 발간하는 생명정보학 분야 최고 학술지인 `생명정보학 브리핑(Briefings in Bioinformatics)’ 2024년 5월호에 게재되고 온라인으로는 4월 15일 발표됐다.
(논문 제목: Community cohesion looseness in gene networks reveals individualized drug targets and resistance, https://academic.oup.com/bib/article/25/3/bbae175/7645997)
한편 이번 연구는 과학기술정보통신부 데이터 기반 디지털 바이오 선도 사업의 지원을 받아 수행됐다.
2024.04.23 조회수 6607 -
 암세포만 골라 유전자 교정 치료하는 신약 개발
최근 크리스퍼(유전자 가위) 기술을 활용한 유전자 교정 치료제 연구가 활발하다. 기존 화학적 항암치료제와는 달리 크리스퍼 기술 기반 유전자 교정 치료제는 질병 표적 유전자를 영구적으로 교정할 수 있어 암 및 유전 질환 치료제로 각광받고 있지만, 생체 내에서 암 조직으로 낮은 전달 효율과 낮은 효능으로 어려움을 겪고 있다.
우리 대학 생명과학과 정현정 교수 연구팀이 크리스퍼 기반 표적 치료제로 항체를 이용한 크리스퍼 단백질을 생체 내 표적 조직에 특이적으로 전달하는 항암 신약을 개발해 암세포 선택적 유전자 교정 및 항암 효능을 보였다고 8일 밝혔다.
유전자 치료에 사용하는 바이러스 기반 전달 방법은 인체 내 면역 부작용, 발암성 등 한계점을 가지고 있다. 이에 선호되는 비 바이러스성 전달 방법으로 단백질 기반의 크리스퍼 기술 전달은 본래의 표적과는 다른 분자를 저해 혹은 활성화하는 효과를 가져오는 오프타깃 효과가 최소화되며 보다 높은 안전성으로 치료제로서 개발이 적합하다. 하지만 크리스퍼 단백질은 분자량이 커서 전달체에 탑재가 어렵고 전달체의 세포 독성 문제 및 낮은 표적 세포로의 전달에 있어 어려움이 있다.
이러한 문제점들을 극복하기 위해 연구팀은 크리스퍼 단백질에 특정 아미노산을 변경시켜 다양한 생체분자를 보다 많이 결합시키고 생체 내 본질적인 생화학 과정을 방해하지 않는 단백질을 개발했다. 연구팀은 기존 비 바이러스성 전달체의 문제 해결 및 표적 세포로의 전달을 위해 개량한 크리스퍼 단백질을 난소암을 표적할 수 있는 항체와 결합함으로써 표적 치료제를 위한 항체 결합 크리스퍼 나노복합체(⍺Her-CrNC, anti-Her2 conjugated CRISPR nanocomplex)를 개발했다.
암세포 표면은 종양 항원(tumor antigen)으로 알려진 항원이 존재한다. 몇몇 종양 항원은 표적이 되어 진단 및 임상시험에 이용되고 있다. 연구팀은 개발한 항체 결합 크리스퍼 나노복합체가 종양 항원을 표적해 난소암세포 및 동물모델에서 암세포 특이적으로 세포 내 전달이 가능하고 세포주기를 관장하는 PLK1* 유전자 교정을 통해 높은 항암효과가 나타남을 확인했다.
* PLK1(polo-like kinase): 세포 분열을 조절하는 인산화효소이며, 암세포 분열과 관련이 깊다고 알려져 있음. 본 연구에서는 PLK1 유전자를 표적하여 암세포 분열을 억제하여 항암 효과를 유도하였음
연구를 주도한 정현정 교수는 “이번 연구는 최초로 크리스퍼 단백질과 항체를 결합해 효과적으로 암세포 특이적 전달 및 항암 효능을 보였다는 점에서 의의가 있다. 아울러, 이번 연구 결과를 기반으로 향후 생체 내 전신 투여를 통한 유전자 교정 치료 및 다양한 암종에 적용할 수 있는 플랫폼 기술로 기대하고 있다”고 말했다.
우리 대학 생명과학과 석박사통합과정 양승주 학생이 제1 저자로 참여한 이번 연구는 국제학술지 `어드밴스드 사이언스(Advanced Science)'에 3월 29일 온라인 게재됐다. (논문명: An Antibody-CRISPR/Cas Conjugate Platform for Target-Specific Delivery and Gene Editing in Cancer)
한편 이번 연구는 과학기술정보통신부와 한국연구재단 및 보건복지부의 지원을 통해 이뤄졌다.
2024.04.08 조회수 9832
암세포만 골라 유전자 교정 치료하는 신약 개발
최근 크리스퍼(유전자 가위) 기술을 활용한 유전자 교정 치료제 연구가 활발하다. 기존 화학적 항암치료제와는 달리 크리스퍼 기술 기반 유전자 교정 치료제는 질병 표적 유전자를 영구적으로 교정할 수 있어 암 및 유전 질환 치료제로 각광받고 있지만, 생체 내에서 암 조직으로 낮은 전달 효율과 낮은 효능으로 어려움을 겪고 있다.
우리 대학 생명과학과 정현정 교수 연구팀이 크리스퍼 기반 표적 치료제로 항체를 이용한 크리스퍼 단백질을 생체 내 표적 조직에 특이적으로 전달하는 항암 신약을 개발해 암세포 선택적 유전자 교정 및 항암 효능을 보였다고 8일 밝혔다.
유전자 치료에 사용하는 바이러스 기반 전달 방법은 인체 내 면역 부작용, 발암성 등 한계점을 가지고 있다. 이에 선호되는 비 바이러스성 전달 방법으로 단백질 기반의 크리스퍼 기술 전달은 본래의 표적과는 다른 분자를 저해 혹은 활성화하는 효과를 가져오는 오프타깃 효과가 최소화되며 보다 높은 안전성으로 치료제로서 개발이 적합하다. 하지만 크리스퍼 단백질은 분자량이 커서 전달체에 탑재가 어렵고 전달체의 세포 독성 문제 및 낮은 표적 세포로의 전달에 있어 어려움이 있다.
이러한 문제점들을 극복하기 위해 연구팀은 크리스퍼 단백질에 특정 아미노산을 변경시켜 다양한 생체분자를 보다 많이 결합시키고 생체 내 본질적인 생화학 과정을 방해하지 않는 단백질을 개발했다. 연구팀은 기존 비 바이러스성 전달체의 문제 해결 및 표적 세포로의 전달을 위해 개량한 크리스퍼 단백질을 난소암을 표적할 수 있는 항체와 결합함으로써 표적 치료제를 위한 항체 결합 크리스퍼 나노복합체(⍺Her-CrNC, anti-Her2 conjugated CRISPR nanocomplex)를 개발했다.
암세포 표면은 종양 항원(tumor antigen)으로 알려진 항원이 존재한다. 몇몇 종양 항원은 표적이 되어 진단 및 임상시험에 이용되고 있다. 연구팀은 개발한 항체 결합 크리스퍼 나노복합체가 종양 항원을 표적해 난소암세포 및 동물모델에서 암세포 특이적으로 세포 내 전달이 가능하고 세포주기를 관장하는 PLK1* 유전자 교정을 통해 높은 항암효과가 나타남을 확인했다.
* PLK1(polo-like kinase): 세포 분열을 조절하는 인산화효소이며, 암세포 분열과 관련이 깊다고 알려져 있음. 본 연구에서는 PLK1 유전자를 표적하여 암세포 분열을 억제하여 항암 효과를 유도하였음
연구를 주도한 정현정 교수는 “이번 연구는 최초로 크리스퍼 단백질과 항체를 결합해 효과적으로 암세포 특이적 전달 및 항암 효능을 보였다는 점에서 의의가 있다. 아울러, 이번 연구 결과를 기반으로 향후 생체 내 전신 투여를 통한 유전자 교정 치료 및 다양한 암종에 적용할 수 있는 플랫폼 기술로 기대하고 있다”고 말했다.
우리 대학 생명과학과 석박사통합과정 양승주 학생이 제1 저자로 참여한 이번 연구는 국제학술지 `어드밴스드 사이언스(Advanced Science)'에 3월 29일 온라인 게재됐다. (논문명: An Antibody-CRISPR/Cas Conjugate Platform for Target-Specific Delivery and Gene Editing in Cancer)
한편 이번 연구는 과학기술정보통신부와 한국연구재단 및 보건복지부의 지원을 통해 이뤄졌다.
2024.04.08 조회수 9832 -
 암 유발 물질 컴퓨터로 예측하다
암은 정상세포와 다르게 세포 내 비정상적인 축적을 통해 유발되는 대사 반응을 하며, 암의 치료 및 진단을 목적으로 이런 암 대사반응에 대해 다방면으로 연구되고 있다. 이에 우리 대학 연구진이 컴퓨터를 통해 24개 암종에 해당하는 1,043명의 암 환자에 대한 대사 모델 구축에 성공했다.
우리 대학 생명화학공학과 김현욱 교수, 이상엽 특훈교수 연구팀이 서울대학교병원 고영일 교수, 윤홍석 교수 및 정창욱 교수 연구팀과의 공동연구를 통해, 암 체세포 유전자 돌연변이와 연관된 새로운 대사물질 및 대사경로를 예측하는 컴퓨터 방법론을 개발했다고 18일 밝혔다.
최근 암 유발 대사물질(oncometabolite)*의 발견과 이를 표적으로 하는 신약들이 미국식품의약국(FDA)의 승인을 받으며 주목받고 있는데, 이에는 급성 골수성 백혈병의 치료제로 사용되고 있는 ‘팁소보(성분명: 아이보시데닙)’ 및 약물 ‘아이드하이파(성분명: 에나시데닙)’가 포함된다.
*암 유발 대사물질 (oncometabolite): 세포 내 비정상적인 축적을 통해 암을 유발하는 대사물질. 이러한 대사물질들은 특정 유전자 돌연변이의 영향으로 대사 과정 중에 비정상적으로 높은 농도로 축적되며, 이러한 축적은 암세포의 성장과 생존을 촉진함. 기존 연구에서 확인된 주요 암 유발 대사물질로는 2-하이드록시글루타레이트(2-hydroxyglutarate), 숙시네이트(succinate), 푸마레이트(fumarate) 등이 보고됨.
하지만, 암 대사 연구와 새로운 암 유발 대사물질 발굴에는 대사체학 등의 방법론이 필요하며, 이를 대규모 환자 샘플에 적용하기 위해서는 상당한 시간과 비용이 소요된다. 이러한 이유로, 암과 관련된 많은 유전자 돌연변이들이 밝혀졌음에도, 그에 상응하는 암 유발 대사물질은 극소수만 알려져 있다.
김현욱 교수 공동연구팀은 세포 대사 정보를 예측할 수 있는 게놈 수준의 대사 모델*에 국제 암 연구 컨소시엄에서 공개하고 있는 암 환자들의 전사체 데이터를 통합해, 24개 암종에 해당하는 1,043명의 암 환자에 대한 대사 모델을 성공적으로 구축했다.
*게놈 수준의 대사모델: 세포의 전체 대사 네트워크를 다루는 컴퓨터 모델로서, 세포 내 모든 대사반응에 대한 정보가 담겨 있으며, 다양한 조건에서 세포의 대사 활성을 예측하는 것이 가능
공동연구팀은 1,043명의 암 환자 특이 대사 모델과 동일 환자들의 암 체세포 돌연변이 데이터를 활용해, 다음의 4단계로 구성된 컴퓨터 방법론을 개발했다 (그림 1). 첫 단계에서는 암 환자 특이 대사 모델을 시뮬레이션해, 환자 별로 모든 대사물질들의 활성을 예측한다. 두 번째 단계로는 특정 유전자 돌연변이가 앞서 예측된 대사물질의 활성에 유의한 차이를 일으키는 짝을 선별한다. 세 번째 단계로, 특정 유전자 돌연변이와 연결된 대사물질들을 대상으로, 이들과 유의하게 연관된 대사경로를 추가로 선별한다. 마지막 단계로서, ‘유전자-대사물질-대사경로’ 조합을 완성해, 컴퓨터 방법론 결과로써 도출하게 된다.
이번 논문의 공동 제1 저자인 이가령 박사(現 다나파버 암센터 및 하버드 의과대학 박사후연구원)와 이상미 박사(現 하버드 의과대학 박사후연구원)는 “이번 연구에서 개발된 방법론은 암 환자 코호트의 돌연변이 및 전사체 데이터를 토대로 다른 암종에 대해서도 쉽게 적용될 수 있으며, 유전자 돌연변이가 대사경로를 통해 어떻게 세포대사에 변화를 일으키는지 체계적으로 예측할 수 있는 최초의 컴퓨터 방법론이라는 데 큰 의의가 있다” 한다고 말했다.
또한 김현욱 교수는 “이번 공동연구의 결과는 향후 암 대사 및 암 유발 대사물질 연구에서 중요한 참고 자료로 활용될 수 있을 것”이라고 강조했다.
한편 이번 논문은 바이오메드 센트럴(BioMed Central) 社가 발행하며, 생명공학 및 유전학 분야의 대표적 국제학술지인 게놈 바이올로지(Genome Biology, JCR 분야 상위 5% 이내)에 게재됐다.
※ 논문명 : Prediction of metabolites associated with somatic mutations in cancers by using genome-scale metabolic models and mutation data
※ 저자 정보 : 이가령(한국과학기술원, 공동 제1 저자), 이상미(한국과학기술원, 공동 제1 저자), 이성영(서울대학교병원, 공동저자), 정창욱(서울대학교병원, 공동저자), 송효진(서울대학교병원, 공동저자), 이상엽(한국과학기술원, 공동저자), 윤홍석(서울대학교병원, 교신저자), 고영일(서울대학교병원, 교신저자), 김현욱(한국과학기술원, 교신저자) 포함 총 9명
이번 연구는 과학기술정보통신부 한국연구재단의 지원을 받아 수행됐다.
2024.03.18 조회수 7417
암 유발 물질 컴퓨터로 예측하다
암은 정상세포와 다르게 세포 내 비정상적인 축적을 통해 유발되는 대사 반응을 하며, 암의 치료 및 진단을 목적으로 이런 암 대사반응에 대해 다방면으로 연구되고 있다. 이에 우리 대학 연구진이 컴퓨터를 통해 24개 암종에 해당하는 1,043명의 암 환자에 대한 대사 모델 구축에 성공했다.
우리 대학 생명화학공학과 김현욱 교수, 이상엽 특훈교수 연구팀이 서울대학교병원 고영일 교수, 윤홍석 교수 및 정창욱 교수 연구팀과의 공동연구를 통해, 암 체세포 유전자 돌연변이와 연관된 새로운 대사물질 및 대사경로를 예측하는 컴퓨터 방법론을 개발했다고 18일 밝혔다.
최근 암 유발 대사물질(oncometabolite)*의 발견과 이를 표적으로 하는 신약들이 미국식품의약국(FDA)의 승인을 받으며 주목받고 있는데, 이에는 급성 골수성 백혈병의 치료제로 사용되고 있는 ‘팁소보(성분명: 아이보시데닙)’ 및 약물 ‘아이드하이파(성분명: 에나시데닙)’가 포함된다.
*암 유발 대사물질 (oncometabolite): 세포 내 비정상적인 축적을 통해 암을 유발하는 대사물질. 이러한 대사물질들은 특정 유전자 돌연변이의 영향으로 대사 과정 중에 비정상적으로 높은 농도로 축적되며, 이러한 축적은 암세포의 성장과 생존을 촉진함. 기존 연구에서 확인된 주요 암 유발 대사물질로는 2-하이드록시글루타레이트(2-hydroxyglutarate), 숙시네이트(succinate), 푸마레이트(fumarate) 등이 보고됨.
하지만, 암 대사 연구와 새로운 암 유발 대사물질 발굴에는 대사체학 등의 방법론이 필요하며, 이를 대규모 환자 샘플에 적용하기 위해서는 상당한 시간과 비용이 소요된다. 이러한 이유로, 암과 관련된 많은 유전자 돌연변이들이 밝혀졌음에도, 그에 상응하는 암 유발 대사물질은 극소수만 알려져 있다.
김현욱 교수 공동연구팀은 세포 대사 정보를 예측할 수 있는 게놈 수준의 대사 모델*에 국제 암 연구 컨소시엄에서 공개하고 있는 암 환자들의 전사체 데이터를 통합해, 24개 암종에 해당하는 1,043명의 암 환자에 대한 대사 모델을 성공적으로 구축했다.
*게놈 수준의 대사모델: 세포의 전체 대사 네트워크를 다루는 컴퓨터 모델로서, 세포 내 모든 대사반응에 대한 정보가 담겨 있으며, 다양한 조건에서 세포의 대사 활성을 예측하는 것이 가능
공동연구팀은 1,043명의 암 환자 특이 대사 모델과 동일 환자들의 암 체세포 돌연변이 데이터를 활용해, 다음의 4단계로 구성된 컴퓨터 방법론을 개발했다 (그림 1). 첫 단계에서는 암 환자 특이 대사 모델을 시뮬레이션해, 환자 별로 모든 대사물질들의 활성을 예측한다. 두 번째 단계로는 특정 유전자 돌연변이가 앞서 예측된 대사물질의 활성에 유의한 차이를 일으키는 짝을 선별한다. 세 번째 단계로, 특정 유전자 돌연변이와 연결된 대사물질들을 대상으로, 이들과 유의하게 연관된 대사경로를 추가로 선별한다. 마지막 단계로서, ‘유전자-대사물질-대사경로’ 조합을 완성해, 컴퓨터 방법론 결과로써 도출하게 된다.
이번 논문의 공동 제1 저자인 이가령 박사(現 다나파버 암센터 및 하버드 의과대학 박사후연구원)와 이상미 박사(現 하버드 의과대학 박사후연구원)는 “이번 연구에서 개발된 방법론은 암 환자 코호트의 돌연변이 및 전사체 데이터를 토대로 다른 암종에 대해서도 쉽게 적용될 수 있으며, 유전자 돌연변이가 대사경로를 통해 어떻게 세포대사에 변화를 일으키는지 체계적으로 예측할 수 있는 최초의 컴퓨터 방법론이라는 데 큰 의의가 있다” 한다고 말했다.
또한 김현욱 교수는 “이번 공동연구의 결과는 향후 암 대사 및 암 유발 대사물질 연구에서 중요한 참고 자료로 활용될 수 있을 것”이라고 강조했다.
한편 이번 논문은 바이오메드 센트럴(BioMed Central) 社가 발행하며, 생명공학 및 유전학 분야의 대표적 국제학술지인 게놈 바이올로지(Genome Biology, JCR 분야 상위 5% 이내)에 게재됐다.
※ 논문명 : Prediction of metabolites associated with somatic mutations in cancers by using genome-scale metabolic models and mutation data
※ 저자 정보 : 이가령(한국과학기술원, 공동 제1 저자), 이상미(한국과학기술원, 공동 제1 저자), 이성영(서울대학교병원, 공동저자), 정창욱(서울대학교병원, 공동저자), 송효진(서울대학교병원, 공동저자), 이상엽(한국과학기술원, 공동저자), 윤홍석(서울대학교병원, 교신저자), 고영일(서울대학교병원, 교신저자), 김현욱(한국과학기술원, 교신저자) 포함 총 9명
이번 연구는 과학기술정보통신부 한국연구재단의 지원을 받아 수행됐다.
2024.03.18 조회수 7417 -
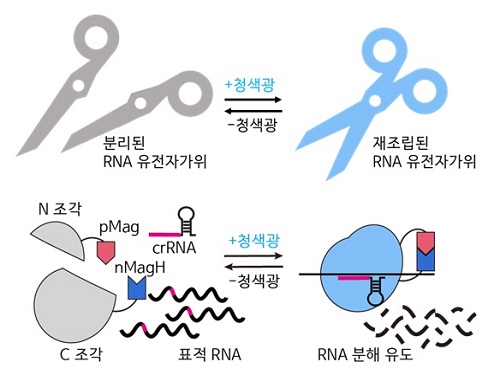 RNA 유전자 가위 정밀제어기술로 유전자 치료 성큼
최근 유전자 치료제 개발에 있어 중요한 역할을 하는 유전자 가위(CRISPR/Cas) 기술은 DNA 편집을 통해 영구적인 치료 효과를 보일 수 있으나, 비표적 효과에 의한 생체 내 부작용에 의한 돌연변이가 발생하였을 때, 대체할 방안이 불명확하다. DNA 편집의 잠재적인 위험성을 극복하여 특이적으로 인식하고 조절할 수 있는 RNA 대상으로 하는 유전자 가위 시스템이 주목받고 있다.
우리 대학 생명과학과 허원도 교수 연구팀이 세계 최초로 RNA 유전자 가위 기술 (CRISPR/Cas13)의 활성을 화학 유전학 및 광유전학으로 조절해 시간 및 공간적으로 표적 RNA의 염기 편집을 수행하는 기술을 개발했고, 동물 모델에서의 RNA 염기 편집 효과를 입증했다고 7일 밝혔다.
허원도 교수 연구팀은 구조가 알려지지 않은 단백질의 구조를 재구조화해, 화학적 및 광유전학적으로 조절 가능한 Cas13 단백질 조각을 예측하고 개발하는 데 성공했다. 이를 통해 개발된 에디터 기술로 RNA 분해 및 RNA 염기 편집을 실시간으로 유도할 수 있으며, RNA 염기 편집의 활성을 가역적으로 조절할 수 있음을 확인했다. 또한, 기존 연구자들이 실험에 이용하던 세포모델에서 더 나아가 세계 최초로 실험 쥐 모델에 해당 시스템을 적용해 광유전학적으로 RNA 염기 편집이 효과적으로 일어나는 것을 입증했다.
이번 연구는 유전자 가위 시스템을 활용한 유도 가능한 RNA 조절 시스템 개발로, 질병과 관련된 돌연변이를 표적으로 하는 RNA 기반 치료법의 발전 및 세포 내 RNA 기반 연구의 적용에 기여할 것으로 기대된다. 특히 생체 내 전달 목적으로 주로 사용되는데 연구팀은 RNA 대상 편집 시스템에서 단백질의 상대적으로 큰 크기를 유전체 전달에 있어서 임상적 적용에 한계점을 가지고 있다는 점을 감안하여 DNA 크기 제한을 분할 시스템으로 극복하고, 실험 쥐의 기관 내에서 다양한 모델 시스템 구축을 통해 생체 내 RNA 연구의 적용 범위를 확장할 수 있다.
연구를 주도한 허원도 교수는 “재결합이 가능한 분할 단백질 Cas13 조각을 개발해, 화학적 및 광유전학적으로 특정 시공간에서 정밀하게 조절되는 RNA를 실험적으로 확인했다. 이 기술은 그동안 실험적 한계로 인해 어려웠던 복잡한 RNA 연구를 촉진할 것으로 기대된다.라고 말했다.” 아울러 “유전자 가위 시스템을 활용한 유도 가능한 RNA 조절 시스템 개발로, 질병과 관련된 돌연변이를 표적으로 하는 RNA 기반 치료법의 발전 및 세포 내 RNA 기반 연구의 적용에 기여할 것으로 기대된다”라고 전했다.
우리 대학 생명과학과 유정혜 박사과정이 제1 저자로 수행한 이번 연구는 저명 국제 학술지 ‘네이처 커뮤니케이션즈 (Nature Communications)’ 2024년 1월 22일 字 온라인판에 게재됐다. (논문명: Programmable RNA base editing with photoactivatable CRISPR-Cas13). (Impact Factor: 17.694). (DOI: https://doi.org/10.1038/s41467-024-44867-2)
한편, 이번 연구는 삼성미래기술육성재단과 정부의 재원으로 한국연구재단 바이오·의료기술개발사업의 지원을 받아 수행됐다.
2024.02.07 조회수 9951
RNA 유전자 가위 정밀제어기술로 유전자 치료 성큼
최근 유전자 치료제 개발에 있어 중요한 역할을 하는 유전자 가위(CRISPR/Cas) 기술은 DNA 편집을 통해 영구적인 치료 효과를 보일 수 있으나, 비표적 효과에 의한 생체 내 부작용에 의한 돌연변이가 발생하였을 때, 대체할 방안이 불명확하다. DNA 편집의 잠재적인 위험성을 극복하여 특이적으로 인식하고 조절할 수 있는 RNA 대상으로 하는 유전자 가위 시스템이 주목받고 있다.
우리 대학 생명과학과 허원도 교수 연구팀이 세계 최초로 RNA 유전자 가위 기술 (CRISPR/Cas13)의 활성을 화학 유전학 및 광유전학으로 조절해 시간 및 공간적으로 표적 RNA의 염기 편집을 수행하는 기술을 개발했고, 동물 모델에서의 RNA 염기 편집 효과를 입증했다고 7일 밝혔다.
허원도 교수 연구팀은 구조가 알려지지 않은 단백질의 구조를 재구조화해, 화학적 및 광유전학적으로 조절 가능한 Cas13 단백질 조각을 예측하고 개발하는 데 성공했다. 이를 통해 개발된 에디터 기술로 RNA 분해 및 RNA 염기 편집을 실시간으로 유도할 수 있으며, RNA 염기 편집의 활성을 가역적으로 조절할 수 있음을 확인했다. 또한, 기존 연구자들이 실험에 이용하던 세포모델에서 더 나아가 세계 최초로 실험 쥐 모델에 해당 시스템을 적용해 광유전학적으로 RNA 염기 편집이 효과적으로 일어나는 것을 입증했다.
이번 연구는 유전자 가위 시스템을 활용한 유도 가능한 RNA 조절 시스템 개발로, 질병과 관련된 돌연변이를 표적으로 하는 RNA 기반 치료법의 발전 및 세포 내 RNA 기반 연구의 적용에 기여할 것으로 기대된다. 특히 생체 내 전달 목적으로 주로 사용되는데 연구팀은 RNA 대상 편집 시스템에서 단백질의 상대적으로 큰 크기를 유전체 전달에 있어서 임상적 적용에 한계점을 가지고 있다는 점을 감안하여 DNA 크기 제한을 분할 시스템으로 극복하고, 실험 쥐의 기관 내에서 다양한 모델 시스템 구축을 통해 생체 내 RNA 연구의 적용 범위를 확장할 수 있다.
연구를 주도한 허원도 교수는 “재결합이 가능한 분할 단백질 Cas13 조각을 개발해, 화학적 및 광유전학적으로 특정 시공간에서 정밀하게 조절되는 RNA를 실험적으로 확인했다. 이 기술은 그동안 실험적 한계로 인해 어려웠던 복잡한 RNA 연구를 촉진할 것으로 기대된다.라고 말했다.” 아울러 “유전자 가위 시스템을 활용한 유도 가능한 RNA 조절 시스템 개발로, 질병과 관련된 돌연변이를 표적으로 하는 RNA 기반 치료법의 발전 및 세포 내 RNA 기반 연구의 적용에 기여할 것으로 기대된다”라고 전했다.
우리 대학 생명과학과 유정혜 박사과정이 제1 저자로 수행한 이번 연구는 저명 국제 학술지 ‘네이처 커뮤니케이션즈 (Nature Communications)’ 2024년 1월 22일 字 온라인판에 게재됐다. (논문명: Programmable RNA base editing with photoactivatable CRISPR-Cas13). (Impact Factor: 17.694). (DOI: https://doi.org/10.1038/s41467-024-44867-2)
한편, 이번 연구는 삼성미래기술육성재단과 정부의 재원으로 한국연구재단 바이오·의료기술개발사업의 지원을 받아 수행됐다.
2024.02.07 조회수 9951 -
 숨겨진 효소 쏙쏙 찾아내는 인공지능 개발
대장균은 가장 많이 연구된 생명체 중 하나에 해당되지만 아직 대장균을 구성한 단백질 30%의 기능에 대해 명확하게 밝혀지지 않았다. 이에 대해 인공지능을 활용하여 아직 명확하게 밝혀진 바 없던 단백질에서 464종의 효소를 발견하였으며, 이 중 3종의 단백질의 예측된 기능을 시험관 내 효소 분석 방법을 통해 검증하는데 성공하였다.
우리 대학 생명화학공학과 이상엽 특훈교수와 캘리포니아대학교 샌디에이고(UCSD) 생명공학과 버나드 펄슨(Bernhard Palsson) 교수 공동연구팀이 단백질 서열을 활용, 해당 단백질의 효소 기능을 예측할 수 있는 인공지능, `딥 EC 트랜스포머(DeepECtransformer)'를 개발해 빠르고 정확하게 효소 기능을 파악할 수 있는 예측 시스템을 구축했다고 24일 밝혔다.
효소는 생물학적 반응을 촉매하는 단백질로서, 생명체 내 존재하는 다양한 화학 반응과 이에 따라 결정되는 생명체의 대사 특성을 파악하기 위해서는 각 효소의 기능을 이해하는 것이 필수적이다. EC 번호(효소 고유 번호, Enzyme Commission number)는 국제생화학 및 분자 생물학연맹 (International Union of Biochemistry and Molecular Biology, IUBMB)가 고안한 효소 기능 분류 체계로서, 다양한 유기체의 대사 특성을 이해하기 위해선 게놈 서열에서 존재하는 효소의 종류와 EC 번호를 빠르게 분석할 수 있는 기술 개발이 필요하다.
단백질의 기능 및 효소 기능 예측을 위해 인공지능을 활용하는 다양한 예측 시스템 또한 보고됐지만, 인공지능의 추론 과정을 직접 확인할 수 없는 블랙박스(black box)의 특징을 가졌거나, 효소 서열 내 아미노산 잔기(최소 단위) 수준으로 해석하지 못하는 문제가 있었다.
공동연구팀은 심층학습 기법과 단백질 상동성 분석 모듈을 활용해 주어진 단백질 서열의 효소 기능을 예측하는 인공지능 딥 EC 트랜스포머(DeepECtransformer)를 개발했다. 연구팀은 이번 연구에서 더 다양한 효소 기능을 정확하게 예측할 수 있도록 단백질 서열 전체 문맥에서 효소 기능에 중요한 정보를 추출하였고, 이를 통해 효소의 EC 번호를 정확하게 예측할 수 있었다. 개발된 인공지능은 총 5,360종류의 EC 번호를 예측할 수 있었다.
공동연구팀은 나아가 딥 EC 트랜스포머의 인공신경망 내 정보 흐름을 분석하여 인공지능이 추론 과정에서 효소 기능에 중요한 활성 부위나 보조 인자 결합 부위 정보를 활용하고 있음을 밝혀냈다. 이처럼 인공지능의 블랙박스를 해석함으로써 인공지능이 학습 과정에서 스스로 효소 기능에 중요한 특징을 파악하고 있음을 연구팀은 확인했다.
이번 논문의 제1 저자인 우리 대학 김기배 박사과정생은 “이번에 개발한 예측 시스템을 활용해 아직 밝혀진 적 없던 효소의 기능을 새롭게 예측하고 실험으로 검증할 수 있었다”고 말했다. 그는 또한 “딥 EC 트랜스포머를 활용해 생명체 내 밝혀지지 않았던 효소를 파악함으로써 유용 화합물을 생합성하기 위해 필요한 효소나 플라스틱을 생분해하기 위해 필요한 효소 등 다양한 대사 과정을 새롭게 밝혀낼 수 있을 것”이라고 덧붙였다.
또한 이상엽 특훈교수는 “효소 기능을 빠르고 정확하게 예측하는 딥 EC 트랜스포머는 기능 유전체학의 핵심 기술로서 시스템 수준에서 전체 효소들의 기능들을 분석할 수 있게 한다”며 “이를 활용해 모든 효소 정보를 포함한 대사 네트워크를 기반으로 친환경 미생물 공장 개발을 수행할 수 있을 것”이라고 밝혔다.
생명화학공학과 김기배 박사과정이 참여한 이번 논문은 국제 학술지 네이처(Nature) 誌가 발행하는 `네이처 커뮤니케이션즈(Nature Communications)'에 동료 심사를 거쳐 11월 14일 字 게재됐다.
※ 논문명 : 트랜스포머 레이어와 딥러닝을 사용하여 효소 인코딩 유전자의 기능적 주석 달기 (Functional annotation of enzyme-encoding genes using deep learning with transformer layers)
※ 저자 정보 : 김기배 (한국과학기술원, 제1 저자), 김지연 (한국과학기술원, 제2 저자), 이종언 (한국과학기술원, 제3 저자), Charles J. Norsigian (UCSD, 제4 저자), Bernhard O. Palsson (UCSD, 제5 저자) 및 이상엽(한국과학기술원, 교신저자) 포함 총 6 명
한편, 이번 연구는 과기정통부가 지원하는 ‘석유대체 친환경 화학기술개발사업의 ‘바이오화학산업 선도를 위한 차세대 바이오리파이너리 원천기술 개발’ 과제(과제책임자 KAIST 이상엽 특훈교수)의 지원을 받아 수행됐다.
2023.11.24 조회수 9122
숨겨진 효소 쏙쏙 찾아내는 인공지능 개발
대장균은 가장 많이 연구된 생명체 중 하나에 해당되지만 아직 대장균을 구성한 단백질 30%의 기능에 대해 명확하게 밝혀지지 않았다. 이에 대해 인공지능을 활용하여 아직 명확하게 밝혀진 바 없던 단백질에서 464종의 효소를 발견하였으며, 이 중 3종의 단백질의 예측된 기능을 시험관 내 효소 분석 방법을 통해 검증하는데 성공하였다.
우리 대학 생명화학공학과 이상엽 특훈교수와 캘리포니아대학교 샌디에이고(UCSD) 생명공학과 버나드 펄슨(Bernhard Palsson) 교수 공동연구팀이 단백질 서열을 활용, 해당 단백질의 효소 기능을 예측할 수 있는 인공지능, `딥 EC 트랜스포머(DeepECtransformer)'를 개발해 빠르고 정확하게 효소 기능을 파악할 수 있는 예측 시스템을 구축했다고 24일 밝혔다.
효소는 생물학적 반응을 촉매하는 단백질로서, 생명체 내 존재하는 다양한 화학 반응과 이에 따라 결정되는 생명체의 대사 특성을 파악하기 위해서는 각 효소의 기능을 이해하는 것이 필수적이다. EC 번호(효소 고유 번호, Enzyme Commission number)는 국제생화학 및 분자 생물학연맹 (International Union of Biochemistry and Molecular Biology, IUBMB)가 고안한 효소 기능 분류 체계로서, 다양한 유기체의 대사 특성을 이해하기 위해선 게놈 서열에서 존재하는 효소의 종류와 EC 번호를 빠르게 분석할 수 있는 기술 개발이 필요하다.
단백질의 기능 및 효소 기능 예측을 위해 인공지능을 활용하는 다양한 예측 시스템 또한 보고됐지만, 인공지능의 추론 과정을 직접 확인할 수 없는 블랙박스(black box)의 특징을 가졌거나, 효소 서열 내 아미노산 잔기(최소 단위) 수준으로 해석하지 못하는 문제가 있었다.
공동연구팀은 심층학습 기법과 단백질 상동성 분석 모듈을 활용해 주어진 단백질 서열의 효소 기능을 예측하는 인공지능 딥 EC 트랜스포머(DeepECtransformer)를 개발했다. 연구팀은 이번 연구에서 더 다양한 효소 기능을 정확하게 예측할 수 있도록 단백질 서열 전체 문맥에서 효소 기능에 중요한 정보를 추출하였고, 이를 통해 효소의 EC 번호를 정확하게 예측할 수 있었다. 개발된 인공지능은 총 5,360종류의 EC 번호를 예측할 수 있었다.
공동연구팀은 나아가 딥 EC 트랜스포머의 인공신경망 내 정보 흐름을 분석하여 인공지능이 추론 과정에서 효소 기능에 중요한 활성 부위나 보조 인자 결합 부위 정보를 활용하고 있음을 밝혀냈다. 이처럼 인공지능의 블랙박스를 해석함으로써 인공지능이 학습 과정에서 스스로 효소 기능에 중요한 특징을 파악하고 있음을 연구팀은 확인했다.
이번 논문의 제1 저자인 우리 대학 김기배 박사과정생은 “이번에 개발한 예측 시스템을 활용해 아직 밝혀진 적 없던 효소의 기능을 새롭게 예측하고 실험으로 검증할 수 있었다”고 말했다. 그는 또한 “딥 EC 트랜스포머를 활용해 생명체 내 밝혀지지 않았던 효소를 파악함으로써 유용 화합물을 생합성하기 위해 필요한 효소나 플라스틱을 생분해하기 위해 필요한 효소 등 다양한 대사 과정을 새롭게 밝혀낼 수 있을 것”이라고 덧붙였다.
또한 이상엽 특훈교수는 “효소 기능을 빠르고 정확하게 예측하는 딥 EC 트랜스포머는 기능 유전체학의 핵심 기술로서 시스템 수준에서 전체 효소들의 기능들을 분석할 수 있게 한다”며 “이를 활용해 모든 효소 정보를 포함한 대사 네트워크를 기반으로 친환경 미생물 공장 개발을 수행할 수 있을 것”이라고 밝혔다.
생명화학공학과 김기배 박사과정이 참여한 이번 논문은 국제 학술지 네이처(Nature) 誌가 발행하는 `네이처 커뮤니케이션즈(Nature Communications)'에 동료 심사를 거쳐 11월 14일 字 게재됐다.
※ 논문명 : 트랜스포머 레이어와 딥러닝을 사용하여 효소 인코딩 유전자의 기능적 주석 달기 (Functional annotation of enzyme-encoding genes using deep learning with transformer layers)
※ 저자 정보 : 김기배 (한국과학기술원, 제1 저자), 김지연 (한국과학기술원, 제2 저자), 이종언 (한국과학기술원, 제3 저자), Charles J. Norsigian (UCSD, 제4 저자), Bernhard O. Palsson (UCSD, 제5 저자) 및 이상엽(한국과학기술원, 교신저자) 포함 총 6 명
한편, 이번 연구는 과기정통부가 지원하는 ‘석유대체 친환경 화학기술개발사업의 ‘바이오화학산업 선도를 위한 차세대 바이오리파이너리 원천기술 개발’ 과제(과제책임자 KAIST 이상엽 특훈교수)의 지원을 받아 수행됐다.
2023.11.24 조회수 9122 -
 유전자 가위와 약물로 동시에 암을 잡는 신약 개발
암은 과거부터 인류의 건강을 위협하는 요인이다. 암을 치료하는 방법으로 임상에서 주로 화학 약물 및 항체 치료가 사용되고 있으나, 심각한 부작용 또는 반복 투여가 필요해 어려움이 있다. 따라서, 영구적인 유전자 조절을 일으키는 유전자가위(CRISPR, 이하 크리스퍼) 기반 유전자 교정 기술을 이용하면 문제를 극복할 수 있지만, 생체 내 전달이 어려워 효과적인 전달 방법이 절실히 필요한 실정이다.
우리 대학 생명과학과 정현정 교수, 화학과 박희성 교수 공동연구팀이 유전자가위 기반 항암 신약으로 크리스퍼 단백질 및 화학 약물을 동시에 생체 내에 전달하는 나노복합체를 개발해 기존 항암제보다 월등한 항암 효능을 보였다고 3일 밝혔다.
크리스퍼 기술은 표적 세포에서 정밀하고 영구적으로 유전자 교정을 일으킬 수 있어 기존 유전자 조절 방법에 비해 암의 치료제로서 큰 장점이 있다. 하지만 크리스퍼의 구성 요소인 단백질과 가이드 RNA를 생체 내에 전달했을 때 극히 낮은 조직 투과성 및 세포 유입 효율로 인해 치료 효능이 떨어진다. 이러한 한계점을 극복하기 위해 지질 나노입자, 고분자, 무기나노입자 등에 나노 전달체에 기반한 전달 방법이 개발됐으나, 여전히 효능이 떨어지고 심각한 독성 및 부작용 문제가 나타났다.
이러한 문제점들을 극복하기 위해 연구팀은 크리스퍼 단백질 Cas9에 서로다른 작용기가 간단히 결합되는 클릭 화학 작용기를 내재하기 위해 비천연 아미노산을 도입한 생직교 반응형 Cas9을 개발했다. 생직교 반응은 살아있는 시스템 내에서 본질적인 생화학 과정을 방해하지 않고 일어나는 반응을 말한다. 연구팀은 기존 나노 전달체의 독성 및 한계를 극복하기 위해 극미량의 고분자 물질을 생직교 반응형 Cas9에 결합시킴으로써 생체 내에 안전하게 전달 및 유전자 교정을 일으킬 수 있음을 확인했다. 또, 항암 신약으로써 효능을 극대화하기 위해 기존에 유방암 항암제로 사용되는 올라파리브(olaparib)을 생직교 반응에 의해 Cas9에 결합시킴으로써 병용 치료를 위한 유전자가위 나노복합체인 콤바인 (ComBiNE, Combinatorial and bioorthogonal nano-editing complex)을 개발했다.
연구팀은 개발한 유전자교정 나노복합체를 이용해 유방암 세포 및 동물모델에서 DNA 복구에 관여하는 유전자 교정 및 올라파리브의 작용으로 기존 항암제 및 단독 치료제에 비해 월등한 항암효과가 나타남을 확인했다.
이번 연구는 최초로 크리스퍼 단백질과 화학 항암제를 단일 제형으로 안전하고 효과적인 생체 내 유전자 교정을 일으켜 높은 항암 효능을 보였다는 점에서 큰 의의가 있다. 연구팀은 이번 연구 결과가 향후 다양한 암종에 대해 유전자 및 화학 약물 기반 병용 치료제로서 적용할 수 있는 강력한 플랫폼 기술로 활용될 것을 기대하고 있다.
우리 대학 생명과학과 마셀 야니스 베하(Marcel Janis Beha) 박사와 석박사통합과정 임산해 학생, 화학과 석박사통합과정 김주찬 학생이 제1 저자로 참여한 이번 연구는 국제학술지 `어드밴스드 사이언스(Advanced Science)'에 7월 23일 온라인 게재됐다. (논문명: Bioorthogonal CRISPR/Cas9-Drug Conjugate: A Combinatorial Nanomedicine Platform)
한편 이번 연구는 한국연구재단, 보건복지부 및 삼성미래기술육성사업의 지원을 통해 이뤄졌다.
2023.08.03 조회수 10708
유전자 가위와 약물로 동시에 암을 잡는 신약 개발
암은 과거부터 인류의 건강을 위협하는 요인이다. 암을 치료하는 방법으로 임상에서 주로 화학 약물 및 항체 치료가 사용되고 있으나, 심각한 부작용 또는 반복 투여가 필요해 어려움이 있다. 따라서, 영구적인 유전자 조절을 일으키는 유전자가위(CRISPR, 이하 크리스퍼) 기반 유전자 교정 기술을 이용하면 문제를 극복할 수 있지만, 생체 내 전달이 어려워 효과적인 전달 방법이 절실히 필요한 실정이다.
우리 대학 생명과학과 정현정 교수, 화학과 박희성 교수 공동연구팀이 유전자가위 기반 항암 신약으로 크리스퍼 단백질 및 화학 약물을 동시에 생체 내에 전달하는 나노복합체를 개발해 기존 항암제보다 월등한 항암 효능을 보였다고 3일 밝혔다.
크리스퍼 기술은 표적 세포에서 정밀하고 영구적으로 유전자 교정을 일으킬 수 있어 기존 유전자 조절 방법에 비해 암의 치료제로서 큰 장점이 있다. 하지만 크리스퍼의 구성 요소인 단백질과 가이드 RNA를 생체 내에 전달했을 때 극히 낮은 조직 투과성 및 세포 유입 효율로 인해 치료 효능이 떨어진다. 이러한 한계점을 극복하기 위해 지질 나노입자, 고분자, 무기나노입자 등에 나노 전달체에 기반한 전달 방법이 개발됐으나, 여전히 효능이 떨어지고 심각한 독성 및 부작용 문제가 나타났다.
이러한 문제점들을 극복하기 위해 연구팀은 크리스퍼 단백질 Cas9에 서로다른 작용기가 간단히 결합되는 클릭 화학 작용기를 내재하기 위해 비천연 아미노산을 도입한 생직교 반응형 Cas9을 개발했다. 생직교 반응은 살아있는 시스템 내에서 본질적인 생화학 과정을 방해하지 않고 일어나는 반응을 말한다. 연구팀은 기존 나노 전달체의 독성 및 한계를 극복하기 위해 극미량의 고분자 물질을 생직교 반응형 Cas9에 결합시킴으로써 생체 내에 안전하게 전달 및 유전자 교정을 일으킬 수 있음을 확인했다. 또, 항암 신약으로써 효능을 극대화하기 위해 기존에 유방암 항암제로 사용되는 올라파리브(olaparib)을 생직교 반응에 의해 Cas9에 결합시킴으로써 병용 치료를 위한 유전자가위 나노복합체인 콤바인 (ComBiNE, Combinatorial and bioorthogonal nano-editing complex)을 개발했다.
연구팀은 개발한 유전자교정 나노복합체를 이용해 유방암 세포 및 동물모델에서 DNA 복구에 관여하는 유전자 교정 및 올라파리브의 작용으로 기존 항암제 및 단독 치료제에 비해 월등한 항암효과가 나타남을 확인했다.
이번 연구는 최초로 크리스퍼 단백질과 화학 항암제를 단일 제형으로 안전하고 효과적인 생체 내 유전자 교정을 일으켜 높은 항암 효능을 보였다는 점에서 큰 의의가 있다. 연구팀은 이번 연구 결과가 향후 다양한 암종에 대해 유전자 및 화학 약물 기반 병용 치료제로서 적용할 수 있는 강력한 플랫폼 기술로 활용될 것을 기대하고 있다.
우리 대학 생명과학과 마셀 야니스 베하(Marcel Janis Beha) 박사와 석박사통합과정 임산해 학생, 화학과 석박사통합과정 김주찬 학생이 제1 저자로 참여한 이번 연구는 국제학술지 `어드밴스드 사이언스(Advanced Science)'에 7월 23일 온라인 게재됐다. (논문명: Bioorthogonal CRISPR/Cas9-Drug Conjugate: A Combinatorial Nanomedicine Platform)
한편 이번 연구는 한국연구재단, 보건복지부 및 삼성미래기술육성사업의 지원을 통해 이뤄졌다.
2023.08.03 조회수 10708