%EC%9C%A0%EC%A0%84%EC%9E%90
-
 인공지능 기반 대장암 3차원 게놈 지도 최초 해독
세계 최초로 예전에 비해 최대 규모로 한국인 대장암 환자 3차원 게놈 지도를 작성하여 화제다.
우리 대학 생명과학과 정인경 교수 연구팀이 서울대학교 암연구소 김태유 교수 연구팀과의 공동연구를 통해 인공지능 기반 알고리즘을 활용, 한국인 대장암 환자의 3차원 게놈 지도를 최초로 제시했으며 이를 토대로 암 세포 특이적인 유전자 조절 기전을 통해 특정 종양유전자들이 과발현되는 현상을 규명했다고 24일 밝혔다.
1차원적 게놈 서열 분석에 기반한 현재의 암 유전체 연구는 종양유전자들의 과발현 기작을 설명하는데 한계가 있었다. 하지만 3차원 공간상에 게놈이 어떻게 배열되는지를 분석하는 3차원 게놈 (3D genome) 구조 연구는 이러한 한계를 극복 가능케 하고 있다. 본 연구에서는 정상 세포에서는 존재하지 않는 암 세포 특이적 염색질 고리(chromatin loop) 구조가 유전자 발현 촉진 인자인 인핸서와 종양유전자 사이의 상호작용을 형성하여 과발현을 유도하는 인핸서 납치(enhancer-hijacking) 현상에 초점을 두어 연구하였다.
우리 대학 생명과학과 김규광 박사과정이 주도한 이번 연구는 게놈간의 공간상 상호작용을 측정할 수 있는 대용량 염색체 구조 포착 Hi-C (High-throughput Chromosome Conformation Capture) 실험 기법을 활용하여 대장암 3차원 게놈 지도를 작성하고 대장암 특이적 3차원 게놈 변화를 환자 개개인별로 분석할 수 있는 인공지능 기반 알고리즘을 개발했다. 그 결과 공동연구팀은 광범위한 규모의 3차원 게놈 구조 변화와 이로 인한 다양한 종양유전자의 활성화를 확인했다.
연구팀은 이번 연구를 통해 암 특이적 3차원 게놈 구조의 변화로 인한 종양유전자 활성 기작을 명확히 제시하였으며 이로 인한 환자 예후와 약물 반응 등 임상적인 특성과의 연관성까지 제시해 맞춤 치료 원천기술 확보에 기여했다.
지금까지 암 세포주에 대한 3차원 게놈 구조 연구는 일부 보고 되었으나, 대규모 환자 암조직에 대한 연구는 조직 내 세포 이질성, 종양 순도, 암세포 이질성 등의 문제로 인한 정밀 암 특이적 3차원 게놈 구조 분석의 한계로 수행되지 못하였다.
반면에 이번 연구에서 연구팀은 AI 기반 알고리즘으로 환자 개인 종양 조직으로부터 얻어진 복잡한 신호를 해석할 수 있었으며 그 결과 최대 규모인 환자 40명의 종양 조직과 인접한 정상 대장 조직을 사용해 3차원 게놈 지도를 작성할 수 있었다. 또한 DNA 서열정보를 보여주는 전장유전체 지도의 경우 다양한 인종에 대해 생산되고 있고 한국인의 전장유전체 지도 또한 개발되었으나 한국인 3차원 게놈 지도, 특히 종양 조직에 대한 3차원 게놈 지도는 이번 연구에서 최초로 제시됐다.
이번 연구 결과는 국제 학술지, `셀 리포츠(Cell Reports, IF=9.995)'에 7월 13일 자로 출판됐다. (논문명: Spatial and clonality-resolved 3D cancer genome alterations reveal enhancer-hijacking as a potential prognostic marker for colorectal cancer)
서울대학교병원 혈액종양내과 김태유 교수는 “이러한 결과는 개별 암 환자들마다 서로 다르게 나타나는 종양 이질성을 이해하는 데 매우 중요한 요소가 될 수 있으며, 이를 이용한 환자 맞춤형 치료 연구의 시발점이 될 것이다”라고 말했다. 생명과학과 정인경 교수는 “기존의 점돌연변이나 유전체 변이만으로는 설명이 어려운 암 유전체를 3차원 게놈 구조 관점에서 재해독하고 신규 암 타겟을 발굴할 수 있는 수 있는 새로운 접근법을 제시했다”라고 밝혔다.
한편 이번 연구는 과학기술정보통신부와 서경배과학재단의 지원을 받아 수행됐다.
2023.07.24 조회수 8751
인공지능 기반 대장암 3차원 게놈 지도 최초 해독
세계 최초로 예전에 비해 최대 규모로 한국인 대장암 환자 3차원 게놈 지도를 작성하여 화제다.
우리 대학 생명과학과 정인경 교수 연구팀이 서울대학교 암연구소 김태유 교수 연구팀과의 공동연구를 통해 인공지능 기반 알고리즘을 활용, 한국인 대장암 환자의 3차원 게놈 지도를 최초로 제시했으며 이를 토대로 암 세포 특이적인 유전자 조절 기전을 통해 특정 종양유전자들이 과발현되는 현상을 규명했다고 24일 밝혔다.
1차원적 게놈 서열 분석에 기반한 현재의 암 유전체 연구는 종양유전자들의 과발현 기작을 설명하는데 한계가 있었다. 하지만 3차원 공간상에 게놈이 어떻게 배열되는지를 분석하는 3차원 게놈 (3D genome) 구조 연구는 이러한 한계를 극복 가능케 하고 있다. 본 연구에서는 정상 세포에서는 존재하지 않는 암 세포 특이적 염색질 고리(chromatin loop) 구조가 유전자 발현 촉진 인자인 인핸서와 종양유전자 사이의 상호작용을 형성하여 과발현을 유도하는 인핸서 납치(enhancer-hijacking) 현상에 초점을 두어 연구하였다.
우리 대학 생명과학과 김규광 박사과정이 주도한 이번 연구는 게놈간의 공간상 상호작용을 측정할 수 있는 대용량 염색체 구조 포착 Hi-C (High-throughput Chromosome Conformation Capture) 실험 기법을 활용하여 대장암 3차원 게놈 지도를 작성하고 대장암 특이적 3차원 게놈 변화를 환자 개개인별로 분석할 수 있는 인공지능 기반 알고리즘을 개발했다. 그 결과 공동연구팀은 광범위한 규모의 3차원 게놈 구조 변화와 이로 인한 다양한 종양유전자의 활성화를 확인했다.
연구팀은 이번 연구를 통해 암 특이적 3차원 게놈 구조의 변화로 인한 종양유전자 활성 기작을 명확히 제시하였으며 이로 인한 환자 예후와 약물 반응 등 임상적인 특성과의 연관성까지 제시해 맞춤 치료 원천기술 확보에 기여했다.
지금까지 암 세포주에 대한 3차원 게놈 구조 연구는 일부 보고 되었으나, 대규모 환자 암조직에 대한 연구는 조직 내 세포 이질성, 종양 순도, 암세포 이질성 등의 문제로 인한 정밀 암 특이적 3차원 게놈 구조 분석의 한계로 수행되지 못하였다.
반면에 이번 연구에서 연구팀은 AI 기반 알고리즘으로 환자 개인 종양 조직으로부터 얻어진 복잡한 신호를 해석할 수 있었으며 그 결과 최대 규모인 환자 40명의 종양 조직과 인접한 정상 대장 조직을 사용해 3차원 게놈 지도를 작성할 수 있었다. 또한 DNA 서열정보를 보여주는 전장유전체 지도의 경우 다양한 인종에 대해 생산되고 있고 한국인의 전장유전체 지도 또한 개발되었으나 한국인 3차원 게놈 지도, 특히 종양 조직에 대한 3차원 게놈 지도는 이번 연구에서 최초로 제시됐다.
이번 연구 결과는 국제 학술지, `셀 리포츠(Cell Reports, IF=9.995)'에 7월 13일 자로 출판됐다. (논문명: Spatial and clonality-resolved 3D cancer genome alterations reveal enhancer-hijacking as a potential prognostic marker for colorectal cancer)
서울대학교병원 혈액종양내과 김태유 교수는 “이러한 결과는 개별 암 환자들마다 서로 다르게 나타나는 종양 이질성을 이해하는 데 매우 중요한 요소가 될 수 있으며, 이를 이용한 환자 맞춤형 치료 연구의 시발점이 될 것이다”라고 말했다. 생명과학과 정인경 교수는 “기존의 점돌연변이나 유전체 변이만으로는 설명이 어려운 암 유전체를 3차원 게놈 구조 관점에서 재해독하고 신규 암 타겟을 발굴할 수 있는 수 있는 새로운 접근법을 제시했다”라고 밝혔다.
한편 이번 연구는 과학기술정보통신부와 서경배과학재단의 지원을 받아 수행됐다.
2023.07.24 조회수 8751 -
 정크 DNA가 노화와 발암에 관여한다
인간 유전체 중 일반적인 단백질 생성 유전자는 전체 염기서열의 1% 정도에 불과하며 나머지 99%의 유전체 영역은 그 기능이 뚜렷하게 알려지지 않아 ‘쓸모없는 DNA’라는 뜻으로 ‘정크 DNA’라고 불리고 있다. 정크 DNA 가운데 약 1/6을 차지하는 L1 점핑 유전자는 활성화될 경우 세포의 유전정보를 파괴하거나 교란하는 역할을 할 수 있어 사람의 진화 과정에서 불활성화(화석화) 됐다고 알려져 있었다. 하지만 이번 연구에서 L1 점핑 유전자가 활성화되며, 노화와 발암 과정에 연관이 있음을 처음 확인하였다.
우리 대학 의과학대학원 주영석 교수 연구팀이 서울대학교병원 외과 김민정 교수, 고려대학교 의과대학 권현우 교수팀과의 공동연구로 ‘L1 점핑 유전자’의 활성화에 의한 사람 대장 상피 세포의 유전체 파괴 현상을 규명했다고 15일 밝혔다.
의과학대학원 남창현 박사과정과 육정환 박사(現 서울대병원 내과 임상조교수)가 공동 제1 저자로 참여한 이번 연구는 국제 학술지 `네이처(Nature)' 5월 10일 字 온라인판(영국 현지시간)에 게재됐다 (논문명 : Widespread somatic L1 retrotransposition in normal colorectal epithelium). 이번 연구에는 한국과학기술정보연구원 (KISTI), 서울대학교병원 내과, 연세대학교 의과대학, 서울시립대학교, 및 KAIST 교원창업기업 지놈인사이트(Genome Insight)의 연구자들도 참여했다.
우리 몸에서 L1 점핑 유전자의 활성화는 유전체 서열의 ‘파괴적 혁신’을 일으킬 수 있기 때문에 인간 종의 진화 과정을 촉진하였다고 알려져 있으나, 사람 개개인의 입장에서는 L1 점핑 유전자의 활성화가 세포 유전체의 파괴 및 암 등 질병 발생을 촉진하여 생존에 불리하기 때문에 현생 인류에서 대다수의 L1 점핑 유전자는 불활성화(화석화)된 것으로 여겨졌다.
이번 연구는 이러한 일반적인 믿음과는 달리 L1 점핑 유전자의 일부는 아직도 특정 조직에서 활성화될 수 있고, 노화 과정에서 이들이 유전체 돌연변이를 빈번하게 생성하고 있음을 명확하게 규명하여, 세포의 노화 및 암 발생 과정을 이해하는 새로운 관점을 제시한 것으로 평가된다.
연구팀은 28명의 개인의 피부(섬유아세포), 혈액 및 대장 상피 조직에서 확보한 총 899개 단일세포의 전장 유전체(whole-genome sequencing) 서열을 생명정보학 기법으로 분석했다. L1 점핑 유전자에 의한 돌연변이의 빈도는 세포 종류에 따라 큰 차이를 보였으며 노화된 대장 상피세포에서 주로 발견됐다. 연구팀은 L1 점핑 유전자의 활성화에 의한 대장 상피세포의 유전체 돌연변이가 태어나기 전 배아 발생단계에서부터 평생에 걸쳐서 지속적으로 일어나고 있음을 확인했다. 연구에 따르면 40세가 된 개인의 대장 상피 세포들은 평균적으로 1개 이상의 L1 점핑 유전자에 의한 돌연변이를 갖게 된다.
연구팀은 L1 점핑 유전자 활성화 기전을 추적하기 위해 DNA 뿐만 아니라 후성 유전체 (DNA 메틸레이션) 서열을 함께 확인하였다. L1 점핑 유전자가 활성화된 세포에서는 후성 유전체의 불안정성이 발견되어 후성 유전체의 변화가 L1 점핑 유전자의 활성을 조절하는 스위치임을 확인하였다. 연구팀은 세포들의 배아발생과정을 추적하여, 이러한 후성 유전체 불안정성의 대다수가 초기 배아 발생과정에 형성되었음을 제시하였다.
이번 연구는 향후 더 많은 조직에서 L1 점핑유전자 활성화에 의한 노화 및 발암 과정을 확인하고 이의 활성화를 억제하여 인체 노화 및 질환 발생을 제어하는 기술개발에 이바지할 수 있을 것으로 연구팀은 기대했다.
의과학대학원 주영석 교수는 "전장유전체 및 생명정보학의 광범위한 적용을 통해 그동안 규명하기 어려웠던 L1 점핑 유전자에 의한 생명현상을 확인한 대표적인 연구ˮ라며 “이번 연구는 DNA 돌연변이가 암이나 질환을 갖고 있는 세포의 전유물이 아니며, 인간의 정상 세포의 노화과정에서 세포 자체의 불안정성에 의해 끊임없이 돌연변이가 생성되고 있음을 보여준다”라고 말했다.
서울대학교병원 외과 김민정 교수는 "임상현장에서 체계적으로 확보한 사람 유래 조직이 실제 인간에서 일어나는 질병 과정을 발견하는 데 큰 역할을 할 수 있음을 보여주는 사례ˮ라며 "향후 임상 및 기초의학의 밀접한 공동연구가 필요하다ˮ라고 말했다.
고려대학교 의과대학 핵의학과 권현우 교수는 “그동안 연구팀에서 고도화한 단일세포 유전체 기술이 큰 결실을 맺게 되어 기쁘다”라며 “앞으로 지속적으로 단일세포 유전체 기술을 선도할 수 있도록 매진할 것”이라고 말했다.
한편 이번 연구는 한국연구재단 리더연구, 한국연구재단 생애첫연구, 한국보건산업진흥원 융합형 의사과학자 양성 지원 사업, 서경배과학재단 신진과학자 연구지원 프로그램의 지원을 받아 수행됐다.
2023.05.15 조회수 9352
정크 DNA가 노화와 발암에 관여한다
인간 유전체 중 일반적인 단백질 생성 유전자는 전체 염기서열의 1% 정도에 불과하며 나머지 99%의 유전체 영역은 그 기능이 뚜렷하게 알려지지 않아 ‘쓸모없는 DNA’라는 뜻으로 ‘정크 DNA’라고 불리고 있다. 정크 DNA 가운데 약 1/6을 차지하는 L1 점핑 유전자는 활성화될 경우 세포의 유전정보를 파괴하거나 교란하는 역할을 할 수 있어 사람의 진화 과정에서 불활성화(화석화) 됐다고 알려져 있었다. 하지만 이번 연구에서 L1 점핑 유전자가 활성화되며, 노화와 발암 과정에 연관이 있음을 처음 확인하였다.
우리 대학 의과학대학원 주영석 교수 연구팀이 서울대학교병원 외과 김민정 교수, 고려대학교 의과대학 권현우 교수팀과의 공동연구로 ‘L1 점핑 유전자’의 활성화에 의한 사람 대장 상피 세포의 유전체 파괴 현상을 규명했다고 15일 밝혔다.
의과학대학원 남창현 박사과정과 육정환 박사(現 서울대병원 내과 임상조교수)가 공동 제1 저자로 참여한 이번 연구는 국제 학술지 `네이처(Nature)' 5월 10일 字 온라인판(영국 현지시간)에 게재됐다 (논문명 : Widespread somatic L1 retrotransposition in normal colorectal epithelium). 이번 연구에는 한국과학기술정보연구원 (KISTI), 서울대학교병원 내과, 연세대학교 의과대학, 서울시립대학교, 및 KAIST 교원창업기업 지놈인사이트(Genome Insight)의 연구자들도 참여했다.
우리 몸에서 L1 점핑 유전자의 활성화는 유전체 서열의 ‘파괴적 혁신’을 일으킬 수 있기 때문에 인간 종의 진화 과정을 촉진하였다고 알려져 있으나, 사람 개개인의 입장에서는 L1 점핑 유전자의 활성화가 세포 유전체의 파괴 및 암 등 질병 발생을 촉진하여 생존에 불리하기 때문에 현생 인류에서 대다수의 L1 점핑 유전자는 불활성화(화석화)된 것으로 여겨졌다.
이번 연구는 이러한 일반적인 믿음과는 달리 L1 점핑 유전자의 일부는 아직도 특정 조직에서 활성화될 수 있고, 노화 과정에서 이들이 유전체 돌연변이를 빈번하게 생성하고 있음을 명확하게 규명하여, 세포의 노화 및 암 발생 과정을 이해하는 새로운 관점을 제시한 것으로 평가된다.
연구팀은 28명의 개인의 피부(섬유아세포), 혈액 및 대장 상피 조직에서 확보한 총 899개 단일세포의 전장 유전체(whole-genome sequencing) 서열을 생명정보학 기법으로 분석했다. L1 점핑 유전자에 의한 돌연변이의 빈도는 세포 종류에 따라 큰 차이를 보였으며 노화된 대장 상피세포에서 주로 발견됐다. 연구팀은 L1 점핑 유전자의 활성화에 의한 대장 상피세포의 유전체 돌연변이가 태어나기 전 배아 발생단계에서부터 평생에 걸쳐서 지속적으로 일어나고 있음을 확인했다. 연구에 따르면 40세가 된 개인의 대장 상피 세포들은 평균적으로 1개 이상의 L1 점핑 유전자에 의한 돌연변이를 갖게 된다.
연구팀은 L1 점핑 유전자 활성화 기전을 추적하기 위해 DNA 뿐만 아니라 후성 유전체 (DNA 메틸레이션) 서열을 함께 확인하였다. L1 점핑 유전자가 활성화된 세포에서는 후성 유전체의 불안정성이 발견되어 후성 유전체의 변화가 L1 점핑 유전자의 활성을 조절하는 스위치임을 확인하였다. 연구팀은 세포들의 배아발생과정을 추적하여, 이러한 후성 유전체 불안정성의 대다수가 초기 배아 발생과정에 형성되었음을 제시하였다.
이번 연구는 향후 더 많은 조직에서 L1 점핑유전자 활성화에 의한 노화 및 발암 과정을 확인하고 이의 활성화를 억제하여 인체 노화 및 질환 발생을 제어하는 기술개발에 이바지할 수 있을 것으로 연구팀은 기대했다.
의과학대학원 주영석 교수는 "전장유전체 및 생명정보학의 광범위한 적용을 통해 그동안 규명하기 어려웠던 L1 점핑 유전자에 의한 생명현상을 확인한 대표적인 연구ˮ라며 “이번 연구는 DNA 돌연변이가 암이나 질환을 갖고 있는 세포의 전유물이 아니며, 인간의 정상 세포의 노화과정에서 세포 자체의 불안정성에 의해 끊임없이 돌연변이가 생성되고 있음을 보여준다”라고 말했다.
서울대학교병원 외과 김민정 교수는 "임상현장에서 체계적으로 확보한 사람 유래 조직이 실제 인간에서 일어나는 질병 과정을 발견하는 데 큰 역할을 할 수 있음을 보여주는 사례ˮ라며 "향후 임상 및 기초의학의 밀접한 공동연구가 필요하다ˮ라고 말했다.
고려대학교 의과대학 핵의학과 권현우 교수는 “그동안 연구팀에서 고도화한 단일세포 유전체 기술이 큰 결실을 맺게 되어 기쁘다”라며 “앞으로 지속적으로 단일세포 유전체 기술을 선도할 수 있도록 매진할 것”이라고 말했다.
한편 이번 연구는 한국연구재단 리더연구, 한국연구재단 생애첫연구, 한국보건산업진흥원 융합형 의사과학자 양성 지원 사업, 서경배과학재단 신진과학자 연구지원 프로그램의 지원을 받아 수행됐다.
2023.05.15 조회수 9352 -
 RNA 유전자 가위 기술로 코로나바이러스 싹둑
엔데믹(endemic)으로 지정된 코로나 19 바이러스(이하 SARS-CoV-2)는 변이가 매우 빈번하고 빠른 RNA 바이러스이다. 따라서 전 세계 연구자들은 신·변종 바이러스 출현에 따른 새로운 팬데믹에 대비하기 위해 범용 코로나 바이러스 감염병 치료제 개발에 몰두하고 있다.
우리 대학 생명과학과 허원도 교수 연구팀과 전북대 강상민 교수 연구팀이 공동연구를 통해 세계 최초로 RNA 유전자 가위 기술을 이용해 RNA 바이러스 유전체 내 슈도낫 부위를 타겟해 바이러스 증식을 강력하게 차단할 수 있는 핵심 타겟부위를 발견했고, 전북대학교 인수공통감염병 연구소와의 협업을 통해 동물모델에서 COVID-19 치료 효과를 입증했다고 1일 밝혔다.
우리 대학 자연과학연구소 유다슬이 연구조교수, 전북대학교 한희정 박사과정, KAIST 생명과학과 유정혜 박사과정, KAIST 생명과학과 김지혜 선임연구원이 공동 제 1저자로 수행한 이번 연구는 저명 국제 학술지 ‘몰레큘러 테라피 (Molecular Therapy)’ 2023년 3월호에 온라인으로 출판됐다. (논문명: Pseudoknot-targeting Cas13b combats SARS-CoV-2 infection by suppressing viral replication). (Impact Factor: 12.91). (DOI: https://doi.org/10.1016/j.ymthe.2023.03.018)
SARS-CoV-2 바이러스는 세포 내 감염 후 매우 빠른 속도로 바이러스 단백질을 복제하고 증식하여 숙주 세포의 기능을 완전히 망가뜨린다. RNA 바이러스 유전체를 직접 분해해 바이러스 증식을 억제하는 시도는 이전부터 있어왔으나 이런 빠른 코로나 증식을 완전히 막기에는 역부족이었다.
연구팀은 RNA 유전자 가위 기술로 코로나 바이러스 내 유전자 발현 조절 중추 역할을 하는 슈도낫 부위를 타겟함으로써 99.9퍼센트의 바이러스 증식 억제 효과가 있음을 입증했다. 이는 슈도낫 부위가 코로나 바이러스의 가장 취약한 급소임을 보여주는 결과이기도 하다.
본 기술은 mRNA 기반 유전자 치료제 전달하는 방식으로 DNA 기반 유전자 치료제에 비해 전달 효율이 매우 높으며 치료제 발현 시간 또한 매우 빠르다. 연구진이 제작한 mRNA 기반 치료제 전달로 감염 세포에 2시간 이내, 감염 동물에 6시간 이내에 RNA 유전자 가위 기술 발현을 유도할 수 있었다.
연구진이 타겟한 슈도낫 부위는 MERS, SARS-CoV 유전체 내에서도 보존성이 높은 염기서열을 가졌으며, SARS-CoV-2 변이체 (알파, 베타, 감마, 델타, 오미크론) 모두에서 동일한 염기서열을 가졌다. 연구진은 전북대학교 인수공통연구소와의 공동연구를 통해 SARS-CoV-2 (Hu-1) 뿐만 아니라 변이체 증식 또한 매우 효과적으로 억제됨을 보여주며 해당 기술의 범용성을 증명하였다. 또한 SARS-CoV-2 감염 쥐 모델에 해당 치료제 기술이 투여된 쥐에서 뚜렷한 COVID-19 치료 효과를 입증했다.
유다슬이 연구조교수는 “이번 연구 결과는 바이러스 유전체 중 단백질을 구성하는 유전자가 아닌 단백질 발현을 조절하는 유전자를 세계 최초로 타겟 했다는 점과, 그것이 다른 유전자 타겟 부위보다 바이러스 증식 억제 효율이 뛰어났다는 점에서 중요한 의미를 갖는다”고 말했다.
허원도 교수는 “우리 RNA유전자가위 연구는 본래 바이러스 감염병 치료 목적으로 시작하지는 않았지만 팬데믹이라는 세계적 재난 상황에서 기여하고자 연구를 시작했고, 전북대 인수공통감염병연구소와 공동연구를 통해 치료 효과를 입증할 수 있었다. 또한 mRNA 백신으로 인류가 빠르게 팬데믹을 극복했듯이 mRNA 치료제 개발로 미래에 출현할 바이러스 감염병에 신속한 대응을 하도록 본 기술을 발전시키겠다”며 앞으로의 계획을 밝혔다.
한편, 이번 연구는 KAIST 코로나대응 과학기술뉴딜사업과 보건복지부 감염병 예방 치료기술개발 사업의 지원을 받아 수행됐다.
2023.05.02 조회수 14105
RNA 유전자 가위 기술로 코로나바이러스 싹둑
엔데믹(endemic)으로 지정된 코로나 19 바이러스(이하 SARS-CoV-2)는 변이가 매우 빈번하고 빠른 RNA 바이러스이다. 따라서 전 세계 연구자들은 신·변종 바이러스 출현에 따른 새로운 팬데믹에 대비하기 위해 범용 코로나 바이러스 감염병 치료제 개발에 몰두하고 있다.
우리 대학 생명과학과 허원도 교수 연구팀과 전북대 강상민 교수 연구팀이 공동연구를 통해 세계 최초로 RNA 유전자 가위 기술을 이용해 RNA 바이러스 유전체 내 슈도낫 부위를 타겟해 바이러스 증식을 강력하게 차단할 수 있는 핵심 타겟부위를 발견했고, 전북대학교 인수공통감염병 연구소와의 협업을 통해 동물모델에서 COVID-19 치료 효과를 입증했다고 1일 밝혔다.
우리 대학 자연과학연구소 유다슬이 연구조교수, 전북대학교 한희정 박사과정, KAIST 생명과학과 유정혜 박사과정, KAIST 생명과학과 김지혜 선임연구원이 공동 제 1저자로 수행한 이번 연구는 저명 국제 학술지 ‘몰레큘러 테라피 (Molecular Therapy)’ 2023년 3월호에 온라인으로 출판됐다. (논문명: Pseudoknot-targeting Cas13b combats SARS-CoV-2 infection by suppressing viral replication). (Impact Factor: 12.91). (DOI: https://doi.org/10.1016/j.ymthe.2023.03.018)
SARS-CoV-2 바이러스는 세포 내 감염 후 매우 빠른 속도로 바이러스 단백질을 복제하고 증식하여 숙주 세포의 기능을 완전히 망가뜨린다. RNA 바이러스 유전체를 직접 분해해 바이러스 증식을 억제하는 시도는 이전부터 있어왔으나 이런 빠른 코로나 증식을 완전히 막기에는 역부족이었다.
연구팀은 RNA 유전자 가위 기술로 코로나 바이러스 내 유전자 발현 조절 중추 역할을 하는 슈도낫 부위를 타겟함으로써 99.9퍼센트의 바이러스 증식 억제 효과가 있음을 입증했다. 이는 슈도낫 부위가 코로나 바이러스의 가장 취약한 급소임을 보여주는 결과이기도 하다.
본 기술은 mRNA 기반 유전자 치료제 전달하는 방식으로 DNA 기반 유전자 치료제에 비해 전달 효율이 매우 높으며 치료제 발현 시간 또한 매우 빠르다. 연구진이 제작한 mRNA 기반 치료제 전달로 감염 세포에 2시간 이내, 감염 동물에 6시간 이내에 RNA 유전자 가위 기술 발현을 유도할 수 있었다.
연구진이 타겟한 슈도낫 부위는 MERS, SARS-CoV 유전체 내에서도 보존성이 높은 염기서열을 가졌으며, SARS-CoV-2 변이체 (알파, 베타, 감마, 델타, 오미크론) 모두에서 동일한 염기서열을 가졌다. 연구진은 전북대학교 인수공통연구소와의 공동연구를 통해 SARS-CoV-2 (Hu-1) 뿐만 아니라 변이체 증식 또한 매우 효과적으로 억제됨을 보여주며 해당 기술의 범용성을 증명하였다. 또한 SARS-CoV-2 감염 쥐 모델에 해당 치료제 기술이 투여된 쥐에서 뚜렷한 COVID-19 치료 효과를 입증했다.
유다슬이 연구조교수는 “이번 연구 결과는 바이러스 유전체 중 단백질을 구성하는 유전자가 아닌 단백질 발현을 조절하는 유전자를 세계 최초로 타겟 했다는 점과, 그것이 다른 유전자 타겟 부위보다 바이러스 증식 억제 효율이 뛰어났다는 점에서 중요한 의미를 갖는다”고 말했다.
허원도 교수는 “우리 RNA유전자가위 연구는 본래 바이러스 감염병 치료 목적으로 시작하지는 않았지만 팬데믹이라는 세계적 재난 상황에서 기여하고자 연구를 시작했고, 전북대 인수공통감염병연구소와 공동연구를 통해 치료 효과를 입증할 수 있었다. 또한 mRNA 백신으로 인류가 빠르게 팬데믹을 극복했듯이 mRNA 치료제 개발로 미래에 출현할 바이러스 감염병에 신속한 대응을 하도록 본 기술을 발전시키겠다”며 앞으로의 계획을 밝혔다.
한편, 이번 연구는 KAIST 코로나대응 과학기술뉴딜사업과 보건복지부 감염병 예방 치료기술개발 사업의 지원을 받아 수행됐다.
2023.05.02 조회수 14105 -
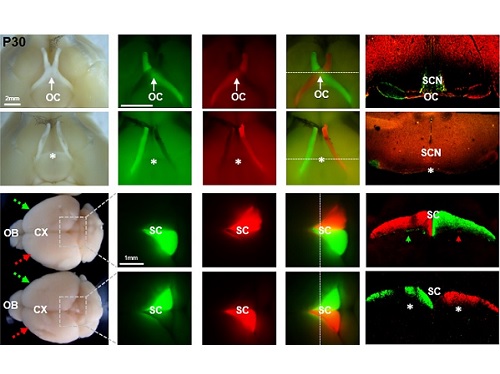 왼쪽 눈이 본 것을 오른쪽 뇌가 알게 하라
인간을 비롯한 대부분 동물의 신체 기관들은 대칭적 구조를 가지고 있다. 이를 통해 몸의 좌우 균형을 맞추고, 움직이고, 반응을 할 수 있게 된다. 동물의 시각이 시작되는 안구 역시 머리의 양쪽에 하나씩 위치하며 한쪽 눈으로 볼 때 보다 더 넓은 영역의 물체를 인식할 수 있게 된다. 사람이나 고양이 같은 경우는 양쪽 눈이 정면을 향하고 있고, 개나 쥐 같은 동물은 눈이 사람보다 측면부를 향해 있고, 많은 물고기의 경우는 두 눈이 완전히 반대쪽을 향하고 있다. 이로 인해 좌우측 눈이 받아들이는 이미지 역시 차이를 보이게 된다. 인간의 경우 좌측과 우측 눈이 인식하는 이미지의 50%에 가까운 영역이 겹치는 반면, 생쥐의 경우는 5% 이하의 영역이 중복이 되며, 물고기는 중복된 영역이 거의 없다. 이들 겹치는 시각 영역 이미지의 위상차를 뇌가 인식해 동물은 물체의 입체감을 느낄 수 있다. 또, 물체가 움직이는 경우에는 좌측 눈과 우측 눈에 감지된 물체의 이미지의 시간차 정보가 뇌에서 처리되어 물체의 이동 경로를 감지하고 예측할 수 있게 된다. 결국, 중복된 시각 영역이 넓을수록 외부 물체의 입체감과 이동을 더 잘 감지할 수 있게 되어, 대부분 포식 동물들이 넓은 중복 영역을 확보하기 위해 안구를 정면에 위치하는 경우가 많다.
이렇게 좌우 안구에서 인식된 이미지를 뇌의 특정 영역에 전달하기 위해 눈에서 나온 시신경은 뇌의 좌우 반구에 모두 연결이 되어 있다. 흥미롭게 좌우 반구로 연결되는 시신경 비율은 좌우 안구 이미지의 중복 비율에 역비례해서, 인간의 경우 50% 시신경이 반대쪽 뇌로 연결되고, 생쥐의 경우 95% 내외, 물고기는 100% 반대쪽으로 연결된다. 시신경이 좌측 또는 우측 중 어느 쪽 뇌로 뻗어 나갈 것인지를 결정하는 과정은 시신경이 눈에서 출발해 시상하부 영역에 도달할 때 시상하부 중간선에 존재하는 경로 결정자(pathway selection cue)에 의해 일어난다고 알려져 있다. 오랜 동안 이러한 동물의 양안 시각계 (binocular visual system)의 핵심인 시상하부 중간선에서 경로 결정에 관련된 메커니즘을 이해하려는 시도가 있어 왔고 일부 경로 결정 인자들이 밝혀진 바도 있다. 하지만, 핵심인자의 부재로 이 과정에 대한 명확한 이해는 부족한 상황이었다.
생명과학과 김진우 교수 연구실에서는 시신경 및 시상하부 중간선에 많이 발현되는 VAX1 유전자에 대한 연구를 수행해 오고 있다. 이 유전자가 결핍된 생쥐와 사람은 시신경이 제대로 성장하지 못하고 시신경이 시상하부에서 교차하지 못하는 발달 이상을 보였다. VAX1이 호메오도메인을 가지는 전사인자의 특성을 가지고 있기 때문에 당연히 시상하부에서 경로 결정자의 유전자 발현을 조절해 시신경 교차(optic chiasm)를 생성할 것이라고 추정하였으나, 김교수 연구팀에서는 VAX1이 시상하부 세포에서 전사인자로 기능하기 보다는 눈의 망막신경절세포에서 뻗어 나온 시신경 축삭(axon)에서 mRNA 번역인자로 작용하여 시신경의 성장을 유도한다는 놀라운 사실을 발견하여 2014년 발표한 바 있다. 하지만, VAX1이 전혀 없는 동물은 두개골 기형 때문에 생존하기 어려워 이러한 VAX1 이상으로 인해 시신경 교차가 없는 동물의 시각 반응 및 행동에 대한 이해는 이루어지지 못하고 있었다.
김 교수 연구실에서 VAX1의 전사인자 기능은 유지한 채 시신경 축삭에 작용하지 못하는 VAX1(AA) 생쥐를 제작하였고, 이 생쥐는 외형적 이상이 전혀 없이 정상적으로 태어나 성장하였다. 다만, VAX1(AA) 생쥐는 모든 시신경이 안구와 같은 쪽 뇌에만 연결되는 시신경 교차 결핍증(agenesis of optic chiasm, AOC)을 나타냈다. 이 생쥐의 시각을 다양한 방법을 통해 검증한 결과, 눈 속의 신경 조직인 망막이 빛을 감지하는 기능은 정상이나 입체 시각이 전혀 없었고, 시력 역시 저하되어 있었다.
흥미로운 점은 VAX1(AA) 생쥐의 눈이 아무런 자극이 없는 상태에서도 지속적으로 상하궤도 운동을 하는 시소안구진탕증(Seesaw Nystagmus)를 보인다는 것이었다. 이러한 시소안구진탕증은 시신경 교차에 이상이 있는 사람과 벨지안쉽도그(Belgian sheepdog)에서도 관찰이 된 바 있어서 시신경 교차 결여가 VAX1(AA) 생쥐의 안구 운동 이상의 원인임을 알 수 있었다.
더욱 흥미로운 점은 VAX1(AA) 생쥐의 시각 운동 반응이 반전되어 있다는 점이었다. 왼쪽 눈에 빛을 주면 오른쪽 동공이 먼저 축소되고, 물체 이동을 감지한 후에는 움직이던 눈이 오히려 정면을 응시하는 등, 시각 정보와 반대되는 안구의 움직임을 보였다. VAX1(AA) 생쥐는 시신경 교차에만 이상이 있고 시각을 처리하는 뇌 부위는 정상적으로 형성이 되어 있기 때문에, 이 결과는 우측 눈에서 오는 신호를 처리해 우측 눈으로 운동 정보를 보내야 할 좌측뇌가 정작 좌측 눈에서 오는 신호를 받아 우측 눈을 자극하는 입력-출력 반전(input-output inversion) 현상 때문으로 해석되었다. 하지만, 아직 VAX1(AA) 생쥐의 좌측 눈에서 들어 온 시각 신호가 어떤 뇌 부위를 안구로 다시 전달되는지에 대한 정보가 거의 전무하기 때문에 이러한 반전된 시각-운동 신경망에 대한 이해는 부족한 상황이다. 이를 해결하기 위해 김교수팀은 시각 자극을 받은 VAX1(AA) 생쥐의 뇌를 자기 공명 영상 분석하는 공동 연구를 시작하였다. 이 연구를 통해 동물의 시각 정보가 어떤 경로로 뇌에서 처리되어 운동 신경을 활성화 할 수 있는지에 대한 이해를 심화할 수 있을 것으로 기대한다.
이번 연구는 국제학술지인 Experimental & Molecular Medicine (https://doi.org/10.1038/s12276-023-00930-4) 2월3일자로 발표됐다. KAIST 생명과학과 김진우 교수 연구팀 민광욱 박사가 제1저자로 연구를 주도하였고, 생명과학과 이승희 교수 연구팀, 바이오및뇌공학과 박영균 교수 연구팀, 연세대학교 이한웅 교수 연구팀, 한국뇌연구원 김남석 박사, 기초과학연구원 이창준 박사 연구팀이 함께 참여하였다. 본 연구는 과학기술정보통신부 중견연구자연구지원사업과 선도연구센터사업, 그리고 KAIST 국제공동연구지원사업의 지원을 받아 수행됐다.
2023.03.02 조회수 9140
왼쪽 눈이 본 것을 오른쪽 뇌가 알게 하라
인간을 비롯한 대부분 동물의 신체 기관들은 대칭적 구조를 가지고 있다. 이를 통해 몸의 좌우 균형을 맞추고, 움직이고, 반응을 할 수 있게 된다. 동물의 시각이 시작되는 안구 역시 머리의 양쪽에 하나씩 위치하며 한쪽 눈으로 볼 때 보다 더 넓은 영역의 물체를 인식할 수 있게 된다. 사람이나 고양이 같은 경우는 양쪽 눈이 정면을 향하고 있고, 개나 쥐 같은 동물은 눈이 사람보다 측면부를 향해 있고, 많은 물고기의 경우는 두 눈이 완전히 반대쪽을 향하고 있다. 이로 인해 좌우측 눈이 받아들이는 이미지 역시 차이를 보이게 된다. 인간의 경우 좌측과 우측 눈이 인식하는 이미지의 50%에 가까운 영역이 겹치는 반면, 생쥐의 경우는 5% 이하의 영역이 중복이 되며, 물고기는 중복된 영역이 거의 없다. 이들 겹치는 시각 영역 이미지의 위상차를 뇌가 인식해 동물은 물체의 입체감을 느낄 수 있다. 또, 물체가 움직이는 경우에는 좌측 눈과 우측 눈에 감지된 물체의 이미지의 시간차 정보가 뇌에서 처리되어 물체의 이동 경로를 감지하고 예측할 수 있게 된다. 결국, 중복된 시각 영역이 넓을수록 외부 물체의 입체감과 이동을 더 잘 감지할 수 있게 되어, 대부분 포식 동물들이 넓은 중복 영역을 확보하기 위해 안구를 정면에 위치하는 경우가 많다.
이렇게 좌우 안구에서 인식된 이미지를 뇌의 특정 영역에 전달하기 위해 눈에서 나온 시신경은 뇌의 좌우 반구에 모두 연결이 되어 있다. 흥미롭게 좌우 반구로 연결되는 시신경 비율은 좌우 안구 이미지의 중복 비율에 역비례해서, 인간의 경우 50% 시신경이 반대쪽 뇌로 연결되고, 생쥐의 경우 95% 내외, 물고기는 100% 반대쪽으로 연결된다. 시신경이 좌측 또는 우측 중 어느 쪽 뇌로 뻗어 나갈 것인지를 결정하는 과정은 시신경이 눈에서 출발해 시상하부 영역에 도달할 때 시상하부 중간선에 존재하는 경로 결정자(pathway selection cue)에 의해 일어난다고 알려져 있다. 오랜 동안 이러한 동물의 양안 시각계 (binocular visual system)의 핵심인 시상하부 중간선에서 경로 결정에 관련된 메커니즘을 이해하려는 시도가 있어 왔고 일부 경로 결정 인자들이 밝혀진 바도 있다. 하지만, 핵심인자의 부재로 이 과정에 대한 명확한 이해는 부족한 상황이었다.
생명과학과 김진우 교수 연구실에서는 시신경 및 시상하부 중간선에 많이 발현되는 VAX1 유전자에 대한 연구를 수행해 오고 있다. 이 유전자가 결핍된 생쥐와 사람은 시신경이 제대로 성장하지 못하고 시신경이 시상하부에서 교차하지 못하는 발달 이상을 보였다. VAX1이 호메오도메인을 가지는 전사인자의 특성을 가지고 있기 때문에 당연히 시상하부에서 경로 결정자의 유전자 발현을 조절해 시신경 교차(optic chiasm)를 생성할 것이라고 추정하였으나, 김교수 연구팀에서는 VAX1이 시상하부 세포에서 전사인자로 기능하기 보다는 눈의 망막신경절세포에서 뻗어 나온 시신경 축삭(axon)에서 mRNA 번역인자로 작용하여 시신경의 성장을 유도한다는 놀라운 사실을 발견하여 2014년 발표한 바 있다. 하지만, VAX1이 전혀 없는 동물은 두개골 기형 때문에 생존하기 어려워 이러한 VAX1 이상으로 인해 시신경 교차가 없는 동물의 시각 반응 및 행동에 대한 이해는 이루어지지 못하고 있었다.
김 교수 연구실에서 VAX1의 전사인자 기능은 유지한 채 시신경 축삭에 작용하지 못하는 VAX1(AA) 생쥐를 제작하였고, 이 생쥐는 외형적 이상이 전혀 없이 정상적으로 태어나 성장하였다. 다만, VAX1(AA) 생쥐는 모든 시신경이 안구와 같은 쪽 뇌에만 연결되는 시신경 교차 결핍증(agenesis of optic chiasm, AOC)을 나타냈다. 이 생쥐의 시각을 다양한 방법을 통해 검증한 결과, 눈 속의 신경 조직인 망막이 빛을 감지하는 기능은 정상이나 입체 시각이 전혀 없었고, 시력 역시 저하되어 있었다.
흥미로운 점은 VAX1(AA) 생쥐의 눈이 아무런 자극이 없는 상태에서도 지속적으로 상하궤도 운동을 하는 시소안구진탕증(Seesaw Nystagmus)를 보인다는 것이었다. 이러한 시소안구진탕증은 시신경 교차에 이상이 있는 사람과 벨지안쉽도그(Belgian sheepdog)에서도 관찰이 된 바 있어서 시신경 교차 결여가 VAX1(AA) 생쥐의 안구 운동 이상의 원인임을 알 수 있었다.
더욱 흥미로운 점은 VAX1(AA) 생쥐의 시각 운동 반응이 반전되어 있다는 점이었다. 왼쪽 눈에 빛을 주면 오른쪽 동공이 먼저 축소되고, 물체 이동을 감지한 후에는 움직이던 눈이 오히려 정면을 응시하는 등, 시각 정보와 반대되는 안구의 움직임을 보였다. VAX1(AA) 생쥐는 시신경 교차에만 이상이 있고 시각을 처리하는 뇌 부위는 정상적으로 형성이 되어 있기 때문에, 이 결과는 우측 눈에서 오는 신호를 처리해 우측 눈으로 운동 정보를 보내야 할 좌측뇌가 정작 좌측 눈에서 오는 신호를 받아 우측 눈을 자극하는 입력-출력 반전(input-output inversion) 현상 때문으로 해석되었다. 하지만, 아직 VAX1(AA) 생쥐의 좌측 눈에서 들어 온 시각 신호가 어떤 뇌 부위를 안구로 다시 전달되는지에 대한 정보가 거의 전무하기 때문에 이러한 반전된 시각-운동 신경망에 대한 이해는 부족한 상황이다. 이를 해결하기 위해 김교수팀은 시각 자극을 받은 VAX1(AA) 생쥐의 뇌를 자기 공명 영상 분석하는 공동 연구를 시작하였다. 이 연구를 통해 동물의 시각 정보가 어떤 경로로 뇌에서 처리되어 운동 신경을 활성화 할 수 있는지에 대한 이해를 심화할 수 있을 것으로 기대한다.
이번 연구는 국제학술지인 Experimental & Molecular Medicine (https://doi.org/10.1038/s12276-023-00930-4) 2월3일자로 발표됐다. KAIST 생명과학과 김진우 교수 연구팀 민광욱 박사가 제1저자로 연구를 주도하였고, 생명과학과 이승희 교수 연구팀, 바이오및뇌공학과 박영균 교수 연구팀, 연세대학교 이한웅 교수 연구팀, 한국뇌연구원 김남석 박사, 기초과학연구원 이창준 박사 연구팀이 함께 참여하였다. 본 연구는 과학기술정보통신부 중견연구자연구지원사업과 선도연구센터사업, 그리고 KAIST 국제공동연구지원사업의 지원을 받아 수행됐다.
2023.03.02 조회수 9140 -
 장 조직의 항상성과 염증성 장염 회복의 핵심 효소 발견
우리 대학 생명과학과 김세윤, 양한슬 교수 공동연구팀이 `장 상피조직의 발생과 염증성 장염 회복과정의 핵심 효소발굴'에 성공했다고 6일 밝혔다.
생명과학과 박승은 박사, 이동은 박사과정 학생이 공동 제1 저자로 참여한 이번 연구 결과는 국제 학술지인 `세포 및 분자 소화기학 및 간장학 (Cellular and Molecular Gastroenterology & Hepatology)'에 지난 8월 19일 字 온라인 게재됐다.
※ 논문명 : Gut Epithelial Inositol Polyphosphate Multikinase Alleviates Experimental Colitis via Governing Tuft Cell Homeostasis
※ 저자 정보 : 박승은 (한국과학기술원, 공동 제1 저자), 이동은 (한국과학기술원, 공동 제1 저자), 정재웅 (연세대학교 의과대학, 제2 저자), 이수형 (반더빌트 의과대학, 공동 저자), 박승주 (한국과학기술원, 공동 저자), 류재승 (한국과학기술원, 공동 저자), 오세규 (카이노젠, 공동 저자), 양한슬 (한국과학기술원, 공동 교신저자), 황성순 (연세대학교 의과대학, 공동 교신저자), 김세윤 (한국과학기술원, 공동 교신저자), 포함 총 10명
염증성 장 질환은 1980년대 중반까지만 해도 국내 발병률은 매우 낮은 질환이었으나 건강보험심사평가원 자료에 따르면 2011년 말 만성 염증성 장 질환인 크론병 환자가 1만3천여 명, 궤양성 대장염 환자가 2만9천여 명으로 집계됐으며 이후로도 환자 수가 빠르게 증가하고 있다.
궤양성 대장염과 크론병과 같은 염증성 장 질환(Inflammatory bowel disease)은 복통, 설사뿐 아니라 전신 무력감, 체중 감소, 장 천공 등의 합병증을 유발해 환자의 삶의 질을 크게 낮추는 심각한 질환이다.
이러한 염증성 장 질환은 아직 발병 메커니즘이 명확하게 규명되지 않았으며 서구화된 식습관과 함께 유전적 이상이나 면역학적 장애 또는 스트레스와 같은 환경적인 요인이 복합적으로 관여하는 것으로 여겨진다.
최근 염증성 장 질환의 환자군 일부에서 이노시톨 대사(체내 합성 또는 음식을 통하여 공급되는 이노시톨 영양소를 이노시톨 인산 물질들로 전환하는 세포 내 생화학적 반응)의 핵심 효소인 이노시톨 폴리인산 카이네이즈(Inositol polyphosphate multikinase, 이하 IPMK) 유전자에서 단일염기변이(single nucleotide polymorphism, SNP) 등이 보고됐다. IPMK 효소는 포유류의 이노시톨 대사에서 매우 중추적인 역할을 하는 단백질로서 세포의 성장과 대사뿐 아니라 면역 반응 및 중추 신경계 기억 조절 등의 다각적인 생물학적 기능을 수행하는 것으로 알려졌으나 소화기계에서의 생물학적 기능은 밝혀진 바가 전혀 없다.
이에 우리 대학 생명과학과 및 줄기세포 연구센터 김세윤 교수, 양한슬 교수 연구팀은 연세대학교 의과대학 황성순 교수 연구팀과의 공동연구를 통해 장 상피세포에서의 IPMK 효소의 역할규명연구를 다각적으로 수행했다.
연구팀은 먼저 장 상피세포에서 IPMK 유전자가 결손된 생쥐 연구모델을 제작했고 이를 분석한 결과에 따르면 정상적인 환경에서는 뚜렷한 장 조직의 구조와 기능에 있어 문제가 발생하지 않았다. 그러나 덱스트란 황산나트륨(DSS)을 투여해 대장염을 유발할 경우, IPMK 유전자가 결손된 생쥐로부터 대장염 회복반응이 현저하게 둔화됨을 관찰했고 이로부터 IPMK 효소가 손상된 대장 조직이 회복되는데 필수적인 인자임을 규명했다.
특히 연구팀은 IPMK 효소가 제거된 장 조직에서는 솔세포(tuft cell)의 발생과 기능에 심각한 장애가 발생함을 관찰했다. 솔세포란 소장과 대장뿐 아니라 흉선, 췌장 등에 존재하는 세포로서 표면에 미세 융모구조를 보이는 독특한 세포다. 장 조직의 전체 세포 중 불과 2~3% 이하로 존재하는 솔세포는 외부로부터의 기생충 감염에 대응하는 면역기능을 수행한다. 또한 솔세포로부터 합성돼 분비되는 아세틸콜린 신경전달물질은 장내 줄기세포 및 신경세포를 자극해 장 조직의 항상성 유지와 손상 복구에 중요한 기능을 담당하는 것으로 알려져 있다. 이러한 중요성에도 불구하고 솔세포의 발생과 기능을 조절하는 분자 타깃에 대한 이해는 극히 부족한 것으로 알려져 있다.
공동연구팀은 첨단 단일세포 RNA 유전자 배열 순서 분석기술(single cell RNA sequencing)을 통해 IPMK 효소가 제거된 장 상피조직에서 아세틸콜린 분비를 담당하는 솔세포군의 발생이 특이적으로 감소함을 규명했다. 또한 이러한 분석과정에서 생쥐의 대장에 존재하는 솔세포는 크게 면역기능을 담당하는 세포군, 아세틸콜린 분비 솔세포군 외에 유전자 발현 능력이 감소한 새로운 솔세포군이 있다는 사실을 발견해 학계에 최초 보고했다.
연구팀 관계자는 "발굴한 IPMK 효소에 기반한 아세틸콜린 분비성 솔세포의 발생 및 기능조절은 향후 장 상피조직의 항상성 유지의 학문적 이해와 더불어 염증성 장 질환의 진단 및 치료기술 개발 등에 적극적으로 활용될 것ˮ이라며 연구의 의의를 설명했다.
한편 이번 연구는 한국연구재단 중견연구자지원사업, 선도연구센터, 서경배과학재단, KAIST 줄기세포 연구센터 지원사업, KAIST 그랜드챌린지 30 (KC30) 사업 및 포스코청암펠로십의 지원을 받아 수행됐다.
2022.10.06 조회수 10689
장 조직의 항상성과 염증성 장염 회복의 핵심 효소 발견
우리 대학 생명과학과 김세윤, 양한슬 교수 공동연구팀이 `장 상피조직의 발생과 염증성 장염 회복과정의 핵심 효소발굴'에 성공했다고 6일 밝혔다.
생명과학과 박승은 박사, 이동은 박사과정 학생이 공동 제1 저자로 참여한 이번 연구 결과는 국제 학술지인 `세포 및 분자 소화기학 및 간장학 (Cellular and Molecular Gastroenterology & Hepatology)'에 지난 8월 19일 字 온라인 게재됐다.
※ 논문명 : Gut Epithelial Inositol Polyphosphate Multikinase Alleviates Experimental Colitis via Governing Tuft Cell Homeostasis
※ 저자 정보 : 박승은 (한국과학기술원, 공동 제1 저자), 이동은 (한국과학기술원, 공동 제1 저자), 정재웅 (연세대학교 의과대학, 제2 저자), 이수형 (반더빌트 의과대학, 공동 저자), 박승주 (한국과학기술원, 공동 저자), 류재승 (한국과학기술원, 공동 저자), 오세규 (카이노젠, 공동 저자), 양한슬 (한국과학기술원, 공동 교신저자), 황성순 (연세대학교 의과대학, 공동 교신저자), 김세윤 (한국과학기술원, 공동 교신저자), 포함 총 10명
염증성 장 질환은 1980년대 중반까지만 해도 국내 발병률은 매우 낮은 질환이었으나 건강보험심사평가원 자료에 따르면 2011년 말 만성 염증성 장 질환인 크론병 환자가 1만3천여 명, 궤양성 대장염 환자가 2만9천여 명으로 집계됐으며 이후로도 환자 수가 빠르게 증가하고 있다.
궤양성 대장염과 크론병과 같은 염증성 장 질환(Inflammatory bowel disease)은 복통, 설사뿐 아니라 전신 무력감, 체중 감소, 장 천공 등의 합병증을 유발해 환자의 삶의 질을 크게 낮추는 심각한 질환이다.
이러한 염증성 장 질환은 아직 발병 메커니즘이 명확하게 규명되지 않았으며 서구화된 식습관과 함께 유전적 이상이나 면역학적 장애 또는 스트레스와 같은 환경적인 요인이 복합적으로 관여하는 것으로 여겨진다.
최근 염증성 장 질환의 환자군 일부에서 이노시톨 대사(체내 합성 또는 음식을 통하여 공급되는 이노시톨 영양소를 이노시톨 인산 물질들로 전환하는 세포 내 생화학적 반응)의 핵심 효소인 이노시톨 폴리인산 카이네이즈(Inositol polyphosphate multikinase, 이하 IPMK) 유전자에서 단일염기변이(single nucleotide polymorphism, SNP) 등이 보고됐다. IPMK 효소는 포유류의 이노시톨 대사에서 매우 중추적인 역할을 하는 단백질로서 세포의 성장과 대사뿐 아니라 면역 반응 및 중추 신경계 기억 조절 등의 다각적인 생물학적 기능을 수행하는 것으로 알려졌으나 소화기계에서의 생물학적 기능은 밝혀진 바가 전혀 없다.
이에 우리 대학 생명과학과 및 줄기세포 연구센터 김세윤 교수, 양한슬 교수 연구팀은 연세대학교 의과대학 황성순 교수 연구팀과의 공동연구를 통해 장 상피세포에서의 IPMK 효소의 역할규명연구를 다각적으로 수행했다.
연구팀은 먼저 장 상피세포에서 IPMK 유전자가 결손된 생쥐 연구모델을 제작했고 이를 분석한 결과에 따르면 정상적인 환경에서는 뚜렷한 장 조직의 구조와 기능에 있어 문제가 발생하지 않았다. 그러나 덱스트란 황산나트륨(DSS)을 투여해 대장염을 유발할 경우, IPMK 유전자가 결손된 생쥐로부터 대장염 회복반응이 현저하게 둔화됨을 관찰했고 이로부터 IPMK 효소가 손상된 대장 조직이 회복되는데 필수적인 인자임을 규명했다.
특히 연구팀은 IPMK 효소가 제거된 장 조직에서는 솔세포(tuft cell)의 발생과 기능에 심각한 장애가 발생함을 관찰했다. 솔세포란 소장과 대장뿐 아니라 흉선, 췌장 등에 존재하는 세포로서 표면에 미세 융모구조를 보이는 독특한 세포다. 장 조직의 전체 세포 중 불과 2~3% 이하로 존재하는 솔세포는 외부로부터의 기생충 감염에 대응하는 면역기능을 수행한다. 또한 솔세포로부터 합성돼 분비되는 아세틸콜린 신경전달물질은 장내 줄기세포 및 신경세포를 자극해 장 조직의 항상성 유지와 손상 복구에 중요한 기능을 담당하는 것으로 알려져 있다. 이러한 중요성에도 불구하고 솔세포의 발생과 기능을 조절하는 분자 타깃에 대한 이해는 극히 부족한 것으로 알려져 있다.
공동연구팀은 첨단 단일세포 RNA 유전자 배열 순서 분석기술(single cell RNA sequencing)을 통해 IPMK 효소가 제거된 장 상피조직에서 아세틸콜린 분비를 담당하는 솔세포군의 발생이 특이적으로 감소함을 규명했다. 또한 이러한 분석과정에서 생쥐의 대장에 존재하는 솔세포는 크게 면역기능을 담당하는 세포군, 아세틸콜린 분비 솔세포군 외에 유전자 발현 능력이 감소한 새로운 솔세포군이 있다는 사실을 발견해 학계에 최초 보고했다.
연구팀 관계자는 "발굴한 IPMK 효소에 기반한 아세틸콜린 분비성 솔세포의 발생 및 기능조절은 향후 장 상피조직의 항상성 유지의 학문적 이해와 더불어 염증성 장 질환의 진단 및 치료기술 개발 등에 적극적으로 활용될 것ˮ이라며 연구의 의의를 설명했다.
한편 이번 연구는 한국연구재단 중견연구자지원사업, 선도연구센터, 서경배과학재단, KAIST 줄기세포 연구센터 지원사업, KAIST 그랜드챌린지 30 (KC30) 사업 및 포스코청암펠로십의 지원을 받아 수행됐다.
2022.10.06 조회수 10689 -
 RNA를 통한 유전자 전사 조절 원리 규명
세포가 어떤 유전자를 얼마나 발현하느냐에 따라 그 세포의 모양, 기능, 수명 등이 결정되므로 유전정보를 처음으로 발현하는 RNA 합성효소의 활성은 세포 내에서 매우 중요하게, 또 정교하게 조절된다. 그러나 이러한 유전자 전사(transcription) 조절의 중요성에도 불구하고 RNA 합성효소가 이러한 단백질과 RNA들에 의해서 어떻게 조절되는지 분자적인 수준에서는 잘 알려져 있지 않았다.
☞ 유전자 전사: DNA의 유전정보가 RNA에 옮겨지는 과정을 말한다. 유전정보의 복사물인 RNA는 단백질 합성에 사용된다.
우리 대학 화학과 강진영 교수 연구팀이 RNA를 통한 RNA 합성효소의 조절 메커니즘을 알아내고자 RNA 합성효소와 RNA 합성효소를 조절하는 바이러스 유래 RNA인 *HK022 putRNA의 결합 구조를 초저온 전자현미경(cryo-EM)으로 규명하여 유전자 전사조절의 기초 원리를 규명했다고 7일 밝혔다.
*HK022 putRNA: HK022 박테리오파지(박테리아를 감염시키는 바이러스)의 RNA로 다른 단백질의 도움 없이 해당 RNA를 만든 RNA 중합효소와 결합해 RNA 합성이 계속 되도록 RNA 중합효소를 조절
화학과 황승하 박사과정이 제1 저자로 참여한 이번 연구는 국제 학술지 `네이처 커뮤니케이션즈 (Nature Communications)'에 지난 8월 15일 출판되었다. (논문명: Structural basis of transcriptional regulation by a nascent RNA element, HK022 putRNA).
HK022 putRNA는 RNA 합성효소와 결합해서 RNA 합성이 멈추지 않고 계속 되도록 도와주는 역할을 한다. 이러한 기능을 이해하기 위해서 본 연구팀은 putRNA와 RNA 합성효소의 결합 복합체(put-associated RNA polymerase elongation complex, putEC)의 세 가지 구조를 초저온 전자현미경으로 규명하였다.
이 연구에서는 활성을 가진 putRNA를 제작하기 위해 장애물 단백질을 RNA 합성에 활용하는 방법을 고안하였으며, 초저온 전자현미경 촬영 결과 예상하지 못했던 세 종류의 복합체 – putRNA가 잘 접혀서 RNA 합성효소와 결합하고 있는 putEC, put RNA가 접히지 않은 put-없는 EC, 잘 접힌 putRNA와 시그마 단백질이 함께 RNA 합성효소와 결합하고 있는 시그마* 결합-putEC – 를 발견할 수 있었다. (그림 1)
*시그마: RNA 합성효소가 유전자 RNA 합성을 처음 시작할 때 필요한 단백질로 RNA 합성이 어느 정도 안정화되면 RNA 합성효소에서 떨어진다.
연구팀은 이들 복합체의 구조를 통해 putRNA가 이전 연구에서 예측된 대로 RNA 합성효소와 안정적으로 결합하고 있지만 예측과 달리 예상보다 더 많은 염기쌍(base pair)을 사용해 RNA 이중나선(double helix) 뿐 아니라 삼중나선(triple helix)을 형성하는 것을 확인하였다. 또한, putRNA가 RNA 합성효소와 결합하면 RNA 합성효소가 RNA 합성을 잠시 멈출 때 가지는 구조의 변화를 방해해서 RNA 합성을 지속하도록 한다는 가설을 제시할 수 있었다.
한편, 시그마 단백질(σ70)은 RNA 합성효소가 전사를 시작할 때 필요한 전사 개시인자로, RNA 합성이 안정되면 RNA 합성효소에서 떨어졌다가 특정 DNA 서열(–10-유사 서열)이 있으면 전사 과정 중이라도 다시 RNA 중합효소와 결합해 RNA 합성을 일시적으로 멈추는 것으로 알려져 있다. 이번 연구에서는 예상치 못하게 관찰된 시그마 결합-putEC 구조를 통해 시그마가 RNA 합성효소와 결합하여 RNA 합성이 잠깐 멈추면 putRNA가 더 잘 접힌다는 것을 알 수 있었다.
이 연구의 교신저자인 강진영 교수는 "RNA 합성효소는 세포 내에 저장된 유전 정보를 처음으로 꺼내어 생명활동에 활용하는, 세포 내에서 제일 중요한 단백질 중 하나이다. 그러나 RNA 합성효소의 큰 크기와 다양한 구조 변화 때문에 이전에 주로 활용하던 X-ray 결정학 방식으로는 그 구조를 관찰하기가 어려웠다. 최근 초저온 전자현미경의 발달로 이제야 조금씩 RNA 합성효소의 작동 원리가 알려지고 있는 상황이다. 이번 연구는 이전에 잘 알려지지 않았던, RNA를 통한 전사 조절의 기초적인 원리를 설명한 것으로, RNA를 통한 RNA 합성효소 조절의 다양한 전략을 밝혀줄 시작점이며, 더 나아가 유전자 발현을 조작할 수 있는 RNA의 개발을 도울 수 있는 정보를 제공할 것이라 기대한다.ˮ고 밝혔다.
한편 이번 연구는 한국연구재단의 이공분야기초연구사업(우수신진연구)과 원천기술개발사업의 지원을 받아 수행됐다.
2022.09.07 조회수 13451
RNA를 통한 유전자 전사 조절 원리 규명
세포가 어떤 유전자를 얼마나 발현하느냐에 따라 그 세포의 모양, 기능, 수명 등이 결정되므로 유전정보를 처음으로 발현하는 RNA 합성효소의 활성은 세포 내에서 매우 중요하게, 또 정교하게 조절된다. 그러나 이러한 유전자 전사(transcription) 조절의 중요성에도 불구하고 RNA 합성효소가 이러한 단백질과 RNA들에 의해서 어떻게 조절되는지 분자적인 수준에서는 잘 알려져 있지 않았다.
☞ 유전자 전사: DNA의 유전정보가 RNA에 옮겨지는 과정을 말한다. 유전정보의 복사물인 RNA는 단백질 합성에 사용된다.
우리 대학 화학과 강진영 교수 연구팀이 RNA를 통한 RNA 합성효소의 조절 메커니즘을 알아내고자 RNA 합성효소와 RNA 합성효소를 조절하는 바이러스 유래 RNA인 *HK022 putRNA의 결합 구조를 초저온 전자현미경(cryo-EM)으로 규명하여 유전자 전사조절의 기초 원리를 규명했다고 7일 밝혔다.
*HK022 putRNA: HK022 박테리오파지(박테리아를 감염시키는 바이러스)의 RNA로 다른 단백질의 도움 없이 해당 RNA를 만든 RNA 중합효소와 결합해 RNA 합성이 계속 되도록 RNA 중합효소를 조절
화학과 황승하 박사과정이 제1 저자로 참여한 이번 연구는 국제 학술지 `네이처 커뮤니케이션즈 (Nature Communications)'에 지난 8월 15일 출판되었다. (논문명: Structural basis of transcriptional regulation by a nascent RNA element, HK022 putRNA).
HK022 putRNA는 RNA 합성효소와 결합해서 RNA 합성이 멈추지 않고 계속 되도록 도와주는 역할을 한다. 이러한 기능을 이해하기 위해서 본 연구팀은 putRNA와 RNA 합성효소의 결합 복합체(put-associated RNA polymerase elongation complex, putEC)의 세 가지 구조를 초저온 전자현미경으로 규명하였다.
이 연구에서는 활성을 가진 putRNA를 제작하기 위해 장애물 단백질을 RNA 합성에 활용하는 방법을 고안하였으며, 초저온 전자현미경 촬영 결과 예상하지 못했던 세 종류의 복합체 – putRNA가 잘 접혀서 RNA 합성효소와 결합하고 있는 putEC, put RNA가 접히지 않은 put-없는 EC, 잘 접힌 putRNA와 시그마 단백질이 함께 RNA 합성효소와 결합하고 있는 시그마* 결합-putEC – 를 발견할 수 있었다. (그림 1)
*시그마: RNA 합성효소가 유전자 RNA 합성을 처음 시작할 때 필요한 단백질로 RNA 합성이 어느 정도 안정화되면 RNA 합성효소에서 떨어진다.
연구팀은 이들 복합체의 구조를 통해 putRNA가 이전 연구에서 예측된 대로 RNA 합성효소와 안정적으로 결합하고 있지만 예측과 달리 예상보다 더 많은 염기쌍(base pair)을 사용해 RNA 이중나선(double helix) 뿐 아니라 삼중나선(triple helix)을 형성하는 것을 확인하였다. 또한, putRNA가 RNA 합성효소와 결합하면 RNA 합성효소가 RNA 합성을 잠시 멈출 때 가지는 구조의 변화를 방해해서 RNA 합성을 지속하도록 한다는 가설을 제시할 수 있었다.
한편, 시그마 단백질(σ70)은 RNA 합성효소가 전사를 시작할 때 필요한 전사 개시인자로, RNA 합성이 안정되면 RNA 합성효소에서 떨어졌다가 특정 DNA 서열(–10-유사 서열)이 있으면 전사 과정 중이라도 다시 RNA 중합효소와 결합해 RNA 합성을 일시적으로 멈추는 것으로 알려져 있다. 이번 연구에서는 예상치 못하게 관찰된 시그마 결합-putEC 구조를 통해 시그마가 RNA 합성효소와 결합하여 RNA 합성이 잠깐 멈추면 putRNA가 더 잘 접힌다는 것을 알 수 있었다.
이 연구의 교신저자인 강진영 교수는 "RNA 합성효소는 세포 내에 저장된 유전 정보를 처음으로 꺼내어 생명활동에 활용하는, 세포 내에서 제일 중요한 단백질 중 하나이다. 그러나 RNA 합성효소의 큰 크기와 다양한 구조 변화 때문에 이전에 주로 활용하던 X-ray 결정학 방식으로는 그 구조를 관찰하기가 어려웠다. 최근 초저온 전자현미경의 발달로 이제야 조금씩 RNA 합성효소의 작동 원리가 알려지고 있는 상황이다. 이번 연구는 이전에 잘 알려지지 않았던, RNA를 통한 전사 조절의 기초적인 원리를 설명한 것으로, RNA를 통한 RNA 합성효소 조절의 다양한 전략을 밝혀줄 시작점이며, 더 나아가 유전자 발현을 조작할 수 있는 RNA의 개발을 도울 수 있는 정보를 제공할 것이라 기대한다.ˮ고 밝혔다.
한편 이번 연구는 한국연구재단의 이공분야기초연구사업(우수신진연구)과 원천기술개발사업의 지원을 받아 수행됐다.
2022.09.07 조회수 13451 -
 질병 세포만 찾아 교정치료 가능한 유전자 가위 시스템 개발
우리 대학 의과학대학원 이지민 교수 연구팀이 한국과학기술연구원(KIST) 오승자 선임연구원, 강원대학교 이주용 교수와 공동 연구를 통해 질병 세포에서만 핵 내 유전자 교정을 수행할 수 있는 유전자 가위 시스템(CRISPR/Cas9)을 개발했다고 14일 밝혔다.
연구팀은 세포 내 마이크로RNA가 특정 서열을 인식해 절단한다는 특성을 활용해, 질병 세포에서 과발현되는 마이크로RNA에 의해 특이적으로 절단될 수 있는 링커를 연결한 유전자 가위 시스템을 설계했다. 이렇게 설계된 시스템은 질병 세포 특이적 마이크로RNA가 적은 정상세포에서는 세포질에 머물러 유전자 교정을 수행하지 않지만, 질병 세포에서는 링커가 절단되면서 유전자 가위가 세포핵으로 들어가 유전자 교정을 수행할 수 있다.
이러한 플랫폼은 유전자 가위를 질병 세포에서만 기능 할 수 있게 해 정상세포와 질병 세포가 혼합돼있는 실제 환자에게도 효과적인 유전자 교정 치료를 진행할 수 있을 것으로 기대된다.
KIST 신철희 박사와 우리 대학 의과학대학원 박수찬 연구원이 공동 제1 저자로 참여한 이번 연구는 국제 학술지 `뉴클레익 엑시드 리서치(Nucleic Acids Research, IF 16.971)' 온라인판에 지난달 30일 자 출판됐다. (논문명 : Cytosolic microRNA-inducible nuclear translocation of Cas9 protein for disease-specific genome modification).
마이크로RNA는 유전자를 전사 후 조절하는 19~24 뉴클레오티드(DNA나 RNA의 기본 단위) 길이의 RNA다. 마이크로RNA는 DNA로부터 전사된 메신저 RNA에 아르고너트(Argonaute; Ago) 단백질을 통해 결합하며, 결합한 메신저 RNA를 절단한다. 마이크로RNA의 비정상적인 발현이 다양한 질병에서 보고되고 있으며, 질병의 치료를 위한 표적 바이오마커로 많이 연구되고 있다.
다양한 질병에서 마이크로RNA를 표적으로 하는 치료법들이 빠르게 연구되고 있지만, 치료 물질의 전달 및 투여량의 문제, 세포 독성 및 비정상적 면역 반응 활성화 등의 문제가 있다.
유전자 가위 시스템은 단일 가이드 RNA(single guide RNA)를 조합해 정교한 유전자 교정을 수행하는 매우 효과적인 도구다. 하지만, 이 시스템의 실제 활용에는 기술적 한계들이 존재한다. 가장 큰 문제는 안정성 문제로, 표적 유전자가 아닌 다른 유전자를 편집하는 오프-타겟 이펙트(off-target effect)다. 또한, 다양한 세포가 혼합된 환경에서는 유전자 교정을 수행하기 어렵다.
연구팀은 이러한 문제를 해결하기 위해 질병 세포 본연의 생태를 활용하는 접근법을 고안했다. 연구팀은 핵 위치 신호(Nuclear localization signal; NLS)가 부착된 기존 유전자 가위(Cas9)에 핵 외 수송신호(Nuclear export signal; NES)를 연결한 질병 세포 마이크로RNA의 메신저 RNA 표적 서열을 결합한 유전자 가위를 제작했고, 이를 유전자 가위 `셀프 체크인'으로 명명했다.
연구팀은 인간 질병 세포에서 과발현되는 마이크로RNA-21의 표적 서열과 실험용 쥐의 마이크로RNA-294의 표적 서열을 연결한 유전자 가위의 인간 질병 세포 내 유전자 교정 기능을 비교했고, 마이크로RNA-21 표적 서열 연결 유전자 가위만이 세포 내 마이크로RNA-21에 의해 절단돼 핵까지 전달되어 기능을 수행할 수 있음을 확인했다.
연구팀은 다양한 폐암 세포에서 마이크로RNA-21의 발현량과 발암 단백질 Ezh2가 양의 상관관계가 있다는 것을 증명했고, `셀프 체크인'을 적용해 마이크로RNA-21이 과발현된 폐암 세포에서 발암 유전자 Ezh2의 유전자 교정을 성공적으로 수행했다.
또한, 암세포는 항암 약물에 지속해서 노출되게 되면, 약물 저항성을 획득하게 되는데, 연구팀은 폐암 세포에서 마이크로RNA-21과 Ezh2의 발현이 항암 약물 시스플라틴을 투여하면 오히려 증가함을 확인했다. 유전자 가위 셀프 체크인 기술을 통한 Ezh2 유전자 교정과 항암제(시스플라틴)의 병행 사용은 폐암 세포의 성장을 더욱 효과적으로 억제할 수 있음을 마우스 실험을 통해서 밝혔다.
연구팀이 개발한 유전자 가위 셀프 체크인 기술은 질병 세포에서만 기능하기 때문에, 오프-타겟 이펙트를 최소화할 수 있다는 장점이 있으며, 세포 내 시스템을 활용한다는 점에서 안정성이 높다고 할 수 있다. 또한, 단일 가이드 RNA 및 메신저 RNA 표적 서열을 상황에 맞게 교체해 사용할 수 있어, 다양한 질병에 적용이 가능할 것으로 기대된다.
연구팀은 "유전자 가위 셀프 체크인 기술은 기존 유전자 가위 시스템의 문제를 개선해, 높은 특이성을 가지고 질병 세포에 대한 유전자를 세포 특이적으로 교정할 수 있다는 것을 확인할 수 있다ˮ 라며 "다양한 질병 연관 마이크로RNA에 대응해 기술을 적용할 수 있을 것이다ˮ 라고 전했다.
한편 이번 연구는 삼성미래기술육성사업, 한국연구재단 이공분야기초연구사업 및 한국과학기술연구원 지원을 받아 수행됐다.
2022.06.14 조회수 15639
질병 세포만 찾아 교정치료 가능한 유전자 가위 시스템 개발
우리 대학 의과학대학원 이지민 교수 연구팀이 한국과학기술연구원(KIST) 오승자 선임연구원, 강원대학교 이주용 교수와 공동 연구를 통해 질병 세포에서만 핵 내 유전자 교정을 수행할 수 있는 유전자 가위 시스템(CRISPR/Cas9)을 개발했다고 14일 밝혔다.
연구팀은 세포 내 마이크로RNA가 특정 서열을 인식해 절단한다는 특성을 활용해, 질병 세포에서 과발현되는 마이크로RNA에 의해 특이적으로 절단될 수 있는 링커를 연결한 유전자 가위 시스템을 설계했다. 이렇게 설계된 시스템은 질병 세포 특이적 마이크로RNA가 적은 정상세포에서는 세포질에 머물러 유전자 교정을 수행하지 않지만, 질병 세포에서는 링커가 절단되면서 유전자 가위가 세포핵으로 들어가 유전자 교정을 수행할 수 있다.
이러한 플랫폼은 유전자 가위를 질병 세포에서만 기능 할 수 있게 해 정상세포와 질병 세포가 혼합돼있는 실제 환자에게도 효과적인 유전자 교정 치료를 진행할 수 있을 것으로 기대된다.
KIST 신철희 박사와 우리 대학 의과학대학원 박수찬 연구원이 공동 제1 저자로 참여한 이번 연구는 국제 학술지 `뉴클레익 엑시드 리서치(Nucleic Acids Research, IF 16.971)' 온라인판에 지난달 30일 자 출판됐다. (논문명 : Cytosolic microRNA-inducible nuclear translocation of Cas9 protein for disease-specific genome modification).
마이크로RNA는 유전자를 전사 후 조절하는 19~24 뉴클레오티드(DNA나 RNA의 기본 단위) 길이의 RNA다. 마이크로RNA는 DNA로부터 전사된 메신저 RNA에 아르고너트(Argonaute; Ago) 단백질을 통해 결합하며, 결합한 메신저 RNA를 절단한다. 마이크로RNA의 비정상적인 발현이 다양한 질병에서 보고되고 있으며, 질병의 치료를 위한 표적 바이오마커로 많이 연구되고 있다.
다양한 질병에서 마이크로RNA를 표적으로 하는 치료법들이 빠르게 연구되고 있지만, 치료 물질의 전달 및 투여량의 문제, 세포 독성 및 비정상적 면역 반응 활성화 등의 문제가 있다.
유전자 가위 시스템은 단일 가이드 RNA(single guide RNA)를 조합해 정교한 유전자 교정을 수행하는 매우 효과적인 도구다. 하지만, 이 시스템의 실제 활용에는 기술적 한계들이 존재한다. 가장 큰 문제는 안정성 문제로, 표적 유전자가 아닌 다른 유전자를 편집하는 오프-타겟 이펙트(off-target effect)다. 또한, 다양한 세포가 혼합된 환경에서는 유전자 교정을 수행하기 어렵다.
연구팀은 이러한 문제를 해결하기 위해 질병 세포 본연의 생태를 활용하는 접근법을 고안했다. 연구팀은 핵 위치 신호(Nuclear localization signal; NLS)가 부착된 기존 유전자 가위(Cas9)에 핵 외 수송신호(Nuclear export signal; NES)를 연결한 질병 세포 마이크로RNA의 메신저 RNA 표적 서열을 결합한 유전자 가위를 제작했고, 이를 유전자 가위 `셀프 체크인'으로 명명했다.
연구팀은 인간 질병 세포에서 과발현되는 마이크로RNA-21의 표적 서열과 실험용 쥐의 마이크로RNA-294의 표적 서열을 연결한 유전자 가위의 인간 질병 세포 내 유전자 교정 기능을 비교했고, 마이크로RNA-21 표적 서열 연결 유전자 가위만이 세포 내 마이크로RNA-21에 의해 절단돼 핵까지 전달되어 기능을 수행할 수 있음을 확인했다.
연구팀은 다양한 폐암 세포에서 마이크로RNA-21의 발현량과 발암 단백질 Ezh2가 양의 상관관계가 있다는 것을 증명했고, `셀프 체크인'을 적용해 마이크로RNA-21이 과발현된 폐암 세포에서 발암 유전자 Ezh2의 유전자 교정을 성공적으로 수행했다.
또한, 암세포는 항암 약물에 지속해서 노출되게 되면, 약물 저항성을 획득하게 되는데, 연구팀은 폐암 세포에서 마이크로RNA-21과 Ezh2의 발현이 항암 약물 시스플라틴을 투여하면 오히려 증가함을 확인했다. 유전자 가위 셀프 체크인 기술을 통한 Ezh2 유전자 교정과 항암제(시스플라틴)의 병행 사용은 폐암 세포의 성장을 더욱 효과적으로 억제할 수 있음을 마우스 실험을 통해서 밝혔다.
연구팀이 개발한 유전자 가위 셀프 체크인 기술은 질병 세포에서만 기능하기 때문에, 오프-타겟 이펙트를 최소화할 수 있다는 장점이 있으며, 세포 내 시스템을 활용한다는 점에서 안정성이 높다고 할 수 있다. 또한, 단일 가이드 RNA 및 메신저 RNA 표적 서열을 상황에 맞게 교체해 사용할 수 있어, 다양한 질병에 적용이 가능할 것으로 기대된다.
연구팀은 "유전자 가위 셀프 체크인 기술은 기존 유전자 가위 시스템의 문제를 개선해, 높은 특이성을 가지고 질병 세포에 대한 유전자를 세포 특이적으로 교정할 수 있다는 것을 확인할 수 있다ˮ 라며 "다양한 질병 연관 마이크로RNA에 대응해 기술을 적용할 수 있을 것이다ˮ 라고 전했다.
한편 이번 연구는 삼성미래기술육성사업, 한국연구재단 이공분야기초연구사업 및 한국과학기술연구원 지원을 받아 수행됐다.
2022.06.14 조회수 15639 -
 RNA 합성의 세 갈래 끝내기 제시
우리 대학 생명과학과 강창원 명예교수와 서울대학교 물리천문학부 홍성철 교수의 공동 연구팀이 우리 대학 생명과학과 서연수 교수, 우리 대학 화학과 강진영 교수 연구팀과 협업 연구를 통해 RNA 합성 종결인자의 작동원리에 관한 여러 오래된 논쟁을 한꺼번에 잠재울 수 있는 ‘세 갈래 끝내기’를 제시했다고 30일 밝혔다.
유전자가 발현되는 첫 단계는 유전자 DNA에 맞춰 RNA 중합효소가 RNA를 합성함으로써 유전정보가 DNA 거푸집에서 RNA 생산물로 복사되는 이른바 전사 과정이다. 이 전사가 어떻게 마무리되는지를 연구하면서, RNA 합성을 끝내게 하는 대장균 전사종결인자 단백질의 작동원리를 규명해, 지난 30년 가까이 여러 학설이 대립해 온 논란에 종지부를 찍을 전망이다.
전사 과정에서 중합효소에 거푸집 DNA와 생산물 RNA가 함께 붙어있는 전사 복합체로부터 RNA가 분리되는 종결 단계에 이르기 전에 종결인자가 RNA의 특정 위치에 있는 종결 신호에 먼저 붙은 후 앞서 있는 중합효소를 ‘쫓아가서’ 전사를 종결한다는 작동원리가 1977년에 처음 제시됐다.
이와 다르게, 종결인자가 중합효소에 미리 붙어있다가 RNA 종결 신호를 ‘기다려서’ 전사를 끝낸다는 원리가 1994년에 제안돼 그 후 지금까지 28년 동안 종결 준비 단계에 관해서 두 학설이 맞서 왔는데, 이번 연구에서 둘 다 실제 일어난다는 것이 확증됐다. 즉, 종결인자가 쫓아가기도 하고 기다리기도 하는데, 서로 배타적이지 않고 함께 공존한다는 점이 새롭다.
그 외에, 생산물 RNA가 분리되는 종결 방식에 관해서도 학설이 분분했다. 예를 들어, 종결인자가 전사 복합체에 있는 RNA를 잡아당겨 벗겨내는 방식으로 분리한다는 주장이 2002년에 나왔고, 이에 반해, 종결인자가 중합효소를 밀어냄으로써 RNA가 분리된다는 설이 2006년에 제안돼 대립했다.
또한, 전사 종결로 RNA가 분리된 후 결과적으로, DNA는 중합효소에 남아서 곧장 재사용돼 쉽게 전사를 다시 시작할 수 있기도 하고, 그렇지 않으면 RNA와 동시에 DNA마저 중합효소에서 떨어져서 전사 복합체가 일시에 무너져 재사용이 지체되기도 한다는 것이 강창원‧홍성철 공동 연구팀에 의해 근래 2020년에 밝혀지기도 했다.
이번 연구에서, 준비에 관한 2가지, 방식 2가지, 결과 2가지 학설이 모두 실제 확증됐다. 그렇다면 준비-방식-결과의 조합으로 총 2×2×2, 즉 8가지가 가능한데, 실제로는 일부가 밀접하게 연계돼서 3가지 조합만이 실행된다고 밝혀졌다. 그래서 ‘세 갈래 끝내기’라고 명명됐다. 더욱이, 각 갈래의 진행 속도가 서로 달라서 세 차례의 기회가 있는 셈이다.
첫 기회의 갈래에서, 쫓아가는 종결인자가 전사 복합체에서 RNA를 잡아당겨 떼어내고 DNA는 중합효소에 남겨두는 방식의 종결을 수행한다. 이것에 실패하면, 쫓아가는 종결인자가 중합효소를 밀어내서 DNA와 RNA 둘 다 떨어뜨리는 종결을 진행하는데, 이 경우가 가장 흔하다. 마지막에, 기다리는 종결인자가 중합효소를 밀쳐내서 모두 갈라놓는 종결을 단행한다.
기다리는 종결인자가 쫓아가는 종결인자보다 중합효소와 먼저 만나기 때문에 더 빠르게 더 일찍 작동하리라고 전문가들이 추정해왔는데, 실제로는 놀랍게도 기다리는 종결인자가 더 느려서 맨 마지막 기회를 얻는 것으로 드러났다. 이 세 갈래의 진행 순서는 서로 다른 염기서열의 여러 종결자 DNA에서 바뀌지 않는다는 것도 확인했다.
공동 연구팀은 이번 연구에서 거푸집 DNA와 생산물 RNA에 각기 다른 형광물질을 붙인 후 중합효소, DNA, RNA 셋이 결합한 전사 복합체가 어떻게 변하는지를 낱개로 실시간 관찰하는 생물리학적 연구기법을 창안하고, 기존에 제안됐던 여러 작동원리를 검증하는 실험을 여러 가지 수행했다.
특히, 종결인자가 어떻게 전사 종결을 유도하는지 보기 위해, 종결인자가 RNA 종결 신호에 먼저 붙은 후 중합효소를 쫓아가서 끝내는 것을 측정하거나, 그렇지 않고 종결인자가 중합효소에 미리 붙어있다가 종결 신호를 기다려서 끝내는 것을 측정하는 분별 계측법을 독창적으로 개발했다.
첨단 기술인 단일분자 실험을 전담한 서울대 물리천문학부 송은호 박사과정 학생이 제1 저자로 참여한 논문(제목: Rho-dependent transcription termination proceeds via three routes)이 저명 국제학술지 네이처 커뮤니케이션스(Nature Communications)에 올해 3월 29일 字 게재됐다. 서울대 엄희수 박사, 우리 대학 팔린다 무나싱하 박사, 우리 대학 황승하 대학원생도 참여하여 저자가 총 8명이다.
이번 공동 연구에 단일분자 형광 기술을 구사하는 물리학자, 유전자 발현이라는 기본적 생명현상을 탐구하는 생명과학자, 중합효소나 종결인자와 같은 단백질의 구조를 규명하는 화학자가 두루 참가했다. 다양한 분야의 전공자가 협업한 다학제 기초연구의 우수 사례이며, 한국연구재단 중견연구자지원사업, KAIST 고위험‧고성과 연구사업 등의 지원을 받았다.
송은호 제1 저자는 "어느 학설이 맞는지를 검증하기 위해, DNA의 한 곳에서 종결하는 종결자의 경우 작동이 단순명료하리라 짐작하고 첫 실험 대상으로 삼았는데, 뜻밖에 여러 작동원리가 모두 관찰됐고 세 갈래로 복잡했다”라며 “여러 곳에서 종결하는 다른 종결자 DNA의 경우 오히려 세 갈래가 다 보이지 않기도 해 첫 선택에 운이 따랐던 것 같다”고 말했다.
2022.03.30 조회수 10760
RNA 합성의 세 갈래 끝내기 제시
우리 대학 생명과학과 강창원 명예교수와 서울대학교 물리천문학부 홍성철 교수의 공동 연구팀이 우리 대학 생명과학과 서연수 교수, 우리 대학 화학과 강진영 교수 연구팀과 협업 연구를 통해 RNA 합성 종결인자의 작동원리에 관한 여러 오래된 논쟁을 한꺼번에 잠재울 수 있는 ‘세 갈래 끝내기’를 제시했다고 30일 밝혔다.
유전자가 발현되는 첫 단계는 유전자 DNA에 맞춰 RNA 중합효소가 RNA를 합성함으로써 유전정보가 DNA 거푸집에서 RNA 생산물로 복사되는 이른바 전사 과정이다. 이 전사가 어떻게 마무리되는지를 연구하면서, RNA 합성을 끝내게 하는 대장균 전사종결인자 단백질의 작동원리를 규명해, 지난 30년 가까이 여러 학설이 대립해 온 논란에 종지부를 찍을 전망이다.
전사 과정에서 중합효소에 거푸집 DNA와 생산물 RNA가 함께 붙어있는 전사 복합체로부터 RNA가 분리되는 종결 단계에 이르기 전에 종결인자가 RNA의 특정 위치에 있는 종결 신호에 먼저 붙은 후 앞서 있는 중합효소를 ‘쫓아가서’ 전사를 종결한다는 작동원리가 1977년에 처음 제시됐다.
이와 다르게, 종결인자가 중합효소에 미리 붙어있다가 RNA 종결 신호를 ‘기다려서’ 전사를 끝낸다는 원리가 1994년에 제안돼 그 후 지금까지 28년 동안 종결 준비 단계에 관해서 두 학설이 맞서 왔는데, 이번 연구에서 둘 다 실제 일어난다는 것이 확증됐다. 즉, 종결인자가 쫓아가기도 하고 기다리기도 하는데, 서로 배타적이지 않고 함께 공존한다는 점이 새롭다.
그 외에, 생산물 RNA가 분리되는 종결 방식에 관해서도 학설이 분분했다. 예를 들어, 종결인자가 전사 복합체에 있는 RNA를 잡아당겨 벗겨내는 방식으로 분리한다는 주장이 2002년에 나왔고, 이에 반해, 종결인자가 중합효소를 밀어냄으로써 RNA가 분리된다는 설이 2006년에 제안돼 대립했다.
또한, 전사 종결로 RNA가 분리된 후 결과적으로, DNA는 중합효소에 남아서 곧장 재사용돼 쉽게 전사를 다시 시작할 수 있기도 하고, 그렇지 않으면 RNA와 동시에 DNA마저 중합효소에서 떨어져서 전사 복합체가 일시에 무너져 재사용이 지체되기도 한다는 것이 강창원‧홍성철 공동 연구팀에 의해 근래 2020년에 밝혀지기도 했다.
이번 연구에서, 준비에 관한 2가지, 방식 2가지, 결과 2가지 학설이 모두 실제 확증됐다. 그렇다면 준비-방식-결과의 조합으로 총 2×2×2, 즉 8가지가 가능한데, 실제로는 일부가 밀접하게 연계돼서 3가지 조합만이 실행된다고 밝혀졌다. 그래서 ‘세 갈래 끝내기’라고 명명됐다. 더욱이, 각 갈래의 진행 속도가 서로 달라서 세 차례의 기회가 있는 셈이다.
첫 기회의 갈래에서, 쫓아가는 종결인자가 전사 복합체에서 RNA를 잡아당겨 떼어내고 DNA는 중합효소에 남겨두는 방식의 종결을 수행한다. 이것에 실패하면, 쫓아가는 종결인자가 중합효소를 밀어내서 DNA와 RNA 둘 다 떨어뜨리는 종결을 진행하는데, 이 경우가 가장 흔하다. 마지막에, 기다리는 종결인자가 중합효소를 밀쳐내서 모두 갈라놓는 종결을 단행한다.
기다리는 종결인자가 쫓아가는 종결인자보다 중합효소와 먼저 만나기 때문에 더 빠르게 더 일찍 작동하리라고 전문가들이 추정해왔는데, 실제로는 놀랍게도 기다리는 종결인자가 더 느려서 맨 마지막 기회를 얻는 것으로 드러났다. 이 세 갈래의 진행 순서는 서로 다른 염기서열의 여러 종결자 DNA에서 바뀌지 않는다는 것도 확인했다.
공동 연구팀은 이번 연구에서 거푸집 DNA와 생산물 RNA에 각기 다른 형광물질을 붙인 후 중합효소, DNA, RNA 셋이 결합한 전사 복합체가 어떻게 변하는지를 낱개로 실시간 관찰하는 생물리학적 연구기법을 창안하고, 기존에 제안됐던 여러 작동원리를 검증하는 실험을 여러 가지 수행했다.
특히, 종결인자가 어떻게 전사 종결을 유도하는지 보기 위해, 종결인자가 RNA 종결 신호에 먼저 붙은 후 중합효소를 쫓아가서 끝내는 것을 측정하거나, 그렇지 않고 종결인자가 중합효소에 미리 붙어있다가 종결 신호를 기다려서 끝내는 것을 측정하는 분별 계측법을 독창적으로 개발했다.
첨단 기술인 단일분자 실험을 전담한 서울대 물리천문학부 송은호 박사과정 학생이 제1 저자로 참여한 논문(제목: Rho-dependent transcription termination proceeds via three routes)이 저명 국제학술지 네이처 커뮤니케이션스(Nature Communications)에 올해 3월 29일 字 게재됐다. 서울대 엄희수 박사, 우리 대학 팔린다 무나싱하 박사, 우리 대학 황승하 대학원생도 참여하여 저자가 총 8명이다.
이번 공동 연구에 단일분자 형광 기술을 구사하는 물리학자, 유전자 발현이라는 기본적 생명현상을 탐구하는 생명과학자, 중합효소나 종결인자와 같은 단백질의 구조를 규명하는 화학자가 두루 참가했다. 다양한 분야의 전공자가 협업한 다학제 기초연구의 우수 사례이며, 한국연구재단 중견연구자지원사업, KAIST 고위험‧고성과 연구사업 등의 지원을 받았다.
송은호 제1 저자는 "어느 학설이 맞는지를 검증하기 위해, DNA의 한 곳에서 종결하는 종결자의 경우 작동이 단순명료하리라 짐작하고 첫 실험 대상으로 삼았는데, 뜻밖에 여러 작동원리가 모두 관찰됐고 세 갈래로 복잡했다”라며 “여러 곳에서 종결하는 다른 종결자 DNA의 경우 오히려 세 갈래가 다 보이지 않기도 해 첫 선택에 운이 따랐던 것 같다”고 말했다.
2022.03.30 조회수 10760 -
 생각만으로 정확하게 로봇팔 조종이 가능한 뇌-기계 인터페이스 개발
우리 대학 바이오및뇌공학과 정재승 교수 연구팀이 3차원 공간상에서 생각만으로 로봇팔을 높은 정확도 (90.9~92.6%)로 조종하는 `뇌-기계 인터페이스 시스템'을 개발했다고 23일 밝혔다.
정 교수 연구팀은 인공지능과 유전자 알고리즘을 사용해 인간의 대뇌 심부에서 측정한 뇌파만으로 팔 움직임의 의도를 파악해 로봇팔을 제어하는 새로운 형태의 뇌-기계 인터페이스 시스템을 개발했다. 뇌 활동만으로 사람의 의도를 파악해 로봇이나 기계가 대신 행동에 옮기는 `뇌-기계 인터페이스' 기술은 최근 급속도로 발전하고 있다. 하지만 손을 움직이는 정도의 의도 파악을 넘어, 팔 움직임의 방향에 대한 의도를 섬세하게 파악해 정교하게 로봇팔을 움직이는 기술은 아직 정확도가 높지 않았다.
하지만 연구팀은 이번 연구에서 조종 `방향'에 대한 의도를 뇌 활동만으로 인식하는 인공지능 모델을 개발했고, 그 결과 3차원 공간상에서 24개의 방향을 90% 이상의 정확도로 정교하게 해석하는 시스템을 개발했다.
게다가 딥러닝 등 기존 기계학습 기술은 높은 사양의 GPU 하드웨어가 필요했지만, 이번 연구에서는 축적 컴퓨팅(Reservoir Computing) 기법을 이용해 낮은 사양의 하드웨어에서도 인공지능 학습이 가능하여 스마트 모바일 기기에서도 폭넓게 응용될 수 있도록 개발해, 향후 메타버스와 스마트 기기에도 폭넓게 적용이 가능할 것으로 기대된다.
우리 대학 김훈희 박사(現 강남대 조교수)가 제1 저자로 참여한 이번 연구는 국제학술지 `어플라이드 소프트 컴퓨팅(Applied Soft Computing)' 2022년 117권 3월호에 출판됐다. (논문명 : An electrocorticographic decoder for arm movement for brain-machine interface using an echo state network and Gaussian readout).
뇌-기계 인터페이스는 사용자의 뇌 활동을 통해 의도를 읽고 로봇이나 기계에 전달하는 기술로서 로봇, 드론, 컴퓨터뿐만 아니라 스마트 모바일 기기, 메타버스 등에서의 이용될 차세대 인터페이스 기술로 각광받고 있다.
특히 기존의 인터페이스가 외부 신체 기관을 통해 명령을 간접 전달(버튼, 터치, 제스처 등)해야 하지만 뇌-기계 인터페이스는 명령을 뇌로부터 직접적 전달한다는 점에서 가장 진보된 인터페이스 기술로 여겨진다.
그러나 뇌파는 개개인의 차이가 매우 크고, 단일 신경 세포로부터 정확한 신호를 읽는 것이 아니라 넓은 영역에 있는 신경 세포 집단의 전기적 신호 특성을 해석해야 하므로 잡음이 크다는 한계점을 가지고 있다.
연구팀은 이러한 문제 해결을 위해 최첨단 인공지능 기법의 하나인 `축적 컴퓨팅 기법'을 이용해 뇌-기계 인터페이스에서 필요한 개개인의 뇌파 신호의 중요 특성을 인공신경망이 자동으로 학습해 찾을 수 있도록 구현했다.
또한 유전자 알고리즘(Genetic Algorithm)을 이용해 인공지능 신경망이 최적의 뇌파 특성을 효율적으로 찾을 수 있게 시스템을 설계했다. 연구팀은 심부 뇌파를 최종 해석하는 리드아웃(Readout)을 가우시안(Gaussian) 모델로 설계해 시각피질 신경 세포가 방향을 표현하는 방법을 모방하는 인공신경망을 개발했다. 이런 리드아웃 방식은 축적 컴퓨팅의 선형 학습 알고리즘을 이용해 일반적 사양의 간단한 하드웨어에서도 빠르게 학습할 수 있어 메타버스, 스마트기기 등 일상생활에서 응용이 가능해진다.
특히, 이번 연구에서 만들어진 뇌-기계 인터페이스 인공지능 모델은 3차원상에서 24가지 방향 즉, 각 차원에서 8가지 방향을 디코딩할 수 있으며 모든 방향에서 평균 90% 이상의 정확도 (90.9%~92.6% 범위)를 보였다. 또한 연구된 뇌-기계 인터페이스는 3차원 공간상에서 로봇팔을 움직이는 상상을 할 때의 뇌파를 해석해 성공적으로 로봇팔을 움직이는 시뮬레이션 결과를 보였다.
인공지능 시스템을 만든 제1 저자인 김훈희 박사는 "공학적인 신호처리 기법에 의존해 온 기존 뇌파 디코딩 방법과는 달리, 인간 뇌의 실제 작동 구조를 모방한 인공신경망을 개발해 좀더 발전된 형태의 뇌-기계 인터페이스 시스템을 개발해 기쁘다ˮ면서 "향후 뇌의 특성을 좀 더 구체적으로 이용한 `뇌 모방 인공지능(Brain-inspired A.I.)'을 이용한 다양한 뇌-기계 인터페이스를 개발할 계획이다ˮ라고 말했다.
이번 연구를 주도한 연구책임자 정재승 교수는 "뇌파를 통해 생각만으로 로봇팔을 구동하는 `뇌-기계 인터페이스 시스템'들이 대부분 고사양 하드웨어가 필요해 실시간 응용으로 나아가기 어렵고 스마트기기 등으로 적용이 어려웠다. 그러나 이번 시스템은 90%~92%의 높은 정확도를 가진 의도 인식 인공지능 시스템을 만들어 메타버스 안에서 아바타를 생각대로 움직이게 하거나 앱을 생각만으로 컨트롤하는 스마트기기 등에 광범위하게 사용될 수 있다ˮ고 말했다.
이번 연구 결과는 사지마비 환자나 사고로 팔을 잃은 환자들을 위한 로봇팔 장착 및 제어 기술부터, 메타버스, 스마트기기, 게임, 엔터테인먼트 애플리케이션 등 다양한 시스템에 뇌-기계 인터페이스를 적용할 가능성을 열어 줄 것으로 기대된다.
이번 연구는 한국연구재단 뇌 원천기술개발사업의 지원을 받아 수행됐다.
2022.02.24 조회수 16127
생각만으로 정확하게 로봇팔 조종이 가능한 뇌-기계 인터페이스 개발
우리 대학 바이오및뇌공학과 정재승 교수 연구팀이 3차원 공간상에서 생각만으로 로봇팔을 높은 정확도 (90.9~92.6%)로 조종하는 `뇌-기계 인터페이스 시스템'을 개발했다고 23일 밝혔다.
정 교수 연구팀은 인공지능과 유전자 알고리즘을 사용해 인간의 대뇌 심부에서 측정한 뇌파만으로 팔 움직임의 의도를 파악해 로봇팔을 제어하는 새로운 형태의 뇌-기계 인터페이스 시스템을 개발했다. 뇌 활동만으로 사람의 의도를 파악해 로봇이나 기계가 대신 행동에 옮기는 `뇌-기계 인터페이스' 기술은 최근 급속도로 발전하고 있다. 하지만 손을 움직이는 정도의 의도 파악을 넘어, 팔 움직임의 방향에 대한 의도를 섬세하게 파악해 정교하게 로봇팔을 움직이는 기술은 아직 정확도가 높지 않았다.
하지만 연구팀은 이번 연구에서 조종 `방향'에 대한 의도를 뇌 활동만으로 인식하는 인공지능 모델을 개발했고, 그 결과 3차원 공간상에서 24개의 방향을 90% 이상의 정확도로 정교하게 해석하는 시스템을 개발했다.
게다가 딥러닝 등 기존 기계학습 기술은 높은 사양의 GPU 하드웨어가 필요했지만, 이번 연구에서는 축적 컴퓨팅(Reservoir Computing) 기법을 이용해 낮은 사양의 하드웨어에서도 인공지능 학습이 가능하여 스마트 모바일 기기에서도 폭넓게 응용될 수 있도록 개발해, 향후 메타버스와 스마트 기기에도 폭넓게 적용이 가능할 것으로 기대된다.
우리 대학 김훈희 박사(現 강남대 조교수)가 제1 저자로 참여한 이번 연구는 국제학술지 `어플라이드 소프트 컴퓨팅(Applied Soft Computing)' 2022년 117권 3월호에 출판됐다. (논문명 : An electrocorticographic decoder for arm movement for brain-machine interface using an echo state network and Gaussian readout).
뇌-기계 인터페이스는 사용자의 뇌 활동을 통해 의도를 읽고 로봇이나 기계에 전달하는 기술로서 로봇, 드론, 컴퓨터뿐만 아니라 스마트 모바일 기기, 메타버스 등에서의 이용될 차세대 인터페이스 기술로 각광받고 있다.
특히 기존의 인터페이스가 외부 신체 기관을 통해 명령을 간접 전달(버튼, 터치, 제스처 등)해야 하지만 뇌-기계 인터페이스는 명령을 뇌로부터 직접적 전달한다는 점에서 가장 진보된 인터페이스 기술로 여겨진다.
그러나 뇌파는 개개인의 차이가 매우 크고, 단일 신경 세포로부터 정확한 신호를 읽는 것이 아니라 넓은 영역에 있는 신경 세포 집단의 전기적 신호 특성을 해석해야 하므로 잡음이 크다는 한계점을 가지고 있다.
연구팀은 이러한 문제 해결을 위해 최첨단 인공지능 기법의 하나인 `축적 컴퓨팅 기법'을 이용해 뇌-기계 인터페이스에서 필요한 개개인의 뇌파 신호의 중요 특성을 인공신경망이 자동으로 학습해 찾을 수 있도록 구현했다.
또한 유전자 알고리즘(Genetic Algorithm)을 이용해 인공지능 신경망이 최적의 뇌파 특성을 효율적으로 찾을 수 있게 시스템을 설계했다. 연구팀은 심부 뇌파를 최종 해석하는 리드아웃(Readout)을 가우시안(Gaussian) 모델로 설계해 시각피질 신경 세포가 방향을 표현하는 방법을 모방하는 인공신경망을 개발했다. 이런 리드아웃 방식은 축적 컴퓨팅의 선형 학습 알고리즘을 이용해 일반적 사양의 간단한 하드웨어에서도 빠르게 학습할 수 있어 메타버스, 스마트기기 등 일상생활에서 응용이 가능해진다.
특히, 이번 연구에서 만들어진 뇌-기계 인터페이스 인공지능 모델은 3차원상에서 24가지 방향 즉, 각 차원에서 8가지 방향을 디코딩할 수 있으며 모든 방향에서 평균 90% 이상의 정확도 (90.9%~92.6% 범위)를 보였다. 또한 연구된 뇌-기계 인터페이스는 3차원 공간상에서 로봇팔을 움직이는 상상을 할 때의 뇌파를 해석해 성공적으로 로봇팔을 움직이는 시뮬레이션 결과를 보였다.
인공지능 시스템을 만든 제1 저자인 김훈희 박사는 "공학적인 신호처리 기법에 의존해 온 기존 뇌파 디코딩 방법과는 달리, 인간 뇌의 실제 작동 구조를 모방한 인공신경망을 개발해 좀더 발전된 형태의 뇌-기계 인터페이스 시스템을 개발해 기쁘다ˮ면서 "향후 뇌의 특성을 좀 더 구체적으로 이용한 `뇌 모방 인공지능(Brain-inspired A.I.)'을 이용한 다양한 뇌-기계 인터페이스를 개발할 계획이다ˮ라고 말했다.
이번 연구를 주도한 연구책임자 정재승 교수는 "뇌파를 통해 생각만으로 로봇팔을 구동하는 `뇌-기계 인터페이스 시스템'들이 대부분 고사양 하드웨어가 필요해 실시간 응용으로 나아가기 어렵고 스마트기기 등으로 적용이 어려웠다. 그러나 이번 시스템은 90%~92%의 높은 정확도를 가진 의도 인식 인공지능 시스템을 만들어 메타버스 안에서 아바타를 생각대로 움직이게 하거나 앱을 생각만으로 컨트롤하는 스마트기기 등에 광범위하게 사용될 수 있다ˮ고 말했다.
이번 연구 결과는 사지마비 환자나 사고로 팔을 잃은 환자들을 위한 로봇팔 장착 및 제어 기술부터, 메타버스, 스마트기기, 게임, 엔터테인먼트 애플리케이션 등 다양한 시스템에 뇌-기계 인터페이스를 적용할 가능성을 열어 줄 것으로 기대된다.
이번 연구는 한국연구재단 뇌 원천기술개발사업의 지원을 받아 수행됐다.
2022.02.24 조회수 16127 -
 단일세포 RNA 시퀀싱을 통한 꽃향기 합성 유전자 발굴
우리 대학 생명과학과 김상규 교수 연구팀이 꽃향기 합성 유전자를 발굴하기 위해 꽃잎 단일세포 RNA 시퀀싱 기술을 개발하고 벤질아세톤(benzylacetone) 꽃향기 합성 경로를 밝혔다.
벤질아세톤은 코요테담배(Nicotiana attenuata) 꽃에서 합성되고 밤에 분비가 되는 향기 물질이다. 이 향기물질은 밤에 활동하는 박각시나방을 유인한다. 그리고 꽃은 꿀을 제공하고 그 대가로 나방은 화분pollen을 멀리 날라준다. 또한 벤질아세톤은 코요테담배 꽃을 먹는 해충을 쫓아내는 기능을 하고 있다. 생태적으로 재미있는 기능을 하고 있는 물질이지만 생합성 경로에 대해서는 완전히 알려진 상태가 아니었다.
일반적으로 식물이 만들어내는 대사물질의 생합성 유전자를 밝히기 위해 사용하는 방법의 단점을 극복하기 위해서 꽃잎 단일세포에서 발현되는 유전자의 연관도를 이용하여 물질대사 경로를 밝힐 수 있다는 것을 이번 연구를 통해서 증명하였다. 특히 유전정보가 제한적으로 알려져 있고 다양한 생태형 ecotype을 가진 식물 집단이 없어도 비모델 식물에서 물질합성 경로에 있는 효소와 그 효소의 발현을 조절하는 전사인자 등도 찾을 수 있는 가능성을 제시하였다.
단일세포 RNA 시퀀싱의 장점을 활용하여 향기합성 유전자가 만들어지는 세포를 구별하고 꽃잎에서 향기가 합성되는 위치도 밝힐 수 있었다.
우리 대학 생명과학과 강문영 석박사통합과정 학생이 제1 저자로 참여한 이번 연구 결과는 'New Phytologist' 학술지에 게재됐다. (관련 논문명: Single-cell RNA-sequencing of Nicotiana attenuata corolla cells reveals the biosynthetic pathway of a floral scent)
한편 이번 연구는 삼성미래기술육성사업과 포스코사이언스펠로십 지원을 받아 수행됐다.
2022.02.14 조회수 8753
단일세포 RNA 시퀀싱을 통한 꽃향기 합성 유전자 발굴
우리 대학 생명과학과 김상규 교수 연구팀이 꽃향기 합성 유전자를 발굴하기 위해 꽃잎 단일세포 RNA 시퀀싱 기술을 개발하고 벤질아세톤(benzylacetone) 꽃향기 합성 경로를 밝혔다.
벤질아세톤은 코요테담배(Nicotiana attenuata) 꽃에서 합성되고 밤에 분비가 되는 향기 물질이다. 이 향기물질은 밤에 활동하는 박각시나방을 유인한다. 그리고 꽃은 꿀을 제공하고 그 대가로 나방은 화분pollen을 멀리 날라준다. 또한 벤질아세톤은 코요테담배 꽃을 먹는 해충을 쫓아내는 기능을 하고 있다. 생태적으로 재미있는 기능을 하고 있는 물질이지만 생합성 경로에 대해서는 완전히 알려진 상태가 아니었다.
일반적으로 식물이 만들어내는 대사물질의 생합성 유전자를 밝히기 위해 사용하는 방법의 단점을 극복하기 위해서 꽃잎 단일세포에서 발현되는 유전자의 연관도를 이용하여 물질대사 경로를 밝힐 수 있다는 것을 이번 연구를 통해서 증명하였다. 특히 유전정보가 제한적으로 알려져 있고 다양한 생태형 ecotype을 가진 식물 집단이 없어도 비모델 식물에서 물질합성 경로에 있는 효소와 그 효소의 발현을 조절하는 전사인자 등도 찾을 수 있는 가능성을 제시하였다.
단일세포 RNA 시퀀싱의 장점을 활용하여 향기합성 유전자가 만들어지는 세포를 구별하고 꽃잎에서 향기가 합성되는 위치도 밝힐 수 있었다.
우리 대학 생명과학과 강문영 석박사통합과정 학생이 제1 저자로 참여한 이번 연구 결과는 'New Phytologist' 학술지에 게재됐다. (관련 논문명: Single-cell RNA-sequencing of Nicotiana attenuata corolla cells reveals the biosynthetic pathway of a floral scent)
한편 이번 연구는 삼성미래기술육성사업과 포스코사이언스펠로십 지원을 받아 수행됐다.
2022.02.14 조회수 8753 -
 유전자 가위로 생체 내 정밀한 유전자 교정에 의한 면역 항암 치료
CRISPR/Cas9 시스템을 이용하여 유전자교정을 일으킴으로써 암의 면역 치료를 유도하는 기술이 우리 대학 연구진에 의해 개발됐다.
우리 대학 생명과학과 정현정 교수, 서울대학교 의과학과 정기훈 교수 공동연구팀이 CRISPR/Cas9 리보핵산단백질을 생체 내에 효과적으로 전달하는 나노복합체를 개발하여 면역 관문 유전자를 교정함으로써 항암 효과를 보이는데 성공했다고 밝혔다.
암은 현대인의 건강을 위협하는 대표적인 요인으로 꼽히고 있다. 암의 치료 방법 중 면역 항암 요법은 부작용이 적고 높은 치료 성적을 보여 다양한 암 유형에 적용할 수 있다. 기존에는 항체 기반 치료법이 주로 임상에서 사용되고 있으며 다양한 고형암의 치료에 승인되었으나, 일시적인 효과로 반복 투여가 필요하다.
CRISPR/Cas9 시스템은 유전체의 서열을 직접 정밀하게 교정할 수 있으며, DNA 이중가닥을 절단하는 Cas9 제한효소와 특정 서열을 표적하는 단일 가이드 RNA로 이루어진다. 유전자교정 치료제의 경우 일반적으로 바이러스 기반 치료 방법을 이용했으나 돌연변이 유발, 비특이적 표적 효과 등으로 인해 한계가 있다. 비바이러스 치료제로 Cas9 단백질 및 단일가닥 RNA를 이용하면 바이러스 치료보다 안전성을 높일 수 있으나 낮은 세포내 전달 효과로 치료 효능이 떨어진다. 전달 효율을 높이기 위해 기존에 다양한 방법이 개발되고 연구됐으나, 일반적으로 과량의 전달체물질을 사용함으로써 생체 내 독성 문제가 나타나는 한계점이 있다. 이러한 문제점을 개선하기 위해 연구팀은 Cas9 단백질에 세포내 유입을 촉진하는 고분자를 접합시켜 극미량의 전달체물질로 고효율 전달이 가능한 Cas9 컨쥬게이트를 제작하여 활용했다.
연구팀은 이러한 Cas9 컨쥬게이트, 단일 가이드 RNA 및 변형된 데옥시뉴클레오타이드(DNA)를 추가해 나노조립된 리보핵단백질 복합체(이하 NanoRNP)를 개발했다. 이 복합체는 Cas9 컨쥬게이트, RNA 및 DNA의 상호작용으로 쉽게 제작할 수 있고, 유전자 교정 치료제로써 단일 요법에 의해 항암 치료가 가능하다는 점이 장점이다.
우리 대학 생명과학과 석박사통합과정 이주희 학생이 제1 저자로 참여한 이번 연구 결과는 재료화학 분야 국제학술지 `케미스트리 오브 머티리얼즈(Chemistry of Materials)'에 12월 20일 字 온라인 게재됐다. (논문명 : Nano-assembly of a Chemically Tailored Cas9 Ribonucleoprotein for In Vivo Gene Editing and Cancer Immunotherapy)
NanoRNP의 경우 Cas9에 부착된 고분자가 강한 양이온성을 지녀 단일 가이드 RNA와 안정적으로 복합체를 형성시키며, 생체내 분해효소로부터 보호하여 활성을 향상시킨다. 본 연구팀은 NanoRNP를 피부암에서 많이 발현되는 프로그램된 세포사멸 리간드-1 (PD-L1) 유전자를 표적하는데 응용하였다. PD-L1은 면역 세포의 표면 수용체에 존재하는 프로그램된 세포사멸 수용체-1 (PD-1)과 상호작용하여 면역 세포의 반응을 억제해 암세포의 세포사멸 회피를 유도한다.
연구팀은 NanoRNP를 이용하여 PD-L1 유전자의 교정으로 유전자결손을 유도하여, 면역 세포들이 활성화되고 종양미세환경의 변화로 면역 세포에 의한 암세포 사멸이 유도됨을 확인했다.
연구팀은 이번 연구 결과를 응용해 향후 암 뿐만 아니라 유전 질환 등 다양한 질병에 적용함으로써 연구를 확대 및 발전시켜 나갈 수 있을 것으로 기대하고 있다.
한편 이번 연구는 한국연구재단 중견연구자지원사업, 범부처전주기의료기기연구개발사업 및 KAIST End Run 사업의 지원을 통해 이뤄졌다.
2021.12.24 조회수 12445
유전자 가위로 생체 내 정밀한 유전자 교정에 의한 면역 항암 치료
CRISPR/Cas9 시스템을 이용하여 유전자교정을 일으킴으로써 암의 면역 치료를 유도하는 기술이 우리 대학 연구진에 의해 개발됐다.
우리 대학 생명과학과 정현정 교수, 서울대학교 의과학과 정기훈 교수 공동연구팀이 CRISPR/Cas9 리보핵산단백질을 생체 내에 효과적으로 전달하는 나노복합체를 개발하여 면역 관문 유전자를 교정함으로써 항암 효과를 보이는데 성공했다고 밝혔다.
암은 현대인의 건강을 위협하는 대표적인 요인으로 꼽히고 있다. 암의 치료 방법 중 면역 항암 요법은 부작용이 적고 높은 치료 성적을 보여 다양한 암 유형에 적용할 수 있다. 기존에는 항체 기반 치료법이 주로 임상에서 사용되고 있으며 다양한 고형암의 치료에 승인되었으나, 일시적인 효과로 반복 투여가 필요하다.
CRISPR/Cas9 시스템은 유전체의 서열을 직접 정밀하게 교정할 수 있으며, DNA 이중가닥을 절단하는 Cas9 제한효소와 특정 서열을 표적하는 단일 가이드 RNA로 이루어진다. 유전자교정 치료제의 경우 일반적으로 바이러스 기반 치료 방법을 이용했으나 돌연변이 유발, 비특이적 표적 효과 등으로 인해 한계가 있다. 비바이러스 치료제로 Cas9 단백질 및 단일가닥 RNA를 이용하면 바이러스 치료보다 안전성을 높일 수 있으나 낮은 세포내 전달 효과로 치료 효능이 떨어진다. 전달 효율을 높이기 위해 기존에 다양한 방법이 개발되고 연구됐으나, 일반적으로 과량의 전달체물질을 사용함으로써 생체 내 독성 문제가 나타나는 한계점이 있다. 이러한 문제점을 개선하기 위해 연구팀은 Cas9 단백질에 세포내 유입을 촉진하는 고분자를 접합시켜 극미량의 전달체물질로 고효율 전달이 가능한 Cas9 컨쥬게이트를 제작하여 활용했다.
연구팀은 이러한 Cas9 컨쥬게이트, 단일 가이드 RNA 및 변형된 데옥시뉴클레오타이드(DNA)를 추가해 나노조립된 리보핵단백질 복합체(이하 NanoRNP)를 개발했다. 이 복합체는 Cas9 컨쥬게이트, RNA 및 DNA의 상호작용으로 쉽게 제작할 수 있고, 유전자 교정 치료제로써 단일 요법에 의해 항암 치료가 가능하다는 점이 장점이다.
우리 대학 생명과학과 석박사통합과정 이주희 학생이 제1 저자로 참여한 이번 연구 결과는 재료화학 분야 국제학술지 `케미스트리 오브 머티리얼즈(Chemistry of Materials)'에 12월 20일 字 온라인 게재됐다. (논문명 : Nano-assembly of a Chemically Tailored Cas9 Ribonucleoprotein for In Vivo Gene Editing and Cancer Immunotherapy)
NanoRNP의 경우 Cas9에 부착된 고분자가 강한 양이온성을 지녀 단일 가이드 RNA와 안정적으로 복합체를 형성시키며, 생체내 분해효소로부터 보호하여 활성을 향상시킨다. 본 연구팀은 NanoRNP를 피부암에서 많이 발현되는 프로그램된 세포사멸 리간드-1 (PD-L1) 유전자를 표적하는데 응용하였다. PD-L1은 면역 세포의 표면 수용체에 존재하는 프로그램된 세포사멸 수용체-1 (PD-1)과 상호작용하여 면역 세포의 반응을 억제해 암세포의 세포사멸 회피를 유도한다.
연구팀은 NanoRNP를 이용하여 PD-L1 유전자의 교정으로 유전자결손을 유도하여, 면역 세포들이 활성화되고 종양미세환경의 변화로 면역 세포에 의한 암세포 사멸이 유도됨을 확인했다.
연구팀은 이번 연구 결과를 응용해 향후 암 뿐만 아니라 유전 질환 등 다양한 질병에 적용함으로써 연구를 확대 및 발전시켜 나갈 수 있을 것으로 기대하고 있다.
한편 이번 연구는 한국연구재단 중견연구자지원사업, 범부처전주기의료기기연구개발사업 및 KAIST End Run 사업의 지원을 통해 이뤄졌다.
2021.12.24 조회수 12445 -
 악성 유방암 세포를 치료 가능한 상태로 되돌리는 암세포 리프로그래밍 기술 개발
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 시스템생물학 연구를 통해 악성 유방암세포를 치료 가능한 상태로 되돌리는 암세포 리프로그래밍 기술을 개발하는 데 성공했다고 30일 밝혔다.
연구팀은 유방암 아류 중에서 가장 악성으로 알려진 삼중음성 유방암(basal-like 혹은 triple negative) 환자들과 호르몬 치료가 가능한 루미날-A 유방암(luminal-A) 환자들의 유전자 네트워크를 컴퓨터시뮬레이션을 통해 분석함으로써 삼중음성 유방암세포를 루미날-A 유방암세포로 변환하는데 필요한 핵심 인자를 규명했다. 그리고 이를 조절해 삼중음성 유방암세포를 루미날-A 유방암세포로 리프로그래밍한 뒤 호르몬 치료를 시행하는 새로운 치료 원리를 개발했다.
우리 대학 최새롬 박사과정, 황채영 박사, 이종훈 박사과정 등이 참여한 이번 연구결과는 미국암학회(AACR)에서 출간하는 국제저널 `캔서 리서치 (Cancer Research)' 11월 30일 字 논문으로 출판됐다. (논문명: Network analysis identifies regulators of basal-like breast cancer reprogramming and endocrine therapy vulnerability)
현재 삼중음성 유방암 환자들에게 적용되는 항암 화학요법은 빠르게 분열해 전이를 일으키는 암세포를 공격해 죽임으로써 암세포의 증식을 억제하는 방식이다. 하지만 이러한 치료는 신체 내 정상적으로 분열하고 있는 세포들까지도 함께 사멸시켜 구토, 설사, 탈모, 골수 기능장애, 무기력 등의 심각한 부작용을 일으킨다. 또한 삼중음성 유방암세포들은 이와 같은 독성항암제에 처음부터 내성을 갖거나 새로운 내성을 획득하면서 결국 약물에 높은 저항성을 가지는 암세포로 진화하게 된다. 따라서 삼중음성 유방암에 대한 현재의 항암치료는 내성을 갖는 암세포를 없애기 위해 더 많은 정상세포의 사멸을 감수해야만 하는 큰 한계를 지니고 있다.
이를 극복하기 위해 암세포만을 특이적으로 공격하는 표적 항암요법과 우리 몸의 면역시스템을 활용한 면역 항암요법이 주목을 받고 있으나 각각 효과와 적용대상이 매우 제한적이며 장기치료 시 여전히 내성 발생의 문제가 보고되고 있다. 이처럼 현재 개발된 항암요법들은 암세포를 죽이려는 공통적인 원리 때문에 근본적인 한계를 가진다.
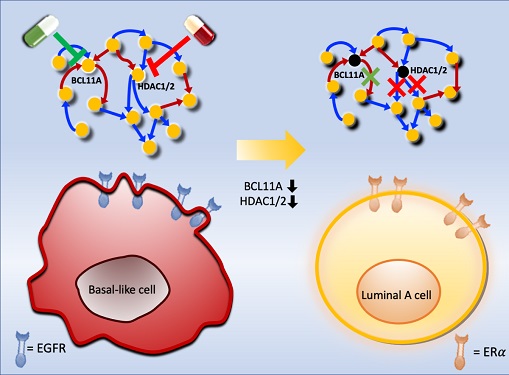
조 교수 연구팀은 시스템생물학 연구기법을 통해 악성 유방암세포인 삼중음성 유방암세포를 호르몬 치료가 가능한 루미날-A 유방암세포로 변환시킨 뒤 치료하는 새로운 개념의 치료전략을 개발했다. 이를 위해 유전자 네트워크의 수학모델을 개발하고 대규모 컴퓨터시뮬레이션 분석과 복잡계 네트워크 제어기술을 적용한 결과 두 개의 핵심 분자 타겟인 `BCL11A'와 `HDAC1/2'를 발굴했다.
조 교수 연구팀은 BCL11A와 HDAC1/2를 억제함으로써 삼중음성 유방암세포를 효과적으로 루미날-A 유방암세포로 변환시킬 수 있음을 분자 세포실험을 통해 증명했다. 삼중음성 유방암세포에서 이 핵심 인자들을 억제했을 때 세포의 분열이 감소하고, 삼중음성 유방암세포의 주요 세포성장 신호 흐름 경로인 `EGFR'과 관련된 인자들의 활동이 감소했으며, 루미날-A 유방암세포의 주요 세포성장 신호흐름 경로인 `ERa' 신호전달 경로 인자들의 활성이 회복되는 것을 확인했다.
이번 연구에서 발굴된 분자 타겟 중 BCL11A 단백질의 활성을 억제할 수 있는 저분자화합물은 아직 개발된 바 없으며 추후 신약개발과 임상실험을 통해 악성 유방암세포를 치료 가능한 세포상태로 리프로그래밍 함으로써 안전하고 효과적으로 치료하는 새로운 치료기술이 실현될 수 있을 것으로 보인다. 특히 이처럼 암세포의 성질을 되돌리거나 변환하는 암세포 리프로그래밍 기반의 새로운 치료전략이 임상에서 실현된다면 현재 항암치료의 많은 부작용과 내성 발생을 근본적으로 해결함으로써 암 환자의 고통을 최소화하고 삶의 질을 크게 향상시킬 수 있을 것으로 기대된다.
조 교수는 "그동안 유방암 중에서도 가장 악성인 삼중음성 유방암은 독성이 강해 큰 부작용을 일으키는 화학 항암치료 외에는 방법이 없었으나 이를 호르몬 치료가 가능하며 덜 악성인 루미날-A 유방암세포로 리프로그래밍해 효과적으로 치료할 수 있는 새로운 가능성을 열었다ˮ라며 "이번 연구는 악성 암세포를 직접 없애려고 하기보다 치료가 수월한 세포 상태로 되돌린 뒤 치료하는 새로운 방식의 항암 치료전략을 제시했다ˮ라고 말했다.
조 교수 연구팀은 2020년 1월에 대장암세포를 정상 대장 세포로 되돌리는 연구에 성공한 바 있으며, 이번 연구 결과는 암세포 리프로그래밍을 통한 가역화 기술 개발의 두 번째 성과다.
이번 연구는 과학기술정보통신부와 한국연구재단의 중견연구자지원사업과 한국전자통신연구소 공동연구사업, KAIST Grand Challenge 30의 지원으로 수행됐다.
2021.11.30 조회수 12689
악성 유방암 세포를 치료 가능한 상태로 되돌리는 암세포 리프로그래밍 기술 개발
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 시스템생물학 연구를 통해 악성 유방암세포를 치료 가능한 상태로 되돌리는 암세포 리프로그래밍 기술을 개발하는 데 성공했다고 30일 밝혔다.
연구팀은 유방암 아류 중에서 가장 악성으로 알려진 삼중음성 유방암(basal-like 혹은 triple negative) 환자들과 호르몬 치료가 가능한 루미날-A 유방암(luminal-A) 환자들의 유전자 네트워크를 컴퓨터시뮬레이션을 통해 분석함으로써 삼중음성 유방암세포를 루미날-A 유방암세포로 변환하는데 필요한 핵심 인자를 규명했다. 그리고 이를 조절해 삼중음성 유방암세포를 루미날-A 유방암세포로 리프로그래밍한 뒤 호르몬 치료를 시행하는 새로운 치료 원리를 개발했다.
우리 대학 최새롬 박사과정, 황채영 박사, 이종훈 박사과정 등이 참여한 이번 연구결과는 미국암학회(AACR)에서 출간하는 국제저널 `캔서 리서치 (Cancer Research)' 11월 30일 字 논문으로 출판됐다. (논문명: Network analysis identifies regulators of basal-like breast cancer reprogramming and endocrine therapy vulnerability)
현재 삼중음성 유방암 환자들에게 적용되는 항암 화학요법은 빠르게 분열해 전이를 일으키는 암세포를 공격해 죽임으로써 암세포의 증식을 억제하는 방식이다. 하지만 이러한 치료는 신체 내 정상적으로 분열하고 있는 세포들까지도 함께 사멸시켜 구토, 설사, 탈모, 골수 기능장애, 무기력 등의 심각한 부작용을 일으킨다. 또한 삼중음성 유방암세포들은 이와 같은 독성항암제에 처음부터 내성을 갖거나 새로운 내성을 획득하면서 결국 약물에 높은 저항성을 가지는 암세포로 진화하게 된다. 따라서 삼중음성 유방암에 대한 현재의 항암치료는 내성을 갖는 암세포를 없애기 위해 더 많은 정상세포의 사멸을 감수해야만 하는 큰 한계를 지니고 있다.
이를 극복하기 위해 암세포만을 특이적으로 공격하는 표적 항암요법과 우리 몸의 면역시스템을 활용한 면역 항암요법이 주목을 받고 있으나 각각 효과와 적용대상이 매우 제한적이며 장기치료 시 여전히 내성 발생의 문제가 보고되고 있다. 이처럼 현재 개발된 항암요법들은 암세포를 죽이려는 공통적인 원리 때문에 근본적인 한계를 가진다.
조 교수 연구팀은 시스템생물학 연구기법을 통해 악성 유방암세포인 삼중음성 유방암세포를 호르몬 치료가 가능한 루미날-A 유방암세포로 변환시킨 뒤 치료하는 새로운 개념의 치료전략을 개발했다. 이를 위해 유전자 네트워크의 수학모델을 개발하고 대규모 컴퓨터시뮬레이션 분석과 복잡계 네트워크 제어기술을 적용한 결과 두 개의 핵심 분자 타겟인 `BCL11A'와 `HDAC1/2'를 발굴했다.
조 교수 연구팀은 BCL11A와 HDAC1/2를 억제함으로써 삼중음성 유방암세포를 효과적으로 루미날-A 유방암세포로 변환시킬 수 있음을 분자 세포실험을 통해 증명했다. 삼중음성 유방암세포에서 이 핵심 인자들을 억제했을 때 세포의 분열이 감소하고, 삼중음성 유방암세포의 주요 세포성장 신호 흐름 경로인 `EGFR'과 관련된 인자들의 활동이 감소했으며, 루미날-A 유방암세포의 주요 세포성장 신호흐름 경로인 `ERa' 신호전달 경로 인자들의 활성이 회복되는 것을 확인했다.
이번 연구에서 발굴된 분자 타겟 중 BCL11A 단백질의 활성을 억제할 수 있는 저분자화합물은 아직 개발된 바 없으며 추후 신약개발과 임상실험을 통해 악성 유방암세포를 치료 가능한 세포상태로 리프로그래밍 함으로써 안전하고 효과적으로 치료하는 새로운 치료기술이 실현될 수 있을 것으로 보인다. 특히 이처럼 암세포의 성질을 되돌리거나 변환하는 암세포 리프로그래밍 기반의 새로운 치료전략이 임상에서 실현된다면 현재 항암치료의 많은 부작용과 내성 발생을 근본적으로 해결함으로써 암 환자의 고통을 최소화하고 삶의 질을 크게 향상시킬 수 있을 것으로 기대된다.
조 교수는 "그동안 유방암 중에서도 가장 악성인 삼중음성 유방암은 독성이 강해 큰 부작용을 일으키는 화학 항암치료 외에는 방법이 없었으나 이를 호르몬 치료가 가능하며 덜 악성인 루미날-A 유방암세포로 리프로그래밍해 효과적으로 치료할 수 있는 새로운 가능성을 열었다ˮ라며 "이번 연구는 악성 암세포를 직접 없애려고 하기보다 치료가 수월한 세포 상태로 되돌린 뒤 치료하는 새로운 방식의 항암 치료전략을 제시했다ˮ라고 말했다.
조 교수 연구팀은 2020년 1월에 대장암세포를 정상 대장 세포로 되돌리는 연구에 성공한 바 있으며, 이번 연구 결과는 암세포 리프로그래밍을 통한 가역화 기술 개발의 두 번째 성과다.
이번 연구는 과학기술정보통신부와 한국연구재단의 중견연구자지원사업과 한국전자통신연구소 공동연구사업, KAIST Grand Challenge 30의 지원으로 수행됐다.
2021.11.30 조회수 12689