%EC%A0%95%EC%9D%B8%EA%B2%BD
-
 인공지능 기반 대장암 3차원 게놈 지도 최초 해독
세계 최초로 예전에 비해 최대 규모로 한국인 대장암 환자 3차원 게놈 지도를 작성하여 화제다.
우리 대학 생명과학과 정인경 교수 연구팀이 서울대학교 암연구소 김태유 교수 연구팀과의 공동연구를 통해 인공지능 기반 알고리즘을 활용, 한국인 대장암 환자의 3차원 게놈 지도를 최초로 제시했으며 이를 토대로 암 세포 특이적인 유전자 조절 기전을 통해 특정 종양유전자들이 과발현되는 현상을 규명했다고 24일 밝혔다.
1차원적 게놈 서열 분석에 기반한 현재의 암 유전체 연구는 종양유전자들의 과발현 기작을 설명하는데 한계가 있었다. 하지만 3차원 공간상에 게놈이 어떻게 배열되는지를 분석하는 3차원 게놈 (3D genome) 구조 연구는 이러한 한계를 극복 가능케 하고 있다. 본 연구에서는 정상 세포에서는 존재하지 않는 암 세포 특이적 염색질 고리(chromatin loop) 구조가 유전자 발현 촉진 인자인 인핸서와 종양유전자 사이의 상호작용을 형성하여 과발현을 유도하는 인핸서 납치(enhancer-hijacking) 현상에 초점을 두어 연구하였다.
우리 대학 생명과학과 김규광 박사과정이 주도한 이번 연구는 게놈간의 공간상 상호작용을 측정할 수 있는 대용량 염색체 구조 포착 Hi-C (High-throughput Chromosome Conformation Capture) 실험 기법을 활용하여 대장암 3차원 게놈 지도를 작성하고 대장암 특이적 3차원 게놈 변화를 환자 개개인별로 분석할 수 있는 인공지능 기반 알고리즘을 개발했다. 그 결과 공동연구팀은 광범위한 규모의 3차원 게놈 구조 변화와 이로 인한 다양한 종양유전자의 활성화를 확인했다.
연구팀은 이번 연구를 통해 암 특이적 3차원 게놈 구조의 변화로 인한 종양유전자 활성 기작을 명확히 제시하였으며 이로 인한 환자 예후와 약물 반응 등 임상적인 특성과의 연관성까지 제시해 맞춤 치료 원천기술 확보에 기여했다.
지금까지 암 세포주에 대한 3차원 게놈 구조 연구는 일부 보고 되었으나, 대규모 환자 암조직에 대한 연구는 조직 내 세포 이질성, 종양 순도, 암세포 이질성 등의 문제로 인한 정밀 암 특이적 3차원 게놈 구조 분석의 한계로 수행되지 못하였다.
반면에 이번 연구에서 연구팀은 AI 기반 알고리즘으로 환자 개인 종양 조직으로부터 얻어진 복잡한 신호를 해석할 수 있었으며 그 결과 최대 규모인 환자 40명의 종양 조직과 인접한 정상 대장 조직을 사용해 3차원 게놈 지도를 작성할 수 있었다. 또한 DNA 서열정보를 보여주는 전장유전체 지도의 경우 다양한 인종에 대해 생산되고 있고 한국인의 전장유전체 지도 또한 개발되었으나 한국인 3차원 게놈 지도, 특히 종양 조직에 대한 3차원 게놈 지도는 이번 연구에서 최초로 제시됐다.
이번 연구 결과는 국제 학술지, `셀 리포츠(Cell Reports, IF=9.995)'에 7월 13일 자로 출판됐다. (논문명: Spatial and clonality-resolved 3D cancer genome alterations reveal enhancer-hijacking as a potential prognostic marker for colorectal cancer)
서울대학교병원 혈액종양내과 김태유 교수는 “이러한 결과는 개별 암 환자들마다 서로 다르게 나타나는 종양 이질성을 이해하는 데 매우 중요한 요소가 될 수 있으며, 이를 이용한 환자 맞춤형 치료 연구의 시발점이 될 것이다”라고 말했다. 생명과학과 정인경 교수는 “기존의 점돌연변이나 유전체 변이만으로는 설명이 어려운 암 유전체를 3차원 게놈 구조 관점에서 재해독하고 신규 암 타겟을 발굴할 수 있는 수 있는 새로운 접근법을 제시했다”라고 밝혔다.
한편 이번 연구는 과학기술정보통신부와 서경배과학재단의 지원을 받아 수행됐다.
2023.07.24 조회수 8691
인공지능 기반 대장암 3차원 게놈 지도 최초 해독
세계 최초로 예전에 비해 최대 규모로 한국인 대장암 환자 3차원 게놈 지도를 작성하여 화제다.
우리 대학 생명과학과 정인경 교수 연구팀이 서울대학교 암연구소 김태유 교수 연구팀과의 공동연구를 통해 인공지능 기반 알고리즘을 활용, 한국인 대장암 환자의 3차원 게놈 지도를 최초로 제시했으며 이를 토대로 암 세포 특이적인 유전자 조절 기전을 통해 특정 종양유전자들이 과발현되는 현상을 규명했다고 24일 밝혔다.
1차원적 게놈 서열 분석에 기반한 현재의 암 유전체 연구는 종양유전자들의 과발현 기작을 설명하는데 한계가 있었다. 하지만 3차원 공간상에 게놈이 어떻게 배열되는지를 분석하는 3차원 게놈 (3D genome) 구조 연구는 이러한 한계를 극복 가능케 하고 있다. 본 연구에서는 정상 세포에서는 존재하지 않는 암 세포 특이적 염색질 고리(chromatin loop) 구조가 유전자 발현 촉진 인자인 인핸서와 종양유전자 사이의 상호작용을 형성하여 과발현을 유도하는 인핸서 납치(enhancer-hijacking) 현상에 초점을 두어 연구하였다.
우리 대학 생명과학과 김규광 박사과정이 주도한 이번 연구는 게놈간의 공간상 상호작용을 측정할 수 있는 대용량 염색체 구조 포착 Hi-C (High-throughput Chromosome Conformation Capture) 실험 기법을 활용하여 대장암 3차원 게놈 지도를 작성하고 대장암 특이적 3차원 게놈 변화를 환자 개개인별로 분석할 수 있는 인공지능 기반 알고리즘을 개발했다. 그 결과 공동연구팀은 광범위한 규모의 3차원 게놈 구조 변화와 이로 인한 다양한 종양유전자의 활성화를 확인했다.
연구팀은 이번 연구를 통해 암 특이적 3차원 게놈 구조의 변화로 인한 종양유전자 활성 기작을 명확히 제시하였으며 이로 인한 환자 예후와 약물 반응 등 임상적인 특성과의 연관성까지 제시해 맞춤 치료 원천기술 확보에 기여했다.
지금까지 암 세포주에 대한 3차원 게놈 구조 연구는 일부 보고 되었으나, 대규모 환자 암조직에 대한 연구는 조직 내 세포 이질성, 종양 순도, 암세포 이질성 등의 문제로 인한 정밀 암 특이적 3차원 게놈 구조 분석의 한계로 수행되지 못하였다.
반면에 이번 연구에서 연구팀은 AI 기반 알고리즘으로 환자 개인 종양 조직으로부터 얻어진 복잡한 신호를 해석할 수 있었으며 그 결과 최대 규모인 환자 40명의 종양 조직과 인접한 정상 대장 조직을 사용해 3차원 게놈 지도를 작성할 수 있었다. 또한 DNA 서열정보를 보여주는 전장유전체 지도의 경우 다양한 인종에 대해 생산되고 있고 한국인의 전장유전체 지도 또한 개발되었으나 한국인 3차원 게놈 지도, 특히 종양 조직에 대한 3차원 게놈 지도는 이번 연구에서 최초로 제시됐다.
이번 연구 결과는 국제 학술지, `셀 리포츠(Cell Reports, IF=9.995)'에 7월 13일 자로 출판됐다. (논문명: Spatial and clonality-resolved 3D cancer genome alterations reveal enhancer-hijacking as a potential prognostic marker for colorectal cancer)
서울대학교병원 혈액종양내과 김태유 교수는 “이러한 결과는 개별 암 환자들마다 서로 다르게 나타나는 종양 이질성을 이해하는 데 매우 중요한 요소가 될 수 있으며, 이를 이용한 환자 맞춤형 치료 연구의 시발점이 될 것이다”라고 말했다. 생명과학과 정인경 교수는 “기존의 점돌연변이나 유전체 변이만으로는 설명이 어려운 암 유전체를 3차원 게놈 구조 관점에서 재해독하고 신규 암 타겟을 발굴할 수 있는 수 있는 새로운 접근법을 제시했다”라고 밝혔다.
한편 이번 연구는 과학기술정보통신부와 서경배과학재단의 지원을 받아 수행됐다.
2023.07.24 조회수 8691 -
 파킨슨병 발병 3차원 게놈 지도 최초 제시
파킨슨병은 60세 이상 인구의 1.2% 이상 발병하는 흔한 퇴행성 뇌 질환으로 급격한 인구 고령화에 따라 전 세계적으로 발병률이 증가하고 있어, 2040년 약 1,420만 명의 환자가 발병할 것으로 예측되고 있다. 현재 파킨슨병의 다양한 발병 원인이 명확하게 규명되지 않은 상황에서, 비정상적으로 발생하는 후성 유전학적 특징들이 파킨슨병 발병에 관여하는 것을 최초로 확인되어 화제다.
우리 대학 생명과학과 정인경 교수 연구팀이 미국 국립보건원(National Institute of Health, NIH) 산하 국립노화연구소(National Institute on Aging, NIA) 엘리에자 매슬리아(Eliezer Masliah) 교수와의 공동연구를 통해 전 세계 최초로 파킨슨병 발병 뇌 조직의 단일세포 3차원 후성유전체 지도를 작성하고, 이를 토대로 656개의 파킨슨병 연관 신규 유전자들을 제시했다고 8일 밝혔다.
이번 연구에서 연구팀은 최신 개발된 단일세포 유전체 기술과 3차원 후성 유전체 기술을 접목하여 신경세포 뿐 아니라 뇌 환경 유지에 주요한 역할을 하는 것으로 알려진 신경교세포 (희소돌기아교세포, 미세아교세포 등)의 후성유전적 변화들이 3차원 게놈 구조를 통해 파킨슨병 발병에 관여하는 것을 밝혔다. 이러한 비정상적인 후성유전학적 특징들은 파킨슨병의 원인 또는 진행에 관여하는 유전자 발현 조절에 핵심적인 역할을 하기 때문에, 본 연구 결과는 차후 진단과 치료 연구에 중요한 단서를 제공하게 될 것이라고 연구팀은 전했다.
이번 연구를 수행한 생명과학과 이정운 박사는 단일세포 수준에서 환자 뇌조직을 분석한 결과 기존의 신경세포에 국한된 연구에서 한발 나아가, 신경교세포 또한 파킨슨병에 중요한 역할을 할 수 있다는 단서를 제시하였다는 점에서 중요한 발견이라고 밝혔다.
이번 연구 결과는 국제 학술지, ‘사이언스 어드벤시스(Science Advances, IF=14.14)'에 4월 14일 게재됐다. (논문명 : Characterization of altered molecular mechanisms in Parkinson’s disease through cell type-resolved multi-omics analyses)
교신 저자인 정인경 교수는 "이번 연구 결과는 퇴행성 뇌 질환의 표적 발굴에 있어 3차원 후성유전체 지도 작성의 중요성을 보였기에 차후 다양한 복합유전질환 규명에도 중요하게 활용될 것이다ˮ라고 말했다.
한편 이번 연구는 서경배과학재단, 보건복지부, 과학기술정보통신부의 지원을 받아 수행됐다.
2023.05.08 조회수 7261
파킨슨병 발병 3차원 게놈 지도 최초 제시
파킨슨병은 60세 이상 인구의 1.2% 이상 발병하는 흔한 퇴행성 뇌 질환으로 급격한 인구 고령화에 따라 전 세계적으로 발병률이 증가하고 있어, 2040년 약 1,420만 명의 환자가 발병할 것으로 예측되고 있다. 현재 파킨슨병의 다양한 발병 원인이 명확하게 규명되지 않은 상황에서, 비정상적으로 발생하는 후성 유전학적 특징들이 파킨슨병 발병에 관여하는 것을 최초로 확인되어 화제다.
우리 대학 생명과학과 정인경 교수 연구팀이 미국 국립보건원(National Institute of Health, NIH) 산하 국립노화연구소(National Institute on Aging, NIA) 엘리에자 매슬리아(Eliezer Masliah) 교수와의 공동연구를 통해 전 세계 최초로 파킨슨병 발병 뇌 조직의 단일세포 3차원 후성유전체 지도를 작성하고, 이를 토대로 656개의 파킨슨병 연관 신규 유전자들을 제시했다고 8일 밝혔다.
이번 연구에서 연구팀은 최신 개발된 단일세포 유전체 기술과 3차원 후성 유전체 기술을 접목하여 신경세포 뿐 아니라 뇌 환경 유지에 주요한 역할을 하는 것으로 알려진 신경교세포 (희소돌기아교세포, 미세아교세포 등)의 후성유전적 변화들이 3차원 게놈 구조를 통해 파킨슨병 발병에 관여하는 것을 밝혔다. 이러한 비정상적인 후성유전학적 특징들은 파킨슨병의 원인 또는 진행에 관여하는 유전자 발현 조절에 핵심적인 역할을 하기 때문에, 본 연구 결과는 차후 진단과 치료 연구에 중요한 단서를 제공하게 될 것이라고 연구팀은 전했다.
이번 연구를 수행한 생명과학과 이정운 박사는 단일세포 수준에서 환자 뇌조직을 분석한 결과 기존의 신경세포에 국한된 연구에서 한발 나아가, 신경교세포 또한 파킨슨병에 중요한 역할을 할 수 있다는 단서를 제시하였다는 점에서 중요한 발견이라고 밝혔다.
이번 연구 결과는 국제 학술지, ‘사이언스 어드벤시스(Science Advances, IF=14.14)'에 4월 14일 게재됐다. (논문명 : Characterization of altered molecular mechanisms in Parkinson’s disease through cell type-resolved multi-omics analyses)
교신 저자인 정인경 교수는 "이번 연구 결과는 퇴행성 뇌 질환의 표적 발굴에 있어 3차원 후성유전체 지도 작성의 중요성을 보였기에 차후 다양한 복합유전질환 규명에도 중요하게 활용될 것이다ˮ라고 말했다.
한편 이번 연구는 서경배과학재단, 보건복지부, 과학기술정보통신부의 지원을 받아 수행됐다.
2023.05.08 조회수 7261 -
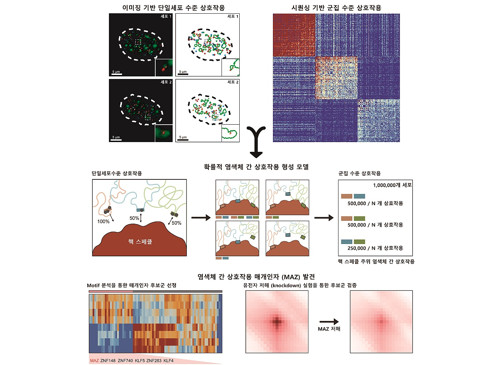 암, 노화 등에 미치는 게놈 3차 구조의 신규 원리 발견
3차원 게놈 구조 연구를 통해, 세포핵 내 게놈이 계층적인 구조로 이루어져 있으며 각 구조가 다양한 유전자 발현 조절에 관여한다는 것이 알려져 있다. 또한 이러한 게놈 3차 구조는 암, 노화 등 다양한 복합질환에서 질환 특이적 유전자 발현과 밀접한 연관이 있음이 최근 밝혀지고 있다. 하지만 기존 게놈 3차 구조는 비교적 관찰이 쉬운 염색체 내 상호작용에 대부분 국한되어 있었고, 더 큰 범위에서의 염색체 간 상호작용에 대해서는 관찰 실험 기법의 한계로 인해 연구가 거의 진행되지 않았다.
우리 대학은 생명과학과 정인경 교수 연구팀이 서울대학교 기계공학부 신용대 교수 연구팀, 부산대학교 최정모 교수 연구팀과의 공동연구를 통해 세포핵 내 3차원 게놈 구조 신규 생성 원리와 이를 조절하는 매개 인자를 발견했다고 10일(월) 밝혔다.
생명과학과 주재건 석박사통합과정과 서울대학교 조성현 연구원이 주도한 이번 연구에서 연구팀은 행렬 분해 기법이란 분석 기법을 활용하여 게놈 3차 구조 데이터로부터 염색체 간 상호작용 정보를 효과적으로 추출할 수 있는 신규 기계 학습 알고리즘을 개발했으며, 이를 DNA 이미징 기법을 통해 검증했다.
연구팀은 해당 분석 알고리즘을 이용해 여러 세포주의 염색체 간 상호작용 정보를 추출 및 분석했고, 핵 스페클(핵 내 존재하는 막이 없는 구조체) 주위에 위치한 염색체 간 상호작용이 여러 세포에서 공통적으로 보존됨을 관찰했다. 또한 연구팀은 단백질 인식 염기서열(DNA motif) 분석을 통해 스페클 주위 염색체 간 상호작용이 MAZ 단백질에 의해 매개됨을 최초로 발견했다.
또한, 연구팀은 단일세포 수준에서 염색체 간 상호작용이 세포마다 다르게 발생한다는 사실을 발견하였다. 연구팀은 염색체 간 상호작용이 기존에 알려져 있던 것과 달리 고정되어 있지 않으며, 핵체와 게놈 지역 사이의 개별 상호작용을 통해 확률적으로 결정된다는 내용을 제시하여 염색체 간 상호작용의 원리를 최초로 규명하였다.
이번 연구 결과는 국제 학술지, `핵산 연구(Nucleic acids research, IF=19.16)'에 4월 5일자 출판됐다. (논문제목: Probabilistic establishment of speckle-associated inter-chromosomal interactions)
이번 연구는 기존에 알려지지 않았던 염색체 간 상호작용의 형성 원리와 매개 인자인 MAZ 단백질의 역할을 밝힘으로써 더 큰 범위에서의 게놈 3차 구조에 대한 근본적인 원리 규명 단서를 제공했다는 점에서 큰 의의가 있다.
이번 연구를 주도한 우리 대학 주재건 석박사통합과정은 "그동안 실험 기법의 한계로 인해 가려져 있었던 염색체 간 상호작용 형성 원리를 밝혀낸 연구이다”라고 설명하였다. 우리 대학 정인경 교수는 “향후 게놈 3차 구조에 따른 유전자 발현 조절 분야와 암 질환 등에서 빈번하게 보고되고 있는 염색체 변이 원인 규명 등에서 핵체 (nuclear body)와 게놈 간 상호작용의 중요성을 이해할 수 있을 것으로 기대되는 성과ˮ 라고 연구 결과를 소개했다.
한편 이번 연구는 서경배과학재단, 삼성미래기술육성재단과 과학기술정보통신부의 지원을 받아 수행됐다.
2023.04.10 조회수 7962
암, 노화 등에 미치는 게놈 3차 구조의 신규 원리 발견
3차원 게놈 구조 연구를 통해, 세포핵 내 게놈이 계층적인 구조로 이루어져 있으며 각 구조가 다양한 유전자 발현 조절에 관여한다는 것이 알려져 있다. 또한 이러한 게놈 3차 구조는 암, 노화 등 다양한 복합질환에서 질환 특이적 유전자 발현과 밀접한 연관이 있음이 최근 밝혀지고 있다. 하지만 기존 게놈 3차 구조는 비교적 관찰이 쉬운 염색체 내 상호작용에 대부분 국한되어 있었고, 더 큰 범위에서의 염색체 간 상호작용에 대해서는 관찰 실험 기법의 한계로 인해 연구가 거의 진행되지 않았다.
우리 대학은 생명과학과 정인경 교수 연구팀이 서울대학교 기계공학부 신용대 교수 연구팀, 부산대학교 최정모 교수 연구팀과의 공동연구를 통해 세포핵 내 3차원 게놈 구조 신규 생성 원리와 이를 조절하는 매개 인자를 발견했다고 10일(월) 밝혔다.
생명과학과 주재건 석박사통합과정과 서울대학교 조성현 연구원이 주도한 이번 연구에서 연구팀은 행렬 분해 기법이란 분석 기법을 활용하여 게놈 3차 구조 데이터로부터 염색체 간 상호작용 정보를 효과적으로 추출할 수 있는 신규 기계 학습 알고리즘을 개발했으며, 이를 DNA 이미징 기법을 통해 검증했다.
연구팀은 해당 분석 알고리즘을 이용해 여러 세포주의 염색체 간 상호작용 정보를 추출 및 분석했고, 핵 스페클(핵 내 존재하는 막이 없는 구조체) 주위에 위치한 염색체 간 상호작용이 여러 세포에서 공통적으로 보존됨을 관찰했다. 또한 연구팀은 단백질 인식 염기서열(DNA motif) 분석을 통해 스페클 주위 염색체 간 상호작용이 MAZ 단백질에 의해 매개됨을 최초로 발견했다.
또한, 연구팀은 단일세포 수준에서 염색체 간 상호작용이 세포마다 다르게 발생한다는 사실을 발견하였다. 연구팀은 염색체 간 상호작용이 기존에 알려져 있던 것과 달리 고정되어 있지 않으며, 핵체와 게놈 지역 사이의 개별 상호작용을 통해 확률적으로 결정된다는 내용을 제시하여 염색체 간 상호작용의 원리를 최초로 규명하였다.
이번 연구 결과는 국제 학술지, `핵산 연구(Nucleic acids research, IF=19.16)'에 4월 5일자 출판됐다. (논문제목: Probabilistic establishment of speckle-associated inter-chromosomal interactions)
이번 연구는 기존에 알려지지 않았던 염색체 간 상호작용의 형성 원리와 매개 인자인 MAZ 단백질의 역할을 밝힘으로써 더 큰 범위에서의 게놈 3차 구조에 대한 근본적인 원리 규명 단서를 제공했다는 점에서 큰 의의가 있다.
이번 연구를 주도한 우리 대학 주재건 석박사통합과정은 "그동안 실험 기법의 한계로 인해 가려져 있었던 염색체 간 상호작용 형성 원리를 밝혀낸 연구이다”라고 설명하였다. 우리 대학 정인경 교수는 “향후 게놈 3차 구조에 따른 유전자 발현 조절 분야와 암 질환 등에서 빈번하게 보고되고 있는 염색체 변이 원인 규명 등에서 핵체 (nuclear body)와 게놈 간 상호작용의 중요성을 이해할 수 있을 것으로 기대되는 성과ˮ 라고 연구 결과를 소개했다.
한편 이번 연구는 서경배과학재단, 삼성미래기술육성재단과 과학기술정보통신부의 지원을 받아 수행됐다.
2023.04.10 조회수 7962 -
 기저 질환이 없는 코로나19 환자의 중증 신규 유전적 위험 인자 규명
우리 대학 생명과학과 정인경 교수 연구팀이 서울대병원 강창경, 고영일, 분당서울대병원 송경호 교수, 경북대병원 문준호 교수, 국립중앙의료원 이지연 교수, 지놈오피니언 社로 이루어진 산·학·병 공동연구를 통해 기저 질환이 없는 저위험군의 신규 코로나19 중증 위험 인자를 발굴하고, 발굴된 인자의 과잉 염증반응에 대한 분자 메커니즘을 제시했다고 29일 밝혔다.
코로나19 바이러스(SARS-CoV2)는 지난 2년이 넘도록 확산하면서 전 세계적으로 6억 명 이상이 감염됐고, 이 중 6백만 명 이상이 사망했다. 이러한 심각성으로 인해 코로나19 바이러스의 병리에 관한 연구가 활발히 진행됐고, 단핵구(큰 크기의 백혈구, Monocyte)의 과잉 염증반응으로 인한 중증 진행 메커니즘 등이 밝혀졌다.
하지만 개별 코로나19 환자마다 면역 반응의 편차가 크게 나타나는 현상에 대해서는 앞서 찾은 연구 결과만으로는 전부 설명할 수 없다. 예를 들어 중증 코로나19 환자 중에서 당뇨병이나 고혈압 등의 기저 질환이 없는 경우도 빈번하기에 이들이 코로나19 감염 시 중증으로 진행될 수 있는 신규 위험 인자를 발굴하는 것은 환자 맞춤형 치료에 있어 매우 중요하다.
우리 대학 생명과학과 최백규, 박성완 석박사통합과정과 서울대병원 강창경 교수가 주도한 이번 연구에서는 기존의 기저 질환이 없는 중증 코로나19 환자의 중증 요인을 알아내기 위해, 국내 4개의 병원이 합동해 총 243명의 코로나19 환자의 임상 정보를 수집 및 분석했다. 연구팀은 그 집단의 임상적 특징을 밝히고, 단일세포 유전자 발현 분석과 후성유전학적 분석을 도입해 관찰된 임상적 특징과 중증 코로나19 내 과잉 염증반응 간의 유전자 발현 조절 메커니즘을 분석했다.
그 결과, 기저 질환이 없는 집단 내 중증 환자는 `클론성조혈증'이라는 특징을 가지고 있는 것을 관찰하였다. 이는 혈액 및 면역 세포를 형성하는 골수 줄기세포 중 후천적 유전자 변이가 있는 집단을 의미한다. 또한 단일세포 유전자 발현 분석을 통해 클론성조혈증을 가진 중증 환자의 경우 단핵구에서 특이적인 과잉 염증반응이 관찰되는 것을 확인했고, 클론성조혈증으로 인해 변화한 후성유전학적 특징이 단핵구 특이적인 과잉 염증반응을 일으키는 유전자 발현을 유도하는 것을 연구팀은 확인했다.
해외 연구단에서도 유사하게 클론성조혈증과 코로나19 간의 관련성에 주목한 연구들이 있었으나 코로나19와의 관련성을 명확히 밝히지 못했고, 과잉 염증반응으로 이어지는 분자 모델 역시 제시하지 못했다. 이에 반해 공동 연구팀은 생물정보학 기반 계층화된 환자 분류법과 환자 유래 다양한 면역 세포를 단 하나의 세포 수준에서 유전자 발현 패턴 및 조절 기전을 해석할 수 있는 단일세포 오믹스 생물학 기법을 적용해 클론성조혈증이 코로나19의 신규 중증 인자임을 명확하게 제시했다. 해당 연구 결과는 앞으로 기저질환이 없는 저위험군 환자라도 클론성조혈증을 갖는 경우 코로나19 감염 시 보다 체계적인 치료 및 관리가 필요함을 의미한다.
이번 연구 결과는 두 개의 국제 학술지, `헤마톨로지카(haematologica, IF=11.04)'에 9월 15일 字 (논문명: Clinical impact of clonal hematopoiesis on severe COVID-19 patients without canonical risk factors) 온라인 게재가 되었으며. ‘실험 및 분자 의학(Experimental & Molecular Medicine, IF=11.590)'에 지난 8월 1일 字 (논문명: Single-cell transcriptome analyses reveal distinct gene expression signatures of severe COVID-19 in the presence of clonal hematopoiesis) 게재 승인됐다.
이번 연구는 장기화된 코로나19 팬데믹 상황 속에서 연구계·의료계·산업계로 이루어진 연구팀 서로 간의 긴밀한 협력을 통해 코로나19 환자의 신규 중증 인자를 밝히고, 그에 대한 분자적 기전을 제시해 환자별 맞춤 치료전략을 제시한 연구로 중개 연구(translational research)의 좋은 예시로 평가받는다.
이번 연구를 수행한 우리 대학 최백규 석박사통합과정은 "최신의 분자실험 기법인 단일세포 오믹스 실험과 생물정보학 분석 기술의 융합이 신규 코로나19 중증 환자의 아형과 관련 유전자 조절 기전을 규명 가능케 하였다ˮ며, "다른 질환에도 바이오 데이터 기반 융합 연구 기법을 적용할 것이다ˮ고 말했다.
분당서울대병원 송경호 교수는 "이번 연구는 임상 현장에서 코로나 환자별 맞춤 치료 전략을 정립하는 데 있어서 중요한 정보를 제공한 연구ˮ라며 "앞으로도 중증 코로나19 환자의 생존율을 높이기 위해 임상 정보를 바탕으로 한 맞춤 치료전략 연구를 이어나가겠다ˮ라고 밝혔다.
지놈오피니언 대표를 겸임하고 있는 서울대병원 고영일 교수는 "회사에서 개발한 클론성조혈증 탐지 및 분석 기술이 코로나19 팬데믹 해결에 도움이 되어 보람차다ˮ면서 "앞으로도 새로운 바이오마커를 발굴 및 분석하는 기술을 개발해 인류의 건강한 삶에 지속적으로 기여하고 싶다ˮ고 말했다.
한편 이번 연구는 서경배과학재단과 과학기술정보통신부의 지원을 받아 수행됐다.
2022.09.29 조회수 11437
기저 질환이 없는 코로나19 환자의 중증 신규 유전적 위험 인자 규명
우리 대학 생명과학과 정인경 교수 연구팀이 서울대병원 강창경, 고영일, 분당서울대병원 송경호 교수, 경북대병원 문준호 교수, 국립중앙의료원 이지연 교수, 지놈오피니언 社로 이루어진 산·학·병 공동연구를 통해 기저 질환이 없는 저위험군의 신규 코로나19 중증 위험 인자를 발굴하고, 발굴된 인자의 과잉 염증반응에 대한 분자 메커니즘을 제시했다고 29일 밝혔다.
코로나19 바이러스(SARS-CoV2)는 지난 2년이 넘도록 확산하면서 전 세계적으로 6억 명 이상이 감염됐고, 이 중 6백만 명 이상이 사망했다. 이러한 심각성으로 인해 코로나19 바이러스의 병리에 관한 연구가 활발히 진행됐고, 단핵구(큰 크기의 백혈구, Monocyte)의 과잉 염증반응으로 인한 중증 진행 메커니즘 등이 밝혀졌다.
하지만 개별 코로나19 환자마다 면역 반응의 편차가 크게 나타나는 현상에 대해서는 앞서 찾은 연구 결과만으로는 전부 설명할 수 없다. 예를 들어 중증 코로나19 환자 중에서 당뇨병이나 고혈압 등의 기저 질환이 없는 경우도 빈번하기에 이들이 코로나19 감염 시 중증으로 진행될 수 있는 신규 위험 인자를 발굴하는 것은 환자 맞춤형 치료에 있어 매우 중요하다.
우리 대학 생명과학과 최백규, 박성완 석박사통합과정과 서울대병원 강창경 교수가 주도한 이번 연구에서는 기존의 기저 질환이 없는 중증 코로나19 환자의 중증 요인을 알아내기 위해, 국내 4개의 병원이 합동해 총 243명의 코로나19 환자의 임상 정보를 수집 및 분석했다. 연구팀은 그 집단의 임상적 특징을 밝히고, 단일세포 유전자 발현 분석과 후성유전학적 분석을 도입해 관찰된 임상적 특징과 중증 코로나19 내 과잉 염증반응 간의 유전자 발현 조절 메커니즘을 분석했다.
그 결과, 기저 질환이 없는 집단 내 중증 환자는 `클론성조혈증'이라는 특징을 가지고 있는 것을 관찰하였다. 이는 혈액 및 면역 세포를 형성하는 골수 줄기세포 중 후천적 유전자 변이가 있는 집단을 의미한다. 또한 단일세포 유전자 발현 분석을 통해 클론성조혈증을 가진 중증 환자의 경우 단핵구에서 특이적인 과잉 염증반응이 관찰되는 것을 확인했고, 클론성조혈증으로 인해 변화한 후성유전학적 특징이 단핵구 특이적인 과잉 염증반응을 일으키는 유전자 발현을 유도하는 것을 연구팀은 확인했다.
해외 연구단에서도 유사하게 클론성조혈증과 코로나19 간의 관련성에 주목한 연구들이 있었으나 코로나19와의 관련성을 명확히 밝히지 못했고, 과잉 염증반응으로 이어지는 분자 모델 역시 제시하지 못했다. 이에 반해 공동 연구팀은 생물정보학 기반 계층화된 환자 분류법과 환자 유래 다양한 면역 세포를 단 하나의 세포 수준에서 유전자 발현 패턴 및 조절 기전을 해석할 수 있는 단일세포 오믹스 생물학 기법을 적용해 클론성조혈증이 코로나19의 신규 중증 인자임을 명확하게 제시했다. 해당 연구 결과는 앞으로 기저질환이 없는 저위험군 환자라도 클론성조혈증을 갖는 경우 코로나19 감염 시 보다 체계적인 치료 및 관리가 필요함을 의미한다.
이번 연구 결과는 두 개의 국제 학술지, `헤마톨로지카(haematologica, IF=11.04)'에 9월 15일 字 (논문명: Clinical impact of clonal hematopoiesis on severe COVID-19 patients without canonical risk factors) 온라인 게재가 되었으며. ‘실험 및 분자 의학(Experimental & Molecular Medicine, IF=11.590)'에 지난 8월 1일 字 (논문명: Single-cell transcriptome analyses reveal distinct gene expression signatures of severe COVID-19 in the presence of clonal hematopoiesis) 게재 승인됐다.
이번 연구는 장기화된 코로나19 팬데믹 상황 속에서 연구계·의료계·산업계로 이루어진 연구팀 서로 간의 긴밀한 협력을 통해 코로나19 환자의 신규 중증 인자를 밝히고, 그에 대한 분자적 기전을 제시해 환자별 맞춤 치료전략을 제시한 연구로 중개 연구(translational research)의 좋은 예시로 평가받는다.
이번 연구를 수행한 우리 대학 최백규 석박사통합과정은 "최신의 분자실험 기법인 단일세포 오믹스 실험과 생물정보학 분석 기술의 융합이 신규 코로나19 중증 환자의 아형과 관련 유전자 조절 기전을 규명 가능케 하였다ˮ며, "다른 질환에도 바이오 데이터 기반 융합 연구 기법을 적용할 것이다ˮ고 말했다.
분당서울대병원 송경호 교수는 "이번 연구는 임상 현장에서 코로나 환자별 맞춤 치료 전략을 정립하는 데 있어서 중요한 정보를 제공한 연구ˮ라며 "앞으로도 중증 코로나19 환자의 생존율을 높이기 위해 임상 정보를 바탕으로 한 맞춤 치료전략 연구를 이어나가겠다ˮ라고 밝혔다.
지놈오피니언 대표를 겸임하고 있는 서울대병원 고영일 교수는 "회사에서 개발한 클론성조혈증 탐지 및 분석 기술이 코로나19 팬데믹 해결에 도움이 되어 보람차다ˮ면서 "앞으로도 새로운 바이오마커를 발굴 및 분석하는 기술을 개발해 인류의 건강한 삶에 지속적으로 기여하고 싶다ˮ고 말했다.
한편 이번 연구는 서경배과학재단과 과학기술정보통신부의 지원을 받아 수행됐다.
2022.09.29 조회수 11437 -
 세계 최대 규모의 3차원 암 게놈 지도 구축
우리 대학 생명과학과 정인경 교수가 한국생명공학연구원 국가생명연구자원정보센터(KOBIC) 이병욱 박사 연구팀과 공동연구를 통해 전 세계 최대 규모의 3차원 암 게놈 지도 데이터베이스를 구축해 공개했다고 28일 밝혔다. (데이터베이스 주소: 3div.kr)
공동연구팀은 인체 정상 조직과 암 조직, 그리고 다양한 세포주 대상 3차원 게놈 지도를 분석 및 데이터베이스화 해, 약 400여 종 이상의 3차원 인간 게놈 지도를 구축했으며, 이를 통해 암세포에서 빈번하게 발생하는 대규모 유전체 구조 변이(structural variation)의 기능을 해독할 수 있는 신규 전략을 제시했다.
정인경 교수, 이병욱 박사가 공동 교신 저자로 참여한 이번 연구 결과는 국제 학술지 `핵산 연구(Nucleic Acid Research)' 저널 11월 27일 字 온라인판에 게재됐다. (논문명 : 3DIV update for 2021: a comprehensive resource of 3D genome and 3D cancer genome)
현재까지 많은 연구를 통해 암세포 유전체에서 발생하는 돌연변이를 규명해 암의 발병 기전을 이해하려는 시도가 있었다. 최근에는 유전자에서 발생하는 점 돌연변이뿐 아니라 대규모 구조 변이에 관한 연구가 활발하게 이루어지고 있으며, 이들을 활용한 신규 암세포의 특이적 유전자 발현 조절 기전 규명의 중요성이 제시되고 있다.
하지만, 대다수의 구조 변이는 DNA가 단백질을 생성하지 않는 비 전사 지역에 존재해, 1차원적 게놈 서열 분석만으로 이들의 기능을 규명하는 데는 한계가 있었다.
한편 지난 10년간 비약적으로 발전한 3차원 게놈 구조 연구는 비 전사 지역에 존재하는 대규모 구조 변이로 인해 생성되거나 소실되는 염색질 고리 구조(chromatin loop)를 3차원 게놈 구조 해독을 통해 규명하면 유전자 조절 기능을 해독할 수 있다는 모델을 제시하고 있다.
이에 정인경 교수 연구팀은 지금까지 공개된 모든 암 유전체의 3차원 게놈 지도를 확보해 전 세계 최대 규모의 3차원 암 유전체 지도를 작성했다. 그리고 대규모 구조 변이와 3차원 게놈 지도를 연결할 수 있는 분석 도구들을 개발했다. 그 결과 연구팀은 대규모 암 유전체 구조 변이에 따른 3차원 게놈 구조의 변화 그리고 이들의 표적 유전자를 규명할 수 있었다.
공동 교신 저자 이병욱 박사는 "최근 세포 내 3차원 게놈 구조 변화가 다양한 질병, 특히 암의 원인이 된다는 것이 밝혀지고 있는데, 이번 연구를 통해 이를 연구할 수 있는 도구들을 세계 최초로 개발했다ˮ라며 "이번 연구 결과를 활용하면 암의 발병 원리를 이해하고 더 나아가 항암제 개발에도 중요한 정보를 제공할 것으로 기대된다ˮ라고 말했다.
정인경 교수는 "암에서 빈번하게 발생하는 대규모 구조 변이의 기능을 3차원 게놈 구조 해독을 통해 정밀하게 규명 가능함을 보여줬다ˮ라며 "이번 연구 결과는 아직 해독이 완벽하게 이루어지고 있지 않은 암 유전체를 정밀하게 해독하는 기술을 한 단계 더 발전시키는 계기가 될 것이다”라고 말했다.
이번 연구는 한국연구재단 기반산업화 인프라 그리고 서경배과학재단의 지원을 통해 수행됐다.
2020.12.28 조회수 52917
세계 최대 규모의 3차원 암 게놈 지도 구축
우리 대학 생명과학과 정인경 교수가 한국생명공학연구원 국가생명연구자원정보센터(KOBIC) 이병욱 박사 연구팀과 공동연구를 통해 전 세계 최대 규모의 3차원 암 게놈 지도 데이터베이스를 구축해 공개했다고 28일 밝혔다. (데이터베이스 주소: 3div.kr)
공동연구팀은 인체 정상 조직과 암 조직, 그리고 다양한 세포주 대상 3차원 게놈 지도를 분석 및 데이터베이스화 해, 약 400여 종 이상의 3차원 인간 게놈 지도를 구축했으며, 이를 통해 암세포에서 빈번하게 발생하는 대규모 유전체 구조 변이(structural variation)의 기능을 해독할 수 있는 신규 전략을 제시했다.
정인경 교수, 이병욱 박사가 공동 교신 저자로 참여한 이번 연구 결과는 국제 학술지 `핵산 연구(Nucleic Acid Research)' 저널 11월 27일 字 온라인판에 게재됐다. (논문명 : 3DIV update for 2021: a comprehensive resource of 3D genome and 3D cancer genome)
현재까지 많은 연구를 통해 암세포 유전체에서 발생하는 돌연변이를 규명해 암의 발병 기전을 이해하려는 시도가 있었다. 최근에는 유전자에서 발생하는 점 돌연변이뿐 아니라 대규모 구조 변이에 관한 연구가 활발하게 이루어지고 있으며, 이들을 활용한 신규 암세포의 특이적 유전자 발현 조절 기전 규명의 중요성이 제시되고 있다.
하지만, 대다수의 구조 변이는 DNA가 단백질을 생성하지 않는 비 전사 지역에 존재해, 1차원적 게놈 서열 분석만으로 이들의 기능을 규명하는 데는 한계가 있었다.
한편 지난 10년간 비약적으로 발전한 3차원 게놈 구조 연구는 비 전사 지역에 존재하는 대규모 구조 변이로 인해 생성되거나 소실되는 염색질 고리 구조(chromatin loop)를 3차원 게놈 구조 해독을 통해 규명하면 유전자 조절 기능을 해독할 수 있다는 모델을 제시하고 있다.
이에 정인경 교수 연구팀은 지금까지 공개된 모든 암 유전체의 3차원 게놈 지도를 확보해 전 세계 최대 규모의 3차원 암 유전체 지도를 작성했다. 그리고 대규모 구조 변이와 3차원 게놈 지도를 연결할 수 있는 분석 도구들을 개발했다. 그 결과 연구팀은 대규모 암 유전체 구조 변이에 따른 3차원 게놈 구조의 변화 그리고 이들의 표적 유전자를 규명할 수 있었다.
공동 교신 저자 이병욱 박사는 "최근 세포 내 3차원 게놈 구조 변화가 다양한 질병, 특히 암의 원인이 된다는 것이 밝혀지고 있는데, 이번 연구를 통해 이를 연구할 수 있는 도구들을 세계 최초로 개발했다ˮ라며 "이번 연구 결과를 활용하면 암의 발병 원리를 이해하고 더 나아가 항암제 개발에도 중요한 정보를 제공할 것으로 기대된다ˮ라고 말했다.
정인경 교수는 "암에서 빈번하게 발생하는 대규모 구조 변이의 기능을 3차원 게놈 구조 해독을 통해 정밀하게 규명 가능함을 보여줬다ˮ라며 "이번 연구 결과는 아직 해독이 완벽하게 이루어지고 있지 않은 암 유전체를 정밀하게 해독하는 기술을 한 단계 더 발전시키는 계기가 될 것이다”라고 말했다.
이번 연구는 한국연구재단 기반산업화 인프라 그리고 서경배과학재단의 지원을 통해 수행됐다.
2020.12.28 조회수 52917 -
 정인경 교수, 인체 조직의 3차원 게놈지도 해독
〈 정인경 교수, 이정운 박사과정 〉
우리 대학 생명과학과 정인경 교수와 美 루드윅 암 연구소(Ludwig Institute of Cancer Research) 빙 렌 (Bing Ren) 교수 공동 연구팀이 인체 조직의 3차원 게놈 지도를 해독하는 데 성공했다.
연구팀은 인체의 27개 부위 조직의 3차원 게놈 지도를 분석해 치매, 심혈관계 질환 등을 포함한 2만 7천여 개 이상의 복합 질환 관련 유전 변이 기능을 예측했다.
정인경 교수, 빙 렌 교수가 공동 교신 저자로 참여한 이번 연구 결과는 국제 학술지 ‘네이처 제네틱스(Nature Genetics) 9월 10일 자 온라인판에 게재됐다. (논문명 : A compendium of promoter-centered long-range chromatin interactions in the human genome)
현재까지 수많은 연구를 통해 알츠하이머병, 파킨슨병, 자가면역질환 등 다양한 복합 질환의 원인을 규명하려는 시도가 이뤄지면서 실제 다수의 질환과 관련한 중요 유전변이가 발견됐다.
하지만 이들 대부분의 유전변이는 DNA가 단백질을 생성하지 않는 비전사 지역에 존재하기 때문에 1차원적 DNA 서열 분석에 기반한 유전체 연구로는 모든 기능을 규명하는 데 한계가 있다.
이에 지난 10년간 비약적으로 발전한 3차원 게놈 구조 연구는 비전사 지역에 존재하는 유전변이도 3차원 게놈 구조에 의해 형성되는 염색질 고리 구조(chromatin loop)를 통해 멀리 떨어진 유전자를 조절할 수 있다는 모델을 제시했다.
그러나 이러한 3차원 게놈 구조 연구는 몇 가지 세포주를 대상으로만 국한돼 있고, 질환과 직접 연관이 있는 각 인체 조직을 표적으로 한 게놈 3차 구조는 규명되지 않았다.
연구팀은 인체 내의 27개 조직을 대상으로 이들 게놈의 3차원 구조를 규명하기 위해 전사촉진 부위만 선택적으로 분석하는 ‘표적 염색질 3차 구조 포착법(promoter-capture Hi-C)’이라 불리는 신규 실험 기법을 활용해 고해상도의 3차원 게놈 참조 지도를 작성했다.
그 결과 인간 게놈에 존재하는 약 90만 개의 게놈 3차원 염색질 고리 구조를 발굴하고, 이들 중 상당수가 각 인체 조직 특이적으로 존재한다는 사실도 규명했다.
연구팀은 3차원 게놈 구조를 기반으로 지금까지 기능이 명확하게 정의되지 않은 2만 7천여 개 이상의 질환 연관 유전 변이의 표적 유전자를 정의해 이들 변이의 기능을 예측했다. 나아가 각 질환의 표적 유전자 유사도에 기반해 질환과 질환 사이의 신규 관계를 규명했고, 이를 바탕으로 여러 질환에 공통으로 관여하는 신규 분자 기전을 제시했다.
정 교수는 “복합 질환 기전 규명을 위해 비전사 게놈의 중요성을 강조하고 존재하는 다수의 중요 유전변이를 3차원 게놈 구조 해독을 통해 규명 가능함을 보였다”라며 “이번 결과는 퇴행성 뇌 질환을 포함 다양한 복합 질환의 신규 기전 규명 및 표적 발굴에 활용될 것이다”라고 말했다.
이번 연구는 한국연구재단 신진연구자지원사업, 보건복지부 질환극복기술개발사업, 서경배 과학재단의 지원을 통해 수행됐다.
□ 그림 설명
그림1. 3차원 게놈 구조 모식도 (modified from Stefano et al., 2016)
2019.09.24 조회수 12500
정인경 교수, 인체 조직의 3차원 게놈지도 해독
〈 정인경 교수, 이정운 박사과정 〉
우리 대학 생명과학과 정인경 교수와 美 루드윅 암 연구소(Ludwig Institute of Cancer Research) 빙 렌 (Bing Ren) 교수 공동 연구팀이 인체 조직의 3차원 게놈 지도를 해독하는 데 성공했다.
연구팀은 인체의 27개 부위 조직의 3차원 게놈 지도를 분석해 치매, 심혈관계 질환 등을 포함한 2만 7천여 개 이상의 복합 질환 관련 유전 변이 기능을 예측했다.
정인경 교수, 빙 렌 교수가 공동 교신 저자로 참여한 이번 연구 결과는 국제 학술지 ‘네이처 제네틱스(Nature Genetics) 9월 10일 자 온라인판에 게재됐다. (논문명 : A compendium of promoter-centered long-range chromatin interactions in the human genome)
현재까지 수많은 연구를 통해 알츠하이머병, 파킨슨병, 자가면역질환 등 다양한 복합 질환의 원인을 규명하려는 시도가 이뤄지면서 실제 다수의 질환과 관련한 중요 유전변이가 발견됐다.
하지만 이들 대부분의 유전변이는 DNA가 단백질을 생성하지 않는 비전사 지역에 존재하기 때문에 1차원적 DNA 서열 분석에 기반한 유전체 연구로는 모든 기능을 규명하는 데 한계가 있다.
이에 지난 10년간 비약적으로 발전한 3차원 게놈 구조 연구는 비전사 지역에 존재하는 유전변이도 3차원 게놈 구조에 의해 형성되는 염색질 고리 구조(chromatin loop)를 통해 멀리 떨어진 유전자를 조절할 수 있다는 모델을 제시했다.
그러나 이러한 3차원 게놈 구조 연구는 몇 가지 세포주를 대상으로만 국한돼 있고, 질환과 직접 연관이 있는 각 인체 조직을 표적으로 한 게놈 3차 구조는 규명되지 않았다.
연구팀은 인체 내의 27개 조직을 대상으로 이들 게놈의 3차원 구조를 규명하기 위해 전사촉진 부위만 선택적으로 분석하는 ‘표적 염색질 3차 구조 포착법(promoter-capture Hi-C)’이라 불리는 신규 실험 기법을 활용해 고해상도의 3차원 게놈 참조 지도를 작성했다.
그 결과 인간 게놈에 존재하는 약 90만 개의 게놈 3차원 염색질 고리 구조를 발굴하고, 이들 중 상당수가 각 인체 조직 특이적으로 존재한다는 사실도 규명했다.
연구팀은 3차원 게놈 구조를 기반으로 지금까지 기능이 명확하게 정의되지 않은 2만 7천여 개 이상의 질환 연관 유전 변이의 표적 유전자를 정의해 이들 변이의 기능을 예측했다. 나아가 각 질환의 표적 유전자 유사도에 기반해 질환과 질환 사이의 신규 관계를 규명했고, 이를 바탕으로 여러 질환에 공통으로 관여하는 신규 분자 기전을 제시했다.
정 교수는 “복합 질환 기전 규명을 위해 비전사 게놈의 중요성을 강조하고 존재하는 다수의 중요 유전변이를 3차원 게놈 구조 해독을 통해 규명 가능함을 보였다”라며 “이번 결과는 퇴행성 뇌 질환을 포함 다양한 복합 질환의 신규 기전 규명 및 표적 발굴에 활용될 것이다”라고 말했다.
이번 연구는 한국연구재단 신진연구자지원사업, 보건복지부 질환극복기술개발사업, 서경배 과학재단의 지원을 통해 수행됐다.
□ 그림 설명
그림1. 3차원 게놈 구조 모식도 (modified from Stefano et al., 2016)
2019.09.24 조회수 12500