%EB%8D%B0%EC%9D%B4%ED%84%B0%EB%B2%A0%EC%9D%B4%EC%8A%A4
-
 세계 최대 규모 암 데이터베이스 구축하다
디지털 암 정보 축적의 시대에는 데이터 생산을 넘어서, 데이터의 수집 및 관리 방법을 정립하고 거대 규모의 빅 데이터를 운용하는 것이 가장 큰 경쟁력이 될 수 있다. 전략적으로는 정밀 임상 정보와 연계할 수 있는 국내 생산 데이터와 다양성에 대한 이해를 도모할 수 있는 대규모 국제 데이터를 모두 수집해 통합하는 것은 매우 중요한 과제다.
우리 대학 의과학대학원 박종은 교수, 바이오및뇌공학과 최정균 교수 공동 연구팀(제1 저자: 강준호 박사, 이준형 박사)이 세계 최대 규모의 암 조직 단일세포 및 공간전사체* 데이터베이스를 구성하고, 이를 바탕으로 삼성서울병원 이세훈 교수 연구팀과 함께 면역 치료의 예후 예측에 중요한 세포 생태계 타입을 보고했다고 22일 밝혔다.
*단일세포 및 공간전사체: 모든 유전자의 발현 양상을 개별 세포 단위에서 혹은 3차원 조직 구조상에서 분석한 데이터
암은 우리 몸 안에서 스스로 진화하는 특성을 가지고 있어 암 조직 내의 세포 생태계를 구성하는 각 세포의 이질성과 이들의 상호작용을 파악하는 것이 가장 중요하다.
최근 발달하고 있는 단일세포 및 공간 전사체는 미세환경을 구성하는 세포들과 그들의 3차원적 배열 및 상호작용을 정량적으로 측정 및 표현한다는 점에서 미세환경의 이질성 개념을 생태계 수준으로 확장해 디지털 정보의 형태로 저장 및 분석할 수 있게 한다.
연구팀은 암세포 생태계 타입들을 전 암종(pan-cancer) 수준에서 규명하기 위해 약 1,000개의 암 환자 조직 샘플, 500여 명의 정상 조직 샘플에 대한 단일세포 전사체 데이터를 30종 이상의 암종에 대해 수집하여 모든 암에 대한 세포 지도가 총망라된 전 암종 단일세포 지도(pan-cancer single-cell atlas)를 구축했다.
내과 전문의가 포함된 연구진이 직접 데이터를 수집하고, 메타데이터 재처리 및 암종 분류를 진행함으로써 암 조직을 구성하는 100여 개의 세포 상태를 규정하고, 이들의 발생빈도를 바탕으로 각 암종별 조직의 상태를 분류했다. 또한 미국의 암 환자 공공 데이터베이스(TCGA) 등의 대규모 코호트 데이터를 활용해 각 세포 상태가 암 환자의 치료 및 예후에 미치는 영향을 분석했다.
특히 여러 세포 상태 간의 상호작용 분석을 통해서 암세포 생태계 네트워크를 구축하였고, 이 중에서 삼차 림프 구조(tertiary lymphoid structure)* 구성요소를 포함하는 인터페론 연관 생태계가 삼성서울병원 이세훈 교수 연구팀의 폐암 코호트를 포함해 면역관문 억제 치료(immune checkpoint inhibitor)**를 받은 여러 암종들에서 면역관문 억제 치료 반응 예측에 효과적임을 확인했다.
*삼차 림프 구조: 림프절과 유사하지만 건강한 조직에서는 형성되지 않고, 만성염증, 감염, 암 등이 있는 곳에서 면역 세포들이 조직화되어 형성되는 구조물
**면역관문 억제치료: T세포 혹은 암세포에서 발현되는 PD-1/PD-L1, CTLA-4와 같은 면역관문(immune checkpoint)을 차단하여 암세포와 싸우는 면역 반응을 활성화시키는 치료방법
연구를 주도한 박종은 교수는 “이번 연구를 통해 세계 최대 규모의 암 조직 데이터베이스를 구축하였고, 이를 바탕으로 면역 치료의 예후 예측에 중요한 영향을 줄 것이다. 또한 소수의 환자에게 아주 좋은 치료반응을 보이나 일부의 경우 면역 관련 부작용을 나타내는 면역 관문 억제제의 치료 대상군 선정에 큰 도움을 줄 것으로 기대된다.”고 말했다.
이번 연구 결과는 국제 학술지 ‘네이쳐 커뮤니케이션즈(Nature Communications)’ 지에 5월 14일 자 출판됐으며, KAIST 세포 아틀라스 웹 포탈 https://cellatlas.kaist.ac.kr 을 통해 공개되고 있다.
한편 이번 연구는 한국연구재단의 차세대바이오유망범용기술연구지원사업과 우수신진연구사업, 한국보건산업진흥원 연구중심병원 육성사업, 융합형의사과학자양성사업 및 포스코사이언스펠로우십의 지원을 받아 수행됐다.
2024.05.22 조회수 7695
세계 최대 규모 암 데이터베이스 구축하다
디지털 암 정보 축적의 시대에는 데이터 생산을 넘어서, 데이터의 수집 및 관리 방법을 정립하고 거대 규모의 빅 데이터를 운용하는 것이 가장 큰 경쟁력이 될 수 있다. 전략적으로는 정밀 임상 정보와 연계할 수 있는 국내 생산 데이터와 다양성에 대한 이해를 도모할 수 있는 대규모 국제 데이터를 모두 수집해 통합하는 것은 매우 중요한 과제다.
우리 대학 의과학대학원 박종은 교수, 바이오및뇌공학과 최정균 교수 공동 연구팀(제1 저자: 강준호 박사, 이준형 박사)이 세계 최대 규모의 암 조직 단일세포 및 공간전사체* 데이터베이스를 구성하고, 이를 바탕으로 삼성서울병원 이세훈 교수 연구팀과 함께 면역 치료의 예후 예측에 중요한 세포 생태계 타입을 보고했다고 22일 밝혔다.
*단일세포 및 공간전사체: 모든 유전자의 발현 양상을 개별 세포 단위에서 혹은 3차원 조직 구조상에서 분석한 데이터
암은 우리 몸 안에서 스스로 진화하는 특성을 가지고 있어 암 조직 내의 세포 생태계를 구성하는 각 세포의 이질성과 이들의 상호작용을 파악하는 것이 가장 중요하다.
최근 발달하고 있는 단일세포 및 공간 전사체는 미세환경을 구성하는 세포들과 그들의 3차원적 배열 및 상호작용을 정량적으로 측정 및 표현한다는 점에서 미세환경의 이질성 개념을 생태계 수준으로 확장해 디지털 정보의 형태로 저장 및 분석할 수 있게 한다.
연구팀은 암세포 생태계 타입들을 전 암종(pan-cancer) 수준에서 규명하기 위해 약 1,000개의 암 환자 조직 샘플, 500여 명의 정상 조직 샘플에 대한 단일세포 전사체 데이터를 30종 이상의 암종에 대해 수집하여 모든 암에 대한 세포 지도가 총망라된 전 암종 단일세포 지도(pan-cancer single-cell atlas)를 구축했다.
내과 전문의가 포함된 연구진이 직접 데이터를 수집하고, 메타데이터 재처리 및 암종 분류를 진행함으로써 암 조직을 구성하는 100여 개의 세포 상태를 규정하고, 이들의 발생빈도를 바탕으로 각 암종별 조직의 상태를 분류했다. 또한 미국의 암 환자 공공 데이터베이스(TCGA) 등의 대규모 코호트 데이터를 활용해 각 세포 상태가 암 환자의 치료 및 예후에 미치는 영향을 분석했다.
특히 여러 세포 상태 간의 상호작용 분석을 통해서 암세포 생태계 네트워크를 구축하였고, 이 중에서 삼차 림프 구조(tertiary lymphoid structure)* 구성요소를 포함하는 인터페론 연관 생태계가 삼성서울병원 이세훈 교수 연구팀의 폐암 코호트를 포함해 면역관문 억제 치료(immune checkpoint inhibitor)**를 받은 여러 암종들에서 면역관문 억제 치료 반응 예측에 효과적임을 확인했다.
*삼차 림프 구조: 림프절과 유사하지만 건강한 조직에서는 형성되지 않고, 만성염증, 감염, 암 등이 있는 곳에서 면역 세포들이 조직화되어 형성되는 구조물
**면역관문 억제치료: T세포 혹은 암세포에서 발현되는 PD-1/PD-L1, CTLA-4와 같은 면역관문(immune checkpoint)을 차단하여 암세포와 싸우는 면역 반응을 활성화시키는 치료방법
연구를 주도한 박종은 교수는 “이번 연구를 통해 세계 최대 규모의 암 조직 데이터베이스를 구축하였고, 이를 바탕으로 면역 치료의 예후 예측에 중요한 영향을 줄 것이다. 또한 소수의 환자에게 아주 좋은 치료반응을 보이나 일부의 경우 면역 관련 부작용을 나타내는 면역 관문 억제제의 치료 대상군 선정에 큰 도움을 줄 것으로 기대된다.”고 말했다.
이번 연구 결과는 국제 학술지 ‘네이쳐 커뮤니케이션즈(Nature Communications)’ 지에 5월 14일 자 출판됐으며, KAIST 세포 아틀라스 웹 포탈 https://cellatlas.kaist.ac.kr 을 통해 공개되고 있다.
한편 이번 연구는 한국연구재단의 차세대바이오유망범용기술연구지원사업과 우수신진연구사업, 한국보건산업진흥원 연구중심병원 육성사업, 융합형의사과학자양성사업 및 포스코사이언스펠로우십의 지원을 받아 수행됐다.
2024.05.22 조회수 7695 -
 세계 최고 성능을 지닌 데이터베이스 관리 시스템(DBMS) 기술 개발
우리 연구진이 방대한 정보를 저장하고 목적에 맞게 검색, 관리할 수 있는 시스템을 통칭하는 데이터베이스관리시스템(DBMS, DataBase Management System)을 세계 최고 수준의 성능으로 끌어올렸다.
우리 대학 전산학부 김민수 교수 연구팀이 데이터베이스 질의 언어 SQL(Structured Query Language, 구조화 질의어) 처리 성능을 대폭 높인 세계 최고 수준의 DBMS 기술을 개발했다.
김 교수 연구팀은 데이터 처리를 위해 산업 표준으로 사용되는 SQL 질의를 기존 DBMS와는 전혀 다른 방법으로 처리함으로써 성능을 기존 옴니사이(OmniSci) DBMS 대비 최대 88배나 높인 신기술을 개발했다. 김 교수팀이 개발한 이 기술은 오라클·마이크로소프트 SQL서버·IBM DB2 등 타 DBMS에도 적용할 수 있어 고성능 SQL 질의 처리가 필요한 다양한 곳에 폭넓게 적용될 수 있을 것으로 기대된다.
대부분의 DBMS는 SQL 질의를 처리할 때 내부적으로 데이터 테이블들을 `왼쪽 깊은 이진 트리(left-deep binary tree)' 형태로 배치해 처리하는 방법을 사용한다. 지난 수십 년간 상용화돼 온 대부분의 DBMS는 데이터 테이블들의 배치 가능한 가지 수가 기하급수적으로 많기 때문에 이를 `왼쪽 깊은 이진 트리' 형태로 배치해 SQL 질의를 처리해 왔다.
임의의 두 테이블이 기본 키(primary key, PK)와 외래 키(foreign key, FK)라 불리는 관계로 결합(조인 연산)하는 경우에는 이러한 방법으로 SQL 질의를 효과적으로 처리할 수 있다. 여기서 기본 키는 각 데이터 행(row)을 유일하게 식별할 수 있는 열(column)이고, 외래 키는 그렇지 않은 열이다.
지난 수십 년간 산업에서 사용되는 DB의 구조가 점점 복잡해지면서 두 테이블은 PK-FK 관계가 아닌 FK-FK 관계, 즉 외래 키와 외래 키의 관계로 결합하는 복잡한 형태의 SQL 질의들이 많아지고 있다. 실제 DBMS의 성능을 측정하는 산업 표준 벤치마크인 TPC-DS에서 전체 벤치마크의 26%가 이런 복잡한 SQL 질의들로 구성돼 있고 기계학습(머신러닝), 생물 정보학 등 다양한 분야들서도 이러한 복잡한 SQL 질의 사용이 점차 증가하는 추세다.
이전에 나온 DBMS들은 두 테이블이 주로 PK-FK 관계로 결합한다는 가정하에 개발됐기 때문에 FK-FK 결합이 필요한 복잡한 SQL 질의를 매우 느리거나 심지어 처리하지 못하는 실패를 거듭해왔다.
김 교수팀은 문제 해결을 위해 테이블들을 하나의 커다란 `왼쪽 깊은 이진 트리' 형태가 아닌 여러 개의 작은 `왼쪽 깊은 이진 트리'를 `n항 조인 연산자'로 묶는 형태로 배치해 처리하는 기술을 개발했다. 이때 각각의 `작은 이진 트리' 안에는 FK-FK 결합 관계가 발생하지 않도록 테이블들을 배치하는 것이 핵심이다.
각각의 `작은 이진 트리'의 처리 결과물을 `n항 조인 연산자'로 결합해 최종 결과물을 구하는 것도 난제로 꼽히는데 연구팀은 `최악-최적(worst-case optimal) 조인 알고리즘'이라는 방법으로 이 문제를 해결했다.
`최악-최적 조인 알고리즘'은 그래프 데이터를 처리할 때 이론적으로 가장 우수하다고 알려진 알고리즘이다. 김 교수 연구팀은 세계에서 가장 먼저 이 알고리즘을 SQL 질의 처리에 적용해 난제를 해결하는 데 성공했다.
김민수 교수 연구팀은 새로 개발한 DBMS 기술을 GPU 기반의 DBMS 개발업체인 미국 옴니사이(OmniSci)社 제품에 적용한 결과, OmniSci DBMS보다 성능이 최대 88배나 향상된 결과를 얻었다. 또 TPC-DS 벤치마크에서도 세계 최고 수준의 성능을 가진 기존의 상용 DBMS보다 5~20배나 더 빠른 사실을 확인했다. TPC-DS는 DBMS의 성능을 측정하기 위한 산업 표준의 최신 벤치마크이다.
교신저자로 참여한 김민수 교수는 "연구팀이 개발한 새로운 기술은 대부분의 DBMS에 적용할 수 있기 때문에 산업적 측면에서 파급 효과가 매우 클 것으로 기대한다ˮ 라고 말했다.
이번 연구에는 김 교수의 제자이자 미국 옴니사이(OmniSci)社에 재직 중인 남윤민 박사가 제1 저자로, 김 교수가 교신저자로 참여했으며 지난 18일 미국 오리건주 포틀랜드에서 열린 데이터베이스 분야 최고의 국제학술대회로 꼽히는 `시그모드(SIGMOD)'에서 발표됐다. (논문명 : SPRINTER: A Fast n-ary Join Query Processing Method for Complex OLAP Queries).
한편, 이 연구는 한국연구재단 선도연구센터 사업 및 중견연구자 지원사업, 과기정통부 IITP SW스타랩 사업의 지원을 받아 수행됐다.
2020.06.23 조회수 21442
세계 최고 성능을 지닌 데이터베이스 관리 시스템(DBMS) 기술 개발
우리 연구진이 방대한 정보를 저장하고 목적에 맞게 검색, 관리할 수 있는 시스템을 통칭하는 데이터베이스관리시스템(DBMS, DataBase Management System)을 세계 최고 수준의 성능으로 끌어올렸다.
우리 대학 전산학부 김민수 교수 연구팀이 데이터베이스 질의 언어 SQL(Structured Query Language, 구조화 질의어) 처리 성능을 대폭 높인 세계 최고 수준의 DBMS 기술을 개발했다.
김 교수 연구팀은 데이터 처리를 위해 산업 표준으로 사용되는 SQL 질의를 기존 DBMS와는 전혀 다른 방법으로 처리함으로써 성능을 기존 옴니사이(OmniSci) DBMS 대비 최대 88배나 높인 신기술을 개발했다. 김 교수팀이 개발한 이 기술은 오라클·마이크로소프트 SQL서버·IBM DB2 등 타 DBMS에도 적용할 수 있어 고성능 SQL 질의 처리가 필요한 다양한 곳에 폭넓게 적용될 수 있을 것으로 기대된다.
대부분의 DBMS는 SQL 질의를 처리할 때 내부적으로 데이터 테이블들을 `왼쪽 깊은 이진 트리(left-deep binary tree)' 형태로 배치해 처리하는 방법을 사용한다. 지난 수십 년간 상용화돼 온 대부분의 DBMS는 데이터 테이블들의 배치 가능한 가지 수가 기하급수적으로 많기 때문에 이를 `왼쪽 깊은 이진 트리' 형태로 배치해 SQL 질의를 처리해 왔다.
임의의 두 테이블이 기본 키(primary key, PK)와 외래 키(foreign key, FK)라 불리는 관계로 결합(조인 연산)하는 경우에는 이러한 방법으로 SQL 질의를 효과적으로 처리할 수 있다. 여기서 기본 키는 각 데이터 행(row)을 유일하게 식별할 수 있는 열(column)이고, 외래 키는 그렇지 않은 열이다.
지난 수십 년간 산업에서 사용되는 DB의 구조가 점점 복잡해지면서 두 테이블은 PK-FK 관계가 아닌 FK-FK 관계, 즉 외래 키와 외래 키의 관계로 결합하는 복잡한 형태의 SQL 질의들이 많아지고 있다. 실제 DBMS의 성능을 측정하는 산업 표준 벤치마크인 TPC-DS에서 전체 벤치마크의 26%가 이런 복잡한 SQL 질의들로 구성돼 있고 기계학습(머신러닝), 생물 정보학 등 다양한 분야들서도 이러한 복잡한 SQL 질의 사용이 점차 증가하는 추세다.
이전에 나온 DBMS들은 두 테이블이 주로 PK-FK 관계로 결합한다는 가정하에 개발됐기 때문에 FK-FK 결합이 필요한 복잡한 SQL 질의를 매우 느리거나 심지어 처리하지 못하는 실패를 거듭해왔다.
김 교수팀은 문제 해결을 위해 테이블들을 하나의 커다란 `왼쪽 깊은 이진 트리' 형태가 아닌 여러 개의 작은 `왼쪽 깊은 이진 트리'를 `n항 조인 연산자'로 묶는 형태로 배치해 처리하는 기술을 개발했다. 이때 각각의 `작은 이진 트리' 안에는 FK-FK 결합 관계가 발생하지 않도록 테이블들을 배치하는 것이 핵심이다.
각각의 `작은 이진 트리'의 처리 결과물을 `n항 조인 연산자'로 결합해 최종 결과물을 구하는 것도 난제로 꼽히는데 연구팀은 `최악-최적(worst-case optimal) 조인 알고리즘'이라는 방법으로 이 문제를 해결했다.
`최악-최적 조인 알고리즘'은 그래프 데이터를 처리할 때 이론적으로 가장 우수하다고 알려진 알고리즘이다. 김 교수 연구팀은 세계에서 가장 먼저 이 알고리즘을 SQL 질의 처리에 적용해 난제를 해결하는 데 성공했다.
김민수 교수 연구팀은 새로 개발한 DBMS 기술을 GPU 기반의 DBMS 개발업체인 미국 옴니사이(OmniSci)社 제품에 적용한 결과, OmniSci DBMS보다 성능이 최대 88배나 향상된 결과를 얻었다. 또 TPC-DS 벤치마크에서도 세계 최고 수준의 성능을 가진 기존의 상용 DBMS보다 5~20배나 더 빠른 사실을 확인했다. TPC-DS는 DBMS의 성능을 측정하기 위한 산업 표준의 최신 벤치마크이다.
교신저자로 참여한 김민수 교수는 "연구팀이 개발한 새로운 기술은 대부분의 DBMS에 적용할 수 있기 때문에 산업적 측면에서 파급 효과가 매우 클 것으로 기대한다ˮ 라고 말했다.
이번 연구에는 김 교수의 제자이자 미국 옴니사이(OmniSci)社에 재직 중인 남윤민 박사가 제1 저자로, 김 교수가 교신저자로 참여했으며 지난 18일 미국 오리건주 포틀랜드에서 열린 데이터베이스 분야 최고의 국제학술대회로 꼽히는 `시그모드(SIGMOD)'에서 발표됐다. (논문명 : SPRINTER: A Fast n-ary Join Query Processing Method for Complex OLAP Queries).
한편, 이 연구는 한국연구재단 선도연구센터 사업 및 중견연구자 지원사업, 과기정통부 IITP SW스타랩 사업의 지원을 받아 수행됐다.
2020.06.23 조회수 21442 -
 조광현 교수, 대장암 유발하는 돌연변이 유전자의 네트워크 원리 규명
〈 왼쪽위부터 시계방향으로 이종훈 박사과정, 공정렬 박사과정, 조광현 교수, 신동관 연구교수 〉
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 대장암이 발병하는 과정에서 생기는 유전자 네트워크의 원리를 규명하는 데 성공했다.
이를 통해 대장암의 근본적인 발병 원리를 밝혀낼 뿐 아니라 향후 새로운 개념의 효과적인 항암제의 분자표적을 찾는데 활용될 것으로 기대된다. 또한 4차 산업혁명의 핵심 기술로 주목받는 IT와 BT의 융합연구인 시스템생물학 연구로 규명해냈다는 의의를 갖는다.
신동관 박사, 이종훈, 공정렬 학생연구원 등이 함께 참여한 이번 연구는 ‘네이처 커뮤니케이션즈(Nature Communications)’ 2일자 온라인 판에 게재됐다.
인간의 암은 유전자 돌연변이에 의해 발생한다. 이 돌연변이의 빈도는 암종에 따라 차이가 나는데 백혈병, 소아암은 10여 개 정도이지만 성인 고형암은 평균 50여 개, 폐암 등의 외부인자로 인한 경우는 수백 개에 이른다.
전 세계 암연구자들은 암 치료를 위해 환자들에게서 빈번하게 발견되는 유전자 돌연변이들을 파악하고 이 중 주요 암 유발 유전자를 찾아내 표적 항암제를 개발하고자 노력했다.
그러나 유전자 돌연변이는 해당 유전자의 기능에만 영향을 주는 게 아니라 그 유전자와 상호작용하는 다른 유전자에게도 영향을 끼친다. 따라서 이러한 유전자 네트워크의 원리를 모른 채 소수의 암 유발 유전자를 대상으로 하는 현재의 치료법은 일부에게만 효과가 있고 쉽게 약물의 내성을 일으키는 한계가 있다.
조 교수 연구팀은 대장암 환자의 대규모 유전체 데이터를 이용해 유전자 상호작용 네트워크에서 나타나는 다중 돌연변이의 협력적 효과에 대한 수학모형을 구축했다.
이는 국제 암유전체컨소시엄에서 발표한 전암 유전체데이터베이스(TCGA: The Cancer Genome Atlas)를 토대로 구축한 것으로, 유전자 네트워크에서 나타나는 돌연변이의 영향력을 정량화하고 이를 이용해 대장암 환자 군을 임상 특징에 따라 군집화 하는데 성공했다.
또한 대규모 컴퓨터 시뮬레이션 분석을 통해 암 발생 과정에서 나타나는 임계전이(critical transition) 현상을 밝혀내 숨겨진 유전자 네트워크의 원리를 최초로 규명했다.
임계전이란 상전이와 같이 물질의 상태가 갑작스럽게 변화하는 현상을 말한다. 암 발생 과정에서는 유전자 돌연변이의 발생 순서를 추적하기 어렵기 때문에 전이 현상이 존재하는지 확인할 수 없었다.
연구팀은 시스템생물학 기반의 연구방법을 이용해 확인한 결과 기존의 대장암에서 잘 알려진 암 유발 유전자 돌연변이의 발생 순서를 따르는 경우에 임계전이 현상을 보임을 발견했다.
이번에 개발한 수학모형을 활용하면 암환자에게 발생하는 다수 유전자 돌연변이의 영향을 가장 효과적으로 저해할 수 있는 새로운 항암 표적 약물이 개발될 것으로 기대된다.
특히 주요 암 유발 유전자 뿐 아니라 돌연변이의 영향을 받는 다른 모든 유전자들을 대상으로 종합적으로 평가해 효과적인 약물 표적을 찾아낼 수 있다.
조 교수는 “지금껏 다수 유전자들의 돌연변이가 암 발생에 어떻게 기여하는지 밝혀진 바가 없었다”며 “이번 연구에서는 시스템생물학으로 암세포의 발달과정에서 유전자 네트워크의 원리를 최초로 밝힘으로써 새로운 차원의 항암제 표적을 발굴할 수 있는 가능성을 제시했다”고 말했다.
이번 연구는 과학기술정보통신부와 한국연구재단의 중견연구자지원사업과 바이오의료기술개발사업의 지원을 받아 수행됐다.
□ 그림 설명
그림1. 유전자 돌연변이의 영향력 전파에 의한 거대 클러스터의 형성
그림2. 암발생 과정에서 돌연변이 협력효과의 임계전이 현상
2017.11.07 조회수 24702
조광현 교수, 대장암 유발하는 돌연변이 유전자의 네트워크 원리 규명
〈 왼쪽위부터 시계방향으로 이종훈 박사과정, 공정렬 박사과정, 조광현 교수, 신동관 연구교수 〉
우리 대학 바이오및뇌공학과 조광현 교수 연구팀이 대장암이 발병하는 과정에서 생기는 유전자 네트워크의 원리를 규명하는 데 성공했다.
이를 통해 대장암의 근본적인 발병 원리를 밝혀낼 뿐 아니라 향후 새로운 개념의 효과적인 항암제의 분자표적을 찾는데 활용될 것으로 기대된다. 또한 4차 산업혁명의 핵심 기술로 주목받는 IT와 BT의 융합연구인 시스템생물학 연구로 규명해냈다는 의의를 갖는다.
신동관 박사, 이종훈, 공정렬 학생연구원 등이 함께 참여한 이번 연구는 ‘네이처 커뮤니케이션즈(Nature Communications)’ 2일자 온라인 판에 게재됐다.
인간의 암은 유전자 돌연변이에 의해 발생한다. 이 돌연변이의 빈도는 암종에 따라 차이가 나는데 백혈병, 소아암은 10여 개 정도이지만 성인 고형암은 평균 50여 개, 폐암 등의 외부인자로 인한 경우는 수백 개에 이른다.
전 세계 암연구자들은 암 치료를 위해 환자들에게서 빈번하게 발견되는 유전자 돌연변이들을 파악하고 이 중 주요 암 유발 유전자를 찾아내 표적 항암제를 개발하고자 노력했다.
그러나 유전자 돌연변이는 해당 유전자의 기능에만 영향을 주는 게 아니라 그 유전자와 상호작용하는 다른 유전자에게도 영향을 끼친다. 따라서 이러한 유전자 네트워크의 원리를 모른 채 소수의 암 유발 유전자를 대상으로 하는 현재의 치료법은 일부에게만 효과가 있고 쉽게 약물의 내성을 일으키는 한계가 있다.
조 교수 연구팀은 대장암 환자의 대규모 유전체 데이터를 이용해 유전자 상호작용 네트워크에서 나타나는 다중 돌연변이의 협력적 효과에 대한 수학모형을 구축했다.
이는 국제 암유전체컨소시엄에서 발표한 전암 유전체데이터베이스(TCGA: The Cancer Genome Atlas)를 토대로 구축한 것으로, 유전자 네트워크에서 나타나는 돌연변이의 영향력을 정량화하고 이를 이용해 대장암 환자 군을 임상 특징에 따라 군집화 하는데 성공했다.
또한 대규모 컴퓨터 시뮬레이션 분석을 통해 암 발생 과정에서 나타나는 임계전이(critical transition) 현상을 밝혀내 숨겨진 유전자 네트워크의 원리를 최초로 규명했다.
임계전이란 상전이와 같이 물질의 상태가 갑작스럽게 변화하는 현상을 말한다. 암 발생 과정에서는 유전자 돌연변이의 발생 순서를 추적하기 어렵기 때문에 전이 현상이 존재하는지 확인할 수 없었다.
연구팀은 시스템생물학 기반의 연구방법을 이용해 확인한 결과 기존의 대장암에서 잘 알려진 암 유발 유전자 돌연변이의 발생 순서를 따르는 경우에 임계전이 현상을 보임을 발견했다.
이번에 개발한 수학모형을 활용하면 암환자에게 발생하는 다수 유전자 돌연변이의 영향을 가장 효과적으로 저해할 수 있는 새로운 항암 표적 약물이 개발될 것으로 기대된다.
특히 주요 암 유발 유전자 뿐 아니라 돌연변이의 영향을 받는 다른 모든 유전자들을 대상으로 종합적으로 평가해 효과적인 약물 표적을 찾아낼 수 있다.
조 교수는 “지금껏 다수 유전자들의 돌연변이가 암 발생에 어떻게 기여하는지 밝혀진 바가 없었다”며 “이번 연구에서는 시스템생물학으로 암세포의 발달과정에서 유전자 네트워크의 원리를 최초로 밝힘으로써 새로운 차원의 항암제 표적을 발굴할 수 있는 가능성을 제시했다”고 말했다.
이번 연구는 과학기술정보통신부와 한국연구재단의 중견연구자지원사업과 바이오의료기술개발사업의 지원을 받아 수행됐다.
□ 그림 설명
그림1. 유전자 돌연변이의 영향력 전파에 의한 거대 클러스터의 형성
그림2. 암발생 과정에서 돌연변이 협력효과의 임계전이 현상
2017.11.07 조회수 24702 -
 우운택 교수, 스마트 관광 증강현실 어플리케이션 개발
〈 우 운 택 교수 〉
우리 대학 문화기술대학원 우운택 교수 연구팀이 스마트 관광 지원을 위한 증강 및 가상현실 어플리케이션을 개발했다.
‘케이 컬처 타임머신(K-Culture Time Machine)’ 어플리케이션은 창덕궁을 대상으로 한 시범 서비스로 iOS 앱스토어에 5월 23일 공개됐다.
개발된 케이 컬처 타임머신은 웨어러블 360도 비디오를 통해 문화유산이나 유적지에 대한 시공간을 넘는 원격 체험을 제공한다.
사용자는 VR기기에 스마트폰을 장착해 제공되는 360도 비디오로 문화 유적지를 원격으로 체험하고 해당 문화유산 및 연관관계가 있는 인물, 장소, 사건 등에 대한 정보를 확인할 수 있다. 또한 소실된 문화유산에 대한 3차원 디지털 복원도 체험할 수 있다.
웨어러블 기기 활용 없이도 모바일 모드를 통해 사용자 주변 유적지 확인, 카메라에 인식된 문화유산을 인식하고 관련된 정보와 콘텐츠를 제공하는 증강현실 기반의 문화유산 가이드가 가능하다.
사용자는 자신의 위치에서 창덕궁 돈화문을 시작으로 인정문, 인정전, 희정당에 이르는 창덕궁 내부를 이동하며 360도 파노라마 이미지나 비디오를 통해 현장을 가상체험 할 수 있다.
현재는 존재하지 않는 인정전 동쪽의 궐내 곽사 지역에는 3D모델을 통한 승정원의 가상 복원을 확인할 수 있다.
위 기능은 웨어러블 기기 없이 스마트폰 상에서도 체험 가능하며 개발 중인 증강현실 기능이 완성되면 현장에서 활용 가능한 수준의 어플리케이션이 될 것으로 기대된다.
우 교수 연구팀은 문화유산 데이터베이스와 증강-가상현실 콘텐츠의 표준화된 메타데이터를 구축하고 이를 적용했다. 이를 활용해 일시적으로 개발 후 소비되는 기존 어플리케이션과는 달리 추가적인 콘텐츠 생성 및 추가가 가능하다.
우 교수는 “증강현실 콘텐츠의 상호 활용성과 재활용성을 증진해 스마트관광 분야의 새로운 시장을 선점할 수 있을 것이다”며 “콘텐츠 개발 비용 절감과 증강현실 콘텐츠 생태계 활성화를 가능하게 하는 다양한 부가 효과도 기대한다”고 말했다.
이번 연구는 ㈜포스트미디어(대표 홍승모)와 문화체육관광부 CT R&D 사업과의 공동 수행을 통해 이뤄졌으며, 관련 연구 성과는 올 7월 캐나다에서 진행되는 HCII 2017 학회를 통해 발표될 예정이다.
□ 그림 설명
그림1. 360 VR 서비스 개념도
그림2. K-Culture Time Machine의 모바일 증강현실 기능 구동 화면
그림3. K-Culture Time Machine의 360도 파노라마 이미지-비디오 기능 화면
2017.05.23 조회수 21076
우운택 교수, 스마트 관광 증강현실 어플리케이션 개발
〈 우 운 택 교수 〉
우리 대학 문화기술대학원 우운택 교수 연구팀이 스마트 관광 지원을 위한 증강 및 가상현실 어플리케이션을 개발했다.
‘케이 컬처 타임머신(K-Culture Time Machine)’ 어플리케이션은 창덕궁을 대상으로 한 시범 서비스로 iOS 앱스토어에 5월 23일 공개됐다.
개발된 케이 컬처 타임머신은 웨어러블 360도 비디오를 통해 문화유산이나 유적지에 대한 시공간을 넘는 원격 체험을 제공한다.
사용자는 VR기기에 스마트폰을 장착해 제공되는 360도 비디오로 문화 유적지를 원격으로 체험하고 해당 문화유산 및 연관관계가 있는 인물, 장소, 사건 등에 대한 정보를 확인할 수 있다. 또한 소실된 문화유산에 대한 3차원 디지털 복원도 체험할 수 있다.
웨어러블 기기 활용 없이도 모바일 모드를 통해 사용자 주변 유적지 확인, 카메라에 인식된 문화유산을 인식하고 관련된 정보와 콘텐츠를 제공하는 증강현실 기반의 문화유산 가이드가 가능하다.
사용자는 자신의 위치에서 창덕궁 돈화문을 시작으로 인정문, 인정전, 희정당에 이르는 창덕궁 내부를 이동하며 360도 파노라마 이미지나 비디오를 통해 현장을 가상체험 할 수 있다.
현재는 존재하지 않는 인정전 동쪽의 궐내 곽사 지역에는 3D모델을 통한 승정원의 가상 복원을 확인할 수 있다.
위 기능은 웨어러블 기기 없이 스마트폰 상에서도 체험 가능하며 개발 중인 증강현실 기능이 완성되면 현장에서 활용 가능한 수준의 어플리케이션이 될 것으로 기대된다.
우 교수 연구팀은 문화유산 데이터베이스와 증강-가상현실 콘텐츠의 표준화된 메타데이터를 구축하고 이를 적용했다. 이를 활용해 일시적으로 개발 후 소비되는 기존 어플리케이션과는 달리 추가적인 콘텐츠 생성 및 추가가 가능하다.
우 교수는 “증강현실 콘텐츠의 상호 활용성과 재활용성을 증진해 스마트관광 분야의 새로운 시장을 선점할 수 있을 것이다”며 “콘텐츠 개발 비용 절감과 증강현실 콘텐츠 생태계 활성화를 가능하게 하는 다양한 부가 효과도 기대한다”고 말했다.
이번 연구는 ㈜포스트미디어(대표 홍승모)와 문화체육관광부 CT R&D 사업과의 공동 수행을 통해 이뤄졌으며, 관련 연구 성과는 올 7월 캐나다에서 진행되는 HCII 2017 학회를 통해 발표될 예정이다.
□ 그림 설명
그림1. 360 VR 서비스 개념도
그림2. K-Culture Time Machine의 모바일 증강현실 기능 구동 화면
그림3. K-Culture Time Machine의 360도 파노라마 이미지-비디오 기능 화면
2017.05.23 조회수 21076 -
 스마트폰으로 시공간 뛰어넘는 문화유산 관람한다
〈 우 운 택 교수 〉
우리 대학 문화기술대학원 김정화, 우운택 교수 공동연구팀이 문화체육관광부의 지원을 받아 스마트 관광 지원을 위한 모바일 증강현실 플랫폼 ‘K-컬쳐 타임머신(K-Culture Time Machine)’을 개발했다.
이 기술은 1월 27일부터 3일간 강원도 하이원리조트에서 열리는 한국 HCI(인간-컴퓨터 상호작용) 학회에서 발표될 예정이다.
이번에 개발한 플랫폼은 증강현실을 통해 문화유산이나 유적지의 과거를 체험하고 엿볼 수 있는 기능을 제공한다.
위치 혹은 객체를 인식한 후 단순한 부가정보만을 제공했던 기존 모바일 증강현실을 넘어 향후 모바일 증강현실 응용 생태계를 구축하는 초석이 될 것으로 기대된다.
연구팀의 핵심 기술은 문화유산 데이터베이스를 연계하는 메타데이터 모델을 구축하고, 이를 기반으로 문화유산 공간의 현재와 과거 정보를 체험할 수 있는 기술이다.
스마트폰에 설치된 플랫폼을 실행한 후 유적지나 문화유산을 스마트폰에 비추면 관련 문화재에 대한 설명이 제공된다. 데이터베이스가 연계됐기 때문에 문화재청, 박물관포탈의 E-뮤지엄, 한국민족문화대백과사전 등의 정보를 한 눈에 볼 수 있다.
연구팀은 또한 문화유산과 관련된 인물, 유물, 장소, 사건 등을 정의하고 연관관계를 분석해 온톨로지를 설계하고 구현했다. 창덕궁 인정전을 예로 들면 유물 카테고리에서 ‘인정전을 구성하는 이화문 장식’을, 사건 카테고리에서 ‘인정전에서 발생한 왕세자탄강진하례’등을 검색할 수 있다.
이를 통해 각자 독립적으로 구축된 정보 간의 연계가 가능하고, 개발 후 소비되는 기존 시스템과 달리 지속적인 서비스를 제공할 수 있다.
K-컬쳐 타임머신은 이름처럼 문화유산의 과거와 현재를 오가며 시공간을 넘나드는 체험을 할 수 있다. 각 시대에 있었던 사건, 사진, 연관성을 지닌 정보를 확인할 수 있고, 증강현실을 통해 현재 문화유산의 모습에서 과거 모습을 투영하는 것이 가능하다.
이 증강현실 플랫폼의 기반인 데이터모델 및 메타데이터 표준은 작년 12월 한국정보통신기술협회로부터 정식 인준됐다.
박물관, 도서관 등 각기 다양한 문화유산을 다루는 기관을 통합하는 유럽의 유로피아나 프로젝트(Europeana Data Model : EDM)처럼 국내의 다양한 문화유산 데이터베이스를 연계하고 활용할 수 있는 온톨로지 데이터 모델 KCHDM(Korean Cultural Heritage Data Motel)을 개발해 표준화했다.
우 교수는 “증강현실 콘텐츠의 메타데이터 체계를 개발하고 표준화해 다양한 증강현실 콘텐츠의 재사용 및 개별 플랫폼과 독립적으로 콘텐츠 공유가 가능케 할 것이다”며 “향후 우리나라의 문화유산 정보시스템과 소셜미디어 기반의 신뢰성 높은 추천 정보, 사용자 프로파일을 증강현실 콘텐츠로 연계 활용해 지속 가능한 증강현실 생태계를 구축하고 일상에 확산할 수 있을 것이다”고 말했다.
이번 기술 개발은 ㈜포스트미디어(대표 홍승모)와의 공동연구를 통해 진행됐다.
연구팀의 모바일 기반 스마트 투어지원 플랫폼과 관련된 기술 논문은 인간-컴퓨터 상호작용 관련 국제학술대회인 HCI International 2015에서 발표될 예정이다.
□ 그림 설명
그림 1. 스마트 유적지 투어 지원 플랫폼 ‘K-Culture Time Machine’의 개념도
그림2. 본 플랫폼을 활용한 모바일 증강현실 어플리케이션에서의 서비스 구동 실제 화면
2016.01.29 조회수 18875
스마트폰으로 시공간 뛰어넘는 문화유산 관람한다
〈 우 운 택 교수 〉
우리 대학 문화기술대학원 김정화, 우운택 교수 공동연구팀이 문화체육관광부의 지원을 받아 스마트 관광 지원을 위한 모바일 증강현실 플랫폼 ‘K-컬쳐 타임머신(K-Culture Time Machine)’을 개발했다.
이 기술은 1월 27일부터 3일간 강원도 하이원리조트에서 열리는 한국 HCI(인간-컴퓨터 상호작용) 학회에서 발표될 예정이다.
이번에 개발한 플랫폼은 증강현실을 통해 문화유산이나 유적지의 과거를 체험하고 엿볼 수 있는 기능을 제공한다.
위치 혹은 객체를 인식한 후 단순한 부가정보만을 제공했던 기존 모바일 증강현실을 넘어 향후 모바일 증강현실 응용 생태계를 구축하는 초석이 될 것으로 기대된다.
연구팀의 핵심 기술은 문화유산 데이터베이스를 연계하는 메타데이터 모델을 구축하고, 이를 기반으로 문화유산 공간의 현재와 과거 정보를 체험할 수 있는 기술이다.
스마트폰에 설치된 플랫폼을 실행한 후 유적지나 문화유산을 스마트폰에 비추면 관련 문화재에 대한 설명이 제공된다. 데이터베이스가 연계됐기 때문에 문화재청, 박물관포탈의 E-뮤지엄, 한국민족문화대백과사전 등의 정보를 한 눈에 볼 수 있다.
연구팀은 또한 문화유산과 관련된 인물, 유물, 장소, 사건 등을 정의하고 연관관계를 분석해 온톨로지를 설계하고 구현했다. 창덕궁 인정전을 예로 들면 유물 카테고리에서 ‘인정전을 구성하는 이화문 장식’을, 사건 카테고리에서 ‘인정전에서 발생한 왕세자탄강진하례’등을 검색할 수 있다.
이를 통해 각자 독립적으로 구축된 정보 간의 연계가 가능하고, 개발 후 소비되는 기존 시스템과 달리 지속적인 서비스를 제공할 수 있다.
K-컬쳐 타임머신은 이름처럼 문화유산의 과거와 현재를 오가며 시공간을 넘나드는 체험을 할 수 있다. 각 시대에 있었던 사건, 사진, 연관성을 지닌 정보를 확인할 수 있고, 증강현실을 통해 현재 문화유산의 모습에서 과거 모습을 투영하는 것이 가능하다.
이 증강현실 플랫폼의 기반인 데이터모델 및 메타데이터 표준은 작년 12월 한국정보통신기술협회로부터 정식 인준됐다.
박물관, 도서관 등 각기 다양한 문화유산을 다루는 기관을 통합하는 유럽의 유로피아나 프로젝트(Europeana Data Model : EDM)처럼 국내의 다양한 문화유산 데이터베이스를 연계하고 활용할 수 있는 온톨로지 데이터 모델 KCHDM(Korean Cultural Heritage Data Motel)을 개발해 표준화했다.
우 교수는 “증강현실 콘텐츠의 메타데이터 체계를 개발하고 표준화해 다양한 증강현실 콘텐츠의 재사용 및 개별 플랫폼과 독립적으로 콘텐츠 공유가 가능케 할 것이다”며 “향후 우리나라의 문화유산 정보시스템과 소셜미디어 기반의 신뢰성 높은 추천 정보, 사용자 프로파일을 증강현실 콘텐츠로 연계 활용해 지속 가능한 증강현실 생태계를 구축하고 일상에 확산할 수 있을 것이다”고 말했다.
이번 기술 개발은 ㈜포스트미디어(대표 홍승모)와의 공동연구를 통해 진행됐다.
연구팀의 모바일 기반 스마트 투어지원 플랫폼과 관련된 기술 논문은 인간-컴퓨터 상호작용 관련 국제학술대회인 HCI International 2015에서 발표될 예정이다.
□ 그림 설명
그림 1. 스마트 유적지 투어 지원 플랫폼 ‘K-Culture Time Machine’의 개념도
그림2. 본 플랫폼을 활용한 모바일 증강현실 어플리케이션에서의 서비스 구동 실제 화면
2016.01.29 조회수 18875 -
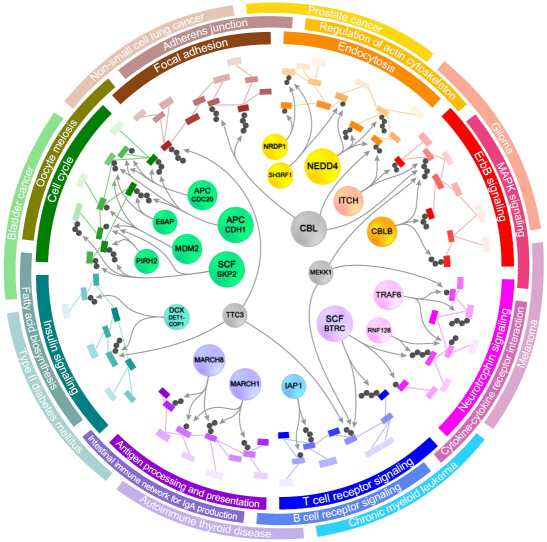 단백질 분해조절 효소 정보 담은 바이오마커 발굴 시스템 개발
- Mol Cell Proteomics지 게재, “바이오마커 개발의 새로운 패러다임 제시” -
단백질의 분해를 조절하는 효소와 기질에 대한 관계정보를 담은 바이오마커* 발굴 시스템(E3Net)이 국내 연구진에 의해 개발되어, 고부가가치의 새로운 바이오마커 개발에 가능성이 열렸다.
※ 바이오마커(Biomarker) : 유전자, 단백질 등에서 유래된 특이한 패턴의 분자적 정보로, 유전적․후천적 영향으로 발생한 신체의 변화를 감지할 수 있는 생물표지인자
우리학교 바이오및뇍 이관수 교수(49세)가 주도하고, 한영웅 박사과정생, 이호동 박사 및 박종철 교수가 참여한 이번 연구는 교육과학기술부(장관 이주호)와 한국연구재단(이사장 이승종)이 추진하는 선도연구센터지원사업(NCRC), 신기술융합형성장동력사업 및 교육과학기술부의 KAIST 미래형 시스템 헬스케어 연구개발사업의 지원으로 수행되었고, 단백질체 연구 분야의 권위 있는 학술지인 ‘Molecular and Cellular Proteomics"지 4월호(4월 1일자)에 게재되었다. (논문명: A system for exploring E3-mediated regulatory networks of cellular functions)이관수 교수 연구팀은 전 세계 바이오 관련 DB(데이터베이스)와 논문(약 2만 편)으로부터 정보를 추출해 단백질 분해를 조절하는 효소(E3 효소)와 기질*들 간의 네트워크를 집대성하여, 이와 관련된 세포의 기능과 질병을 분석하는 ‘E3Net’ 시스템을 개발하였다.
※ 기질(substrate) : 효소와 특이적으로 결합하여 화학반응을 일으키는 분자로, 소화작용은 우리의 몸속에서 일어나는 효소와 기질간의 반응의 대표적인 사례
세포는 시시각각 변하는 환경에 대응하여 필요한 단백질들을 생산, 폐기 및 재활용하는 정교한 시스템을 가지고 있는데, 만일 이 과정에서 오류가 생기면 ‘질병’으로 이어질 수 있다.
따라서 단백질 분해를 조절하는 E3 효소와 기질 간의 관계를 파악하면 관련 질병을 치료하거나 예방할 수 있게 된다. 특히 E3 효소는 단백질 분해의 80%를 담당하는 것으로 알려져 수많은 질병이 관련되어 있을 것으로 예측되고 있다.
그러나 E3 효소와 기질 간의 정보들이 개별 논문과 DB에 흩어져 있어, 단백질 분해 조절과 관련된 세포의 기능과 질병의 특성을 종합적․체계적으로 분석할 수 없었다.
이 교수팀은 모든 E3 효소(2,201개)와 기질(4,896개) 및 그 조절관계(1,671개)에 대한 정보를 통합하여 E3 효소 조절 네트워크 내에 존재하는 관련된 세포의 기능과 질병을 시스템적으로 분석할 수 있는 E3Net을 구축하는데 성공하였다.
이 네트워크는 지금까지 구축된 조절정보를 모두 합친 것보다 무려 10배에 이르는 방대한 양으로, E3 효소가 독자적으로 또는 협력해서 조절하는 세포의 기능과 관련 질병을 정확히 파악할 수 있는 토대가 마련된 첫 사례로서 의미가 크다.
연구팀은 E3Net을 이용하면 각각의 질병과 관련된 단백질들의 분해조절을 담당하는 E3 효소들을 찾을 수 있고, 분해조절 원리와 세포기능 네트워크를 함께 파악하여 질병의 발생 원인이나 환자에 적합한 맞춤형 치료방법을 제공할 수 있는 바이오마커를 발굴할 수 있을 것으로 기대한다.
실제 연구팀은 E3Net을 활용해 암, 뇌심혈관 질환 및 당뇨병 등 현대인의 대표적 질환과 관련된 E3 바이오마커 후보 수십 개를 새롭게 발견하는 등 눈에 띄는 성과를 거두었고, 현재 이를 검증할 후속 연구를 계획하고 있다. 이관수 교수는 “이번 연구결과로 E3 효소와 관련된 단백질 분해조절의 네트워크가 구축되고, 이 네트워크에 존재하는 세포의 기능과 질병의 특이성을 시스템적으로 분석할 수 있게 됨에 따라, E3 효소와 관련된 세포의 기능 연구와 질병 연구에 새로운 전기가 마련되었다”고 연구의의를 밝혔다.
2012.05.01 조회수 25247
단백질 분해조절 효소 정보 담은 바이오마커 발굴 시스템 개발
- Mol Cell Proteomics지 게재, “바이오마커 개발의 새로운 패러다임 제시” -
단백질의 분해를 조절하는 효소와 기질에 대한 관계정보를 담은 바이오마커* 발굴 시스템(E3Net)이 국내 연구진에 의해 개발되어, 고부가가치의 새로운 바이오마커 개발에 가능성이 열렸다.
※ 바이오마커(Biomarker) : 유전자, 단백질 등에서 유래된 특이한 패턴의 분자적 정보로, 유전적․후천적 영향으로 발생한 신체의 변화를 감지할 수 있는 생물표지인자
우리학교 바이오및뇍 이관수 교수(49세)가 주도하고, 한영웅 박사과정생, 이호동 박사 및 박종철 교수가 참여한 이번 연구는 교육과학기술부(장관 이주호)와 한국연구재단(이사장 이승종)이 추진하는 선도연구센터지원사업(NCRC), 신기술융합형성장동력사업 및 교육과학기술부의 KAIST 미래형 시스템 헬스케어 연구개발사업의 지원으로 수행되었고, 단백질체 연구 분야의 권위 있는 학술지인 ‘Molecular and Cellular Proteomics"지 4월호(4월 1일자)에 게재되었다. (논문명: A system for exploring E3-mediated regulatory networks of cellular functions)이관수 교수 연구팀은 전 세계 바이오 관련 DB(데이터베이스)와 논문(약 2만 편)으로부터 정보를 추출해 단백질 분해를 조절하는 효소(E3 효소)와 기질*들 간의 네트워크를 집대성하여, 이와 관련된 세포의 기능과 질병을 분석하는 ‘E3Net’ 시스템을 개발하였다.
※ 기질(substrate) : 효소와 특이적으로 결합하여 화학반응을 일으키는 분자로, 소화작용은 우리의 몸속에서 일어나는 효소와 기질간의 반응의 대표적인 사례
세포는 시시각각 변하는 환경에 대응하여 필요한 단백질들을 생산, 폐기 및 재활용하는 정교한 시스템을 가지고 있는데, 만일 이 과정에서 오류가 생기면 ‘질병’으로 이어질 수 있다.
따라서 단백질 분해를 조절하는 E3 효소와 기질 간의 관계를 파악하면 관련 질병을 치료하거나 예방할 수 있게 된다. 특히 E3 효소는 단백질 분해의 80%를 담당하는 것으로 알려져 수많은 질병이 관련되어 있을 것으로 예측되고 있다.
그러나 E3 효소와 기질 간의 정보들이 개별 논문과 DB에 흩어져 있어, 단백질 분해 조절과 관련된 세포의 기능과 질병의 특성을 종합적․체계적으로 분석할 수 없었다.
이 교수팀은 모든 E3 효소(2,201개)와 기질(4,896개) 및 그 조절관계(1,671개)에 대한 정보를 통합하여 E3 효소 조절 네트워크 내에 존재하는 관련된 세포의 기능과 질병을 시스템적으로 분석할 수 있는 E3Net을 구축하는데 성공하였다.
이 네트워크는 지금까지 구축된 조절정보를 모두 합친 것보다 무려 10배에 이르는 방대한 양으로, E3 효소가 독자적으로 또는 협력해서 조절하는 세포의 기능과 관련 질병을 정확히 파악할 수 있는 토대가 마련된 첫 사례로서 의미가 크다.
연구팀은 E3Net을 이용하면 각각의 질병과 관련된 단백질들의 분해조절을 담당하는 E3 효소들을 찾을 수 있고, 분해조절 원리와 세포기능 네트워크를 함께 파악하여 질병의 발생 원인이나 환자에 적합한 맞춤형 치료방법을 제공할 수 있는 바이오마커를 발굴할 수 있을 것으로 기대한다.
실제 연구팀은 E3Net을 활용해 암, 뇌심혈관 질환 및 당뇨병 등 현대인의 대표적 질환과 관련된 E3 바이오마커 후보 수십 개를 새롭게 발견하는 등 눈에 띄는 성과를 거두었고, 현재 이를 검증할 후속 연구를 계획하고 있다. 이관수 교수는 “이번 연구결과로 E3 효소와 관련된 단백질 분해조절의 네트워크가 구축되고, 이 네트워크에 존재하는 세포의 기능과 질병의 특이성을 시스템적으로 분석할 수 있게 됨에 따라, E3 효소와 관련된 세포의 기능 연구와 질병 연구에 새로운 전기가 마련되었다”고 연구의의를 밝혔다.
2012.05.01 조회수 25247