-
 표적 DNA 저렴하게 분석 가능한 유전자 진단 기술 개발
박 현 규 교수
우리 대학 생명화학공학과 박현규 교수 연구팀이 특정 단백질이나 효소를 인식하는 물질인 압타머(Aptamer : 표적 물질과 결합할 수 있는 특성을 가진 DNA)를 이용해 다양한 표적 DNA를 분석할 수 있는 기술을 개발했다.
이 기술을 통해 메르스와 같은 신종 바이러스 병원균 감염 여부 등 다양한 유전자를 기존에 비해 저렴한 가격으로 진단할 수 있을 것으로 기대된다.
이번 연구결과는 영국왕립화학회가 발행하는 케미컬 커뮤니케이션즈(Chemical communications) 6월호 후면 표지논문으로 선정됐다.
기존의 분자 비콘(Molecular beacon) 프로브 기반 유전자 분석은 분석 대상인 표적 DNA가 변경되면 이에 대응하는 새로운 분자 비콘 프로브가 필요했다. 따라서 다양한 표적 DNA를 분석하는데 많은 비용이 필요하다는 한계가 있었다.
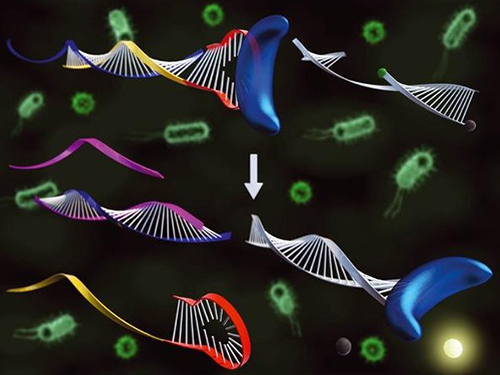
문제 해결을 위해 연구팀은 DNA 중합효소와 결합해 활성을 저해시키는 압타머를 고안했다. 그리고 이를 역으로 이용해 표적 DNA가 존재하는 경우에만 압타머가 DNA 중합효소와 결합하지 않고 활성을 유지할 수 있게 조절하는 기술을 최초로 개발했다.
이 기술 개발로 조절된 DNA 중합효소의 활성이 핵산 신장 및 절단 반응을 일으키고 그 결과로 형광 프로브(TaqMan probe)의 형광신호 측정이 가능해졌다. 따라서 동일한 형광 프로브를 이용해 다양한 표적 DNA를 민감하게 검출할 수 있는 새로운 유전자 진단 기술 개발이 가능해졌다.
이 기술은 표적 DNA의 종류에 따라 새로운 프로브를 사용해야 했던 기존 기술과 달리 동일한 형광 프로브를 이용하기 때문에 다양한 표적핵산을 값싸고 손쉽게 검출할 수 있다. 기술을 응용하면 과거에 비해 여러 가지 다른 병원균의 감염 여부를 저렴하고 수월하게 파악할 수 있다.
박 교수는 “메르스처럼 새로운 병원체에 대한 진단 키트를 용이하게 제작할 수 있어 여러 병원균에 대해 신속히 대응할 수 있다”며 “향후 유전자 진단 분야에서 새 원천기술로 널리 활용될 것으로 기대된다”고 말했다.
이번 연구는 미래창조과학부가 추진하는 글로벌프론티어사업(바이오나노헬스가드연구단)의 지원을 받아 수행됐다.
□ 그림 설명
그림 1. 표적핵산에 의한 DNA 중합효소 활성 변화를 이용해 표적 핵산을 검출한 모식도
표적 DNA 저렴하게 분석 가능한 유전자 진단 기술 개발
박 현 규 교수
우리 대학 생명화학공학과 박현규 교수 연구팀이 특정 단백질이나 효소를 인식하는 물질인 압타머(Aptamer : 표적 물질과 결합할 수 있는 특성을 가진 DNA)를 이용해 다양한 표적 DNA를 분석할 수 있는 기술을 개발했다.
이 기술을 통해 메르스와 같은 신종 바이러스 병원균 감염 여부 등 다양한 유전자를 기존에 비해 저렴한 가격으로 진단할 수 있을 것으로 기대된다.
이번 연구결과는 영국왕립화학회가 발행하는 케미컬 커뮤니케이션즈(Chemical communications) 6월호 후면 표지논문으로 선정됐다.
기존의 분자 비콘(Molecular beacon) 프로브 기반 유전자 분석은 분석 대상인 표적 DNA가 변경되면 이에 대응하는 새로운 분자 비콘 프로브가 필요했다. 따라서 다양한 표적 DNA를 분석하는데 많은 비용이 필요하다는 한계가 있었다.
문제 해결을 위해 연구팀은 DNA 중합효소와 결합해 활성을 저해시키는 압타머를 고안했다. 그리고 이를 역으로 이용해 표적 DNA가 존재하는 경우에만 압타머가 DNA 중합효소와 결합하지 않고 활성을 유지할 수 있게 조절하는 기술을 최초로 개발했다.
이 기술 개발로 조절된 DNA 중합효소의 활성이 핵산 신장 및 절단 반응을 일으키고 그 결과로 형광 프로브(TaqMan probe)의 형광신호 측정이 가능해졌다. 따라서 동일한 형광 프로브를 이용해 다양한 표적 DNA를 민감하게 검출할 수 있는 새로운 유전자 진단 기술 개발이 가능해졌다.
이 기술은 표적 DNA의 종류에 따라 새로운 프로브를 사용해야 했던 기존 기술과 달리 동일한 형광 프로브를 이용하기 때문에 다양한 표적핵산을 값싸고 손쉽게 검출할 수 있다. 기술을 응용하면 과거에 비해 여러 가지 다른 병원균의 감염 여부를 저렴하고 수월하게 파악할 수 있다.
박 교수는 “메르스처럼 새로운 병원체에 대한 진단 키트를 용이하게 제작할 수 있어 여러 병원균에 대해 신속히 대응할 수 있다”며 “향후 유전자 진단 분야에서 새 원천기술로 널리 활용될 것으로 기대된다”고 말했다.
이번 연구는 미래창조과학부가 추진하는 글로벌프론티어사업(바이오나노헬스가드연구단)의 지원을 받아 수행됐다.
□ 그림 설명
그림 1. 표적핵산에 의한 DNA 중합효소 활성 변화를 이용해 표적 핵산을 검출한 모식도
2015.07.27
조회수 12502
-
 DNA 기반 반도체 핵심 원천기술 개발
박현규 교수
- 분자 비콘을 이용해 모든(8가지) 논리게이트 구현하는 데 성공 -- 스몰(Small)誌 7월호 표지논문으로 실려 -
초소형 미래 바이오전자기기를 구현하기 위한 핵심기술 개발됐다.
우리 학교 생명화학공학과 박현규 교수 연구팀이 DNA를 이용해 모든 논리게이트를 구현하는 데 성공, 나노분야의 세계적 학술지 ‘스몰(Small)’ 7월호(23일자) 표지논문으로 실렸다.
현재 최첨단 기술로도 10nm(나노미터) 이하의 실리콘 기반 반도체 제작은 불가능한 것으로 알려져 있지만, DNA는 굵기가 2nm 정도로 가늘기 때문에 보다 저렴하면서도 획기적인 집적도를 가진 반도체를 만들 수 있을 것으로 기대된다.
2나노급 반도체가 개발되면 우표 크기의 메모리 반도체에 고화질 영화 10000편을 저장하는 등 현재 상용화중인 20나노급 반도체보다 약 100배의 용량을 담을 수 있게 된다.
DNA는 네 종류의 염기인 아데닌(adenine, A), 시토신(cytosine, C), 구아닌(guanin, G), 티민(thymine, T)이 연속적으로 연결돼 있는데 A는 T와, G는 C와 각각 특이적으로 결합하는 특성을 갖고 있다.
특정 DNA는 특이적으로 결합하는 염기서열을 지닌 또 다른 DNA와 결합해 이중나선 구조를 형성하는 데, 연구팀은 이러한 DNA의 특이적 결합 특성과 구조 변화에 따른 형광신호 특성이 있는 고리모양의 분자 비콘을 이용했다.
연구팀은 생체 DNA물질을 디지털 회로에서 사용되는 논리게이트와 같은 역할을 담당하도록 입력신호로 사용해 고리모양의 DNA가 열리거나 닫히도록 했다.
고리모양 DNA가 열린 형태에서는 형광신호가 증가하고 닫힌 상태에서는 형광 신호가 감소하며 이로 인해 발생하는 형광신호의 변화를 출력신호로 사용했다.
연구팀은 제한적인 시스템만을 구현하는 기존의 논리게이트의 문제점을 극복, 8가지 모든 논리게이트(AND, OR, XOR, INHIBIT, NAND, NOR, XNOR, IMPlCATION)를 구현하는 데 성공해 반도체 기술로써의 적용 가능성을 높였다.
이와 함께, 각각의 논리게이트의 연결을 통한 다중 논리게이트(Multilevel circuits)와 논리게이트의 재생성을 보여주는 데도 성공했다.
박현규 교수는 “하나의 분자 비콘을 모든 게이트 구성을 위한 보편적인 요소로 사용해 저렴하면서도 초고집적 바이오 전자기기의 가능성을 높였다”며 “앞으로 분자 수준의 전자 소자 연구에 큰 변화가 있을 것으로 예상된다”고 말했다.
이번 연구를 주도한 박기수 박사과정 학생(제1저자)은 “DNA는 10개의 염기서열 길이가 3.4nm이고 굵기가 2nm밖에 되지 않는 매우 작은 물질이기 때문에 이를 이용해 전자 소자를 구현하면 획기적인 집적도 향상을 이룰 수 있다”며 “간단한 시스템 디자인을 통해 정확한 논리게이트를 구현해 내 DNA 반도체를 탑재한 바이오컴퓨터가 곧 현실로 다가올 것”이라고 말했다.XOR 게이트 : 입력 DNA A(input A)와 입력 DNA B(input B) 둘 중 하나만 있을 때는 고리모양 DNA가 열려서 형광 신호가 나오고(출력신호 1), DNA A와 B가 모두 없을 경우와 모두 있는 경우에는 고리모양 DNA가 고리모양을 유지하여 형광을 발생하지 않게 함으로써 XOR 논리게이트를 정확하게 구현했다.
DNA 기반 반도체 핵심 원천기술 개발
박현규 교수
- 분자 비콘을 이용해 모든(8가지) 논리게이트 구현하는 데 성공 -- 스몰(Small)誌 7월호 표지논문으로 실려 -
초소형 미래 바이오전자기기를 구현하기 위한 핵심기술 개발됐다.
우리 학교 생명화학공학과 박현규 교수 연구팀이 DNA를 이용해 모든 논리게이트를 구현하는 데 성공, 나노분야의 세계적 학술지 ‘스몰(Small)’ 7월호(23일자) 표지논문으로 실렸다.
현재 최첨단 기술로도 10nm(나노미터) 이하의 실리콘 기반 반도체 제작은 불가능한 것으로 알려져 있지만, DNA는 굵기가 2nm 정도로 가늘기 때문에 보다 저렴하면서도 획기적인 집적도를 가진 반도체를 만들 수 있을 것으로 기대된다.
2나노급 반도체가 개발되면 우표 크기의 메모리 반도체에 고화질 영화 10000편을 저장하는 등 현재 상용화중인 20나노급 반도체보다 약 100배의 용량을 담을 수 있게 된다.
DNA는 네 종류의 염기인 아데닌(adenine, A), 시토신(cytosine, C), 구아닌(guanin, G), 티민(thymine, T)이 연속적으로 연결돼 있는데 A는 T와, G는 C와 각각 특이적으로 결합하는 특성을 갖고 있다.
특정 DNA는 특이적으로 결합하는 염기서열을 지닌 또 다른 DNA와 결합해 이중나선 구조를 형성하는 데, 연구팀은 이러한 DNA의 특이적 결합 특성과 구조 변화에 따른 형광신호 특성이 있는 고리모양의 분자 비콘을 이용했다.
연구팀은 생체 DNA물질을 디지털 회로에서 사용되는 논리게이트와 같은 역할을 담당하도록 입력신호로 사용해 고리모양의 DNA가 열리거나 닫히도록 했다.
고리모양 DNA가 열린 형태에서는 형광신호가 증가하고 닫힌 상태에서는 형광 신호가 감소하며 이로 인해 발생하는 형광신호의 변화를 출력신호로 사용했다.
연구팀은 제한적인 시스템만을 구현하는 기존의 논리게이트의 문제점을 극복, 8가지 모든 논리게이트(AND, OR, XOR, INHIBIT, NAND, NOR, XNOR, IMPlCATION)를 구현하는 데 성공해 반도체 기술로써의 적용 가능성을 높였다.
이와 함께, 각각의 논리게이트의 연결을 통한 다중 논리게이트(Multilevel circuits)와 논리게이트의 재생성을 보여주는 데도 성공했다.
박현규 교수는 “하나의 분자 비콘을 모든 게이트 구성을 위한 보편적인 요소로 사용해 저렴하면서도 초고집적 바이오 전자기기의 가능성을 높였다”며 “앞으로 분자 수준의 전자 소자 연구에 큰 변화가 있을 것으로 예상된다”고 말했다.
이번 연구를 주도한 박기수 박사과정 학생(제1저자)은 “DNA는 10개의 염기서열 길이가 3.4nm이고 굵기가 2nm밖에 되지 않는 매우 작은 물질이기 때문에 이를 이용해 전자 소자를 구현하면 획기적인 집적도 향상을 이룰 수 있다”며 “간단한 시스템 디자인을 통해 정확한 논리게이트를 구현해 내 DNA 반도체를 탑재한 바이오컴퓨터가 곧 현실로 다가올 것”이라고 말했다.XOR 게이트 : 입력 DNA A(input A)와 입력 DNA B(input B) 둘 중 하나만 있을 때는 고리모양 DNA가 열려서 형광 신호가 나오고(출력신호 1), DNA A와 B가 모두 없을 경우와 모두 있는 경우에는 고리모양 DNA가 고리모양을 유지하여 형광을 발생하지 않게 함으로써 XOR 논리게이트를 정확하게 구현했다.
2012.09.18
조회수 16762
 표적 DNA 저렴하게 분석 가능한 유전자 진단 기술 개발
박 현 규 교수
우리 대학 생명화학공학과 박현규 교수 연구팀이 특정 단백질이나 효소를 인식하는 물질인 압타머(Aptamer : 표적 물질과 결합할 수 있는 특성을 가진 DNA)를 이용해 다양한 표적 DNA를 분석할 수 있는 기술을 개발했다.
이 기술을 통해 메르스와 같은 신종 바이러스 병원균 감염 여부 등 다양한 유전자를 기존에 비해 저렴한 가격으로 진단할 수 있을 것으로 기대된다.
이번 연구결과는 영국왕립화학회가 발행하는 케미컬 커뮤니케이션즈(Chemical communications) 6월호 후면 표지논문으로 선정됐다.
기존의 분자 비콘(Molecular beacon) 프로브 기반 유전자 분석은 분석 대상인 표적 DNA가 변경되면 이에 대응하는 새로운 분자 비콘 프로브가 필요했다. 따라서 다양한 표적 DNA를 분석하는데 많은 비용이 필요하다는 한계가 있었다.
문제 해결을 위해 연구팀은 DNA 중합효소와 결합해 활성을 저해시키는 압타머를 고안했다. 그리고 이를 역으로 이용해 표적 DNA가 존재하는 경우에만 압타머가 DNA 중합효소와 결합하지 않고 활성을 유지할 수 있게 조절하는 기술을 최초로 개발했다.
이 기술 개발로 조절된 DNA 중합효소의 활성이 핵산 신장 및 절단 반응을 일으키고 그 결과로 형광 프로브(TaqMan probe)의 형광신호 측정이 가능해졌다. 따라서 동일한 형광 프로브를 이용해 다양한 표적 DNA를 민감하게 검출할 수 있는 새로운 유전자 진단 기술 개발이 가능해졌다.
이 기술은 표적 DNA의 종류에 따라 새로운 프로브를 사용해야 했던 기존 기술과 달리 동일한 형광 프로브를 이용하기 때문에 다양한 표적핵산을 값싸고 손쉽게 검출할 수 있다. 기술을 응용하면 과거에 비해 여러 가지 다른 병원균의 감염 여부를 저렴하고 수월하게 파악할 수 있다.
박 교수는 “메르스처럼 새로운 병원체에 대한 진단 키트를 용이하게 제작할 수 있어 여러 병원균에 대해 신속히 대응할 수 있다”며 “향후 유전자 진단 분야에서 새 원천기술로 널리 활용될 것으로 기대된다”고 말했다.
이번 연구는 미래창조과학부가 추진하는 글로벌프론티어사업(바이오나노헬스가드연구단)의 지원을 받아 수행됐다.
□ 그림 설명
그림 1. 표적핵산에 의한 DNA 중합효소 활성 변화를 이용해 표적 핵산을 검출한 모식도
2015.07.27 조회수 12502
표적 DNA 저렴하게 분석 가능한 유전자 진단 기술 개발
박 현 규 교수
우리 대학 생명화학공학과 박현규 교수 연구팀이 특정 단백질이나 효소를 인식하는 물질인 압타머(Aptamer : 표적 물질과 결합할 수 있는 특성을 가진 DNA)를 이용해 다양한 표적 DNA를 분석할 수 있는 기술을 개발했다.
이 기술을 통해 메르스와 같은 신종 바이러스 병원균 감염 여부 등 다양한 유전자를 기존에 비해 저렴한 가격으로 진단할 수 있을 것으로 기대된다.
이번 연구결과는 영국왕립화학회가 발행하는 케미컬 커뮤니케이션즈(Chemical communications) 6월호 후면 표지논문으로 선정됐다.
기존의 분자 비콘(Molecular beacon) 프로브 기반 유전자 분석은 분석 대상인 표적 DNA가 변경되면 이에 대응하는 새로운 분자 비콘 프로브가 필요했다. 따라서 다양한 표적 DNA를 분석하는데 많은 비용이 필요하다는 한계가 있었다.
문제 해결을 위해 연구팀은 DNA 중합효소와 결합해 활성을 저해시키는 압타머를 고안했다. 그리고 이를 역으로 이용해 표적 DNA가 존재하는 경우에만 압타머가 DNA 중합효소와 결합하지 않고 활성을 유지할 수 있게 조절하는 기술을 최초로 개발했다.
이 기술 개발로 조절된 DNA 중합효소의 활성이 핵산 신장 및 절단 반응을 일으키고 그 결과로 형광 프로브(TaqMan probe)의 형광신호 측정이 가능해졌다. 따라서 동일한 형광 프로브를 이용해 다양한 표적 DNA를 민감하게 검출할 수 있는 새로운 유전자 진단 기술 개발이 가능해졌다.
이 기술은 표적 DNA의 종류에 따라 새로운 프로브를 사용해야 했던 기존 기술과 달리 동일한 형광 프로브를 이용하기 때문에 다양한 표적핵산을 값싸고 손쉽게 검출할 수 있다. 기술을 응용하면 과거에 비해 여러 가지 다른 병원균의 감염 여부를 저렴하고 수월하게 파악할 수 있다.
박 교수는 “메르스처럼 새로운 병원체에 대한 진단 키트를 용이하게 제작할 수 있어 여러 병원균에 대해 신속히 대응할 수 있다”며 “향후 유전자 진단 분야에서 새 원천기술로 널리 활용될 것으로 기대된다”고 말했다.
이번 연구는 미래창조과학부가 추진하는 글로벌프론티어사업(바이오나노헬스가드연구단)의 지원을 받아 수행됐다.
□ 그림 설명
그림 1. 표적핵산에 의한 DNA 중합효소 활성 변화를 이용해 표적 핵산을 검출한 모식도
2015.07.27 조회수 12502 DNA 기반 반도체 핵심 원천기술 개발
박현규 교수
- 분자 비콘을 이용해 모든(8가지) 논리게이트 구현하는 데 성공 -- 스몰(Small)誌 7월호 표지논문으로 실려 -
초소형 미래 바이오전자기기를 구현하기 위한 핵심기술 개발됐다.
우리 학교 생명화학공학과 박현규 교수 연구팀이 DNA를 이용해 모든 논리게이트를 구현하는 데 성공, 나노분야의 세계적 학술지 ‘스몰(Small)’ 7월호(23일자) 표지논문으로 실렸다.
현재 최첨단 기술로도 10nm(나노미터) 이하의 실리콘 기반 반도체 제작은 불가능한 것으로 알려져 있지만, DNA는 굵기가 2nm 정도로 가늘기 때문에 보다 저렴하면서도 획기적인 집적도를 가진 반도체를 만들 수 있을 것으로 기대된다.
2나노급 반도체가 개발되면 우표 크기의 메모리 반도체에 고화질 영화 10000편을 저장하는 등 현재 상용화중인 20나노급 반도체보다 약 100배의 용량을 담을 수 있게 된다.
DNA는 네 종류의 염기인 아데닌(adenine, A), 시토신(cytosine, C), 구아닌(guanin, G), 티민(thymine, T)이 연속적으로 연결돼 있는데 A는 T와, G는 C와 각각 특이적으로 결합하는 특성을 갖고 있다.
특정 DNA는 특이적으로 결합하는 염기서열을 지닌 또 다른 DNA와 결합해 이중나선 구조를 형성하는 데, 연구팀은 이러한 DNA의 특이적 결합 특성과 구조 변화에 따른 형광신호 특성이 있는 고리모양의 분자 비콘을 이용했다.
연구팀은 생체 DNA물질을 디지털 회로에서 사용되는 논리게이트와 같은 역할을 담당하도록 입력신호로 사용해 고리모양의 DNA가 열리거나 닫히도록 했다.
고리모양 DNA가 열린 형태에서는 형광신호가 증가하고 닫힌 상태에서는 형광 신호가 감소하며 이로 인해 발생하는 형광신호의 변화를 출력신호로 사용했다.
연구팀은 제한적인 시스템만을 구현하는 기존의 논리게이트의 문제점을 극복, 8가지 모든 논리게이트(AND, OR, XOR, INHIBIT, NAND, NOR, XNOR, IMPlCATION)를 구현하는 데 성공해 반도체 기술로써의 적용 가능성을 높였다.
이와 함께, 각각의 논리게이트의 연결을 통한 다중 논리게이트(Multilevel circuits)와 논리게이트의 재생성을 보여주는 데도 성공했다.
박현규 교수는 “하나의 분자 비콘을 모든 게이트 구성을 위한 보편적인 요소로 사용해 저렴하면서도 초고집적 바이오 전자기기의 가능성을 높였다”며 “앞으로 분자 수준의 전자 소자 연구에 큰 변화가 있을 것으로 예상된다”고 말했다.
이번 연구를 주도한 박기수 박사과정 학생(제1저자)은 “DNA는 10개의 염기서열 길이가 3.4nm이고 굵기가 2nm밖에 되지 않는 매우 작은 물질이기 때문에 이를 이용해 전자 소자를 구현하면 획기적인 집적도 향상을 이룰 수 있다”며 “간단한 시스템 디자인을 통해 정확한 논리게이트를 구현해 내 DNA 반도체를 탑재한 바이오컴퓨터가 곧 현실로 다가올 것”이라고 말했다.XOR 게이트 : 입력 DNA A(input A)와 입력 DNA B(input B) 둘 중 하나만 있을 때는 고리모양 DNA가 열려서 형광 신호가 나오고(출력신호 1), DNA A와 B가 모두 없을 경우와 모두 있는 경우에는 고리모양 DNA가 고리모양을 유지하여 형광을 발생하지 않게 함으로써 XOR 논리게이트를 정확하게 구현했다.
2012.09.18 조회수 16762
DNA 기반 반도체 핵심 원천기술 개발
박현규 교수
- 분자 비콘을 이용해 모든(8가지) 논리게이트 구현하는 데 성공 -- 스몰(Small)誌 7월호 표지논문으로 실려 -
초소형 미래 바이오전자기기를 구현하기 위한 핵심기술 개발됐다.
우리 학교 생명화학공학과 박현규 교수 연구팀이 DNA를 이용해 모든 논리게이트를 구현하는 데 성공, 나노분야의 세계적 학술지 ‘스몰(Small)’ 7월호(23일자) 표지논문으로 실렸다.
현재 최첨단 기술로도 10nm(나노미터) 이하의 실리콘 기반 반도체 제작은 불가능한 것으로 알려져 있지만, DNA는 굵기가 2nm 정도로 가늘기 때문에 보다 저렴하면서도 획기적인 집적도를 가진 반도체를 만들 수 있을 것으로 기대된다.
2나노급 반도체가 개발되면 우표 크기의 메모리 반도체에 고화질 영화 10000편을 저장하는 등 현재 상용화중인 20나노급 반도체보다 약 100배의 용량을 담을 수 있게 된다.
DNA는 네 종류의 염기인 아데닌(adenine, A), 시토신(cytosine, C), 구아닌(guanin, G), 티민(thymine, T)이 연속적으로 연결돼 있는데 A는 T와, G는 C와 각각 특이적으로 결합하는 특성을 갖고 있다.
특정 DNA는 특이적으로 결합하는 염기서열을 지닌 또 다른 DNA와 결합해 이중나선 구조를 형성하는 데, 연구팀은 이러한 DNA의 특이적 결합 특성과 구조 변화에 따른 형광신호 특성이 있는 고리모양의 분자 비콘을 이용했다.
연구팀은 생체 DNA물질을 디지털 회로에서 사용되는 논리게이트와 같은 역할을 담당하도록 입력신호로 사용해 고리모양의 DNA가 열리거나 닫히도록 했다.
고리모양 DNA가 열린 형태에서는 형광신호가 증가하고 닫힌 상태에서는 형광 신호가 감소하며 이로 인해 발생하는 형광신호의 변화를 출력신호로 사용했다.
연구팀은 제한적인 시스템만을 구현하는 기존의 논리게이트의 문제점을 극복, 8가지 모든 논리게이트(AND, OR, XOR, INHIBIT, NAND, NOR, XNOR, IMPlCATION)를 구현하는 데 성공해 반도체 기술로써의 적용 가능성을 높였다.
이와 함께, 각각의 논리게이트의 연결을 통한 다중 논리게이트(Multilevel circuits)와 논리게이트의 재생성을 보여주는 데도 성공했다.
박현규 교수는 “하나의 분자 비콘을 모든 게이트 구성을 위한 보편적인 요소로 사용해 저렴하면서도 초고집적 바이오 전자기기의 가능성을 높였다”며 “앞으로 분자 수준의 전자 소자 연구에 큰 변화가 있을 것으로 예상된다”고 말했다.
이번 연구를 주도한 박기수 박사과정 학생(제1저자)은 “DNA는 10개의 염기서열 길이가 3.4nm이고 굵기가 2nm밖에 되지 않는 매우 작은 물질이기 때문에 이를 이용해 전자 소자를 구현하면 획기적인 집적도 향상을 이룰 수 있다”며 “간단한 시스템 디자인을 통해 정확한 논리게이트를 구현해 내 DNA 반도체를 탑재한 바이오컴퓨터가 곧 현실로 다가올 것”이라고 말했다.XOR 게이트 : 입력 DNA A(input A)와 입력 DNA B(input B) 둘 중 하나만 있을 때는 고리모양 DNA가 열려서 형광 신호가 나오고(출력신호 1), DNA A와 B가 모두 없을 경우와 모두 있는 경우에는 고리모양 DNA가 고리모양을 유지하여 형광을 발생하지 않게 함으로써 XOR 논리게이트를 정확하게 구현했다.
2012.09.18 조회수 16762